
Puntuación de propensión (Propensity score) en la inferencia causal
Introducción
Vamos a continuar nuestra inmersión en el mundo de la inferencia causal y su moderno conjunto de herramientas. Obviamente, la tarea resultará algo más amplia: hoy aplicaremos métodos de inferencia causal al trading. Ya hemos empezado a aprender lo básico e incluso hemos escrito nuestros primeros meta-aprendices, que, por cierto, han resultado ser bastante sólidos. Siendo más concretos, podemos afirmar que los modelos obtenidos con ellos son robustos. Vale la pena precisar que las primeras eran solo para el lector, porque para el escritor son simplemente un experimento más. Así que no hay vuelta atrás, tendremos que continuar hasta que se agote todo el tema de la inferencia causal en el trading. Al fin y al cabo, los enfoques de la inferencia causal pueden variar, y querríamos abarcar este interesante tema lo más ampliamente posible. O quizá no deberíamos proseguir, pues uno de los resultados potenciales nos es desconocido, y ahí reside la paradoja básica de la inferencia causal (estoy bromeando).
En este artículo abordaremos el tema del emparejamiento, que ya tratamos brevemente en el artículo anterior, o más bien una de sus variedades: el emparejamiento por puntuación de propensión.
Esto es importante porque disponemos de un cierto conjunto de datos etiquetados que son heterogéneos. Por ejemplo, en el mercado de divisas, cada ejemplo de entrenamiento individual puede pertenecer a la zona de alta o baja volatilidad, además, algunos ejemplos pueden aparecer con mayor frecuencia en la muestra y otros con menor frecuencia. Al intentar determinar el efecto causal medio (ATE) en una muestra de este tipo, inevitablemente nos encontraremos con estimaciones desplazadas si suponemos que todos los ejemplos de la muestra tienen la misma propensión a someterse a un tratamiento. Y cuando intentamos obtener un efecto medio condicional del tratamiento (CATE) podemos encontrarnos con una molestia llamada "la maldición de la dimensionalidad".
El emparejamiento supone una familia de métodos para estimar los efectos causales comparando observaciones (o unidades) similares en los grupos de tratamiento y control. El objetivo del emparejamiento es realizar comparaciones entre unidades similares para lograr una estimación lo más precisa posible del verdadero efecto causal.
Algunos autores de materiales sobre la inferencia causal sugieren que el emparejamiento debería verse como un paso de preprocesamiento de datos sobre el que se puede utilizar cualquier estimador (por ejemplo, un meta-aprendiz). Si disponemos de datos suficientes para descartar algunas observaciones de manera potencial, suele resultar útil utilizar el emparejamiento como paso previo al tratamiento.
Imagine que dispone de un conjunto de datos para analizar. Estos datos contienen 1 000 observaciones. ¿Cuáles son las probabilidades de encontrar al menos una coincidencia exacta para cada fila si en su conjuntos de datos hay 18 variables? La respuesta dependerá obviamente de varios factores. ¿Cuántas variables son binarias? ¿Cuántas son continuas? ¿Cuántas de ellas son categóricas? ¿Cuál es el número de niveles de las variables categóricas? ¿Las variables son independientes o están correlacionadas entre sí?
Aleksander Molak en su libro "Causal inference and discovery in Python" ofrece un buen ejemplo ilustrativo.
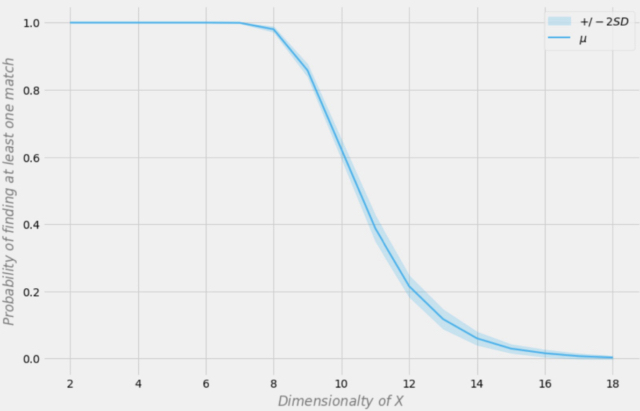
Probabilidad de encontrar una coincidencia exacta según la dimensionalidad del conjunto de datos
Supongamos que tenemos 1 000 observaciones en nuestra muestra. En la figura anterior, el eje X representa la dimensionalidad del conjunto de datos (el número de variables del conjunto de datos), mientras que el eje Y representa la probabilidad de encontrar al menos una coincidencia exacta en cada fila.
La línea azul supone la probabilidad media, y las zonas sombreadas representan +/- dos desviaciones estándar. El conjunto de datos se creó utilizando distribuciones Bernoulli independientes con p = 0,5. Por lo tanto, cada variable es binaria e independiente.
Así, decidí poner a prueba esta afirmación del libro y escribí un script en Python que calcule esta probabilidad.
import numpy as np import matplotlib.pyplot as plt def calculate_exact_match_probability(dimensions): num_samples = 1000 num_trials = 1000 match_count = 0 for _ in range(num_trials): dataset = np.random.randint(2, size=(num_samples, dimensions)) row_sums = np.sum(dataset, axis=1) if any(row_sums == dimensions): match_count += 1 return match_count / num_trials def plot_probability_curve(max_dimensions): dimensions_range = list(range(1, max_dimensions + 1)) probabilities = [calculate_exact_match_probability(dim) for dim in dimensions_range] mean_probability = np.mean(probabilities) std_dev = np.std(probabilities) plt.plot(dimensions_range, probabilities, label='Exact Match Probability') plt.axhline(mean_probability, color='blue', linestyle='--', label='Mean Probability') plt.xlabel('Number of Variables') plt.ylabel('Probability') plt.title('Exact Match Probability in Binary Dataset') plt.legend() plt.show()
De hecho, para 1 000 observaciones, las probabilidades coincidían. A modo de experimento, puede calcularlos usted mismo para las dimensionalidades de sus conjuntos de datos. Para nuestro futuro trabajo, bastará con ser conscientes de este hecho: si hay demasiadas características (covariables) en los datos en comparación con el número de ejemplos de entrenamiento, la capacidad de generalizar dichos datos usando un clasificador será limitada. Aquí casi siempre funciona el siguiente principio: cuantos más datos, más precisas serán las estimaciones estadísticas.
*En la práctica, esto no siempre es así, ya que deberá cumplirse la condición i.i.d. (independent and identically-distributed, independiente e idénticamente distribuida).
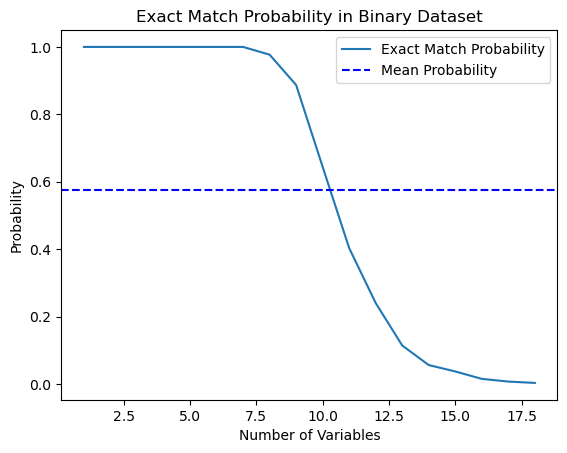
Como podemos ver, la probabilidad de encontrar una coincidencia exacta en un conjunto de datos aleatorios binarios de 18 dimensiones es esencialmente cero. En el mundo real, rara vez trabajamos con conjuntos de datos puramente binarios y, en el caso de los datos continuos, la correspondencia multidimensional se hace aún más difícil. Y esto supone un grave problema para el emparejamiento, incluso en el caso aproximado. ¿Cómo podemos resolver este problema?
Reducción de la dimensionalidad de los datos utilizando la puntuación de propensión
Podemos resolver la maldición de la dimensionalidad usando la puntuación de propensión. Las valoraciones de propensión son estimaciones de la probabilidad de que una unidad determinada sea asignada al grupo experimental según sus características. Según el teorema de la puntuación de propensión (Rosenbaum y Rubin, 1983), si tenemos datos no mezclados para las características X, también tendremos datos no mezclados dada la puntuación de propensión, suponiendo la positividad. Positividad significa que las distribuciones tratadas y no tratadas deben solaparse. Se trata de una suposición sobre la positividad de la inferencia causal; también tiene un sentido intuitivo. Si el grupo de prueba y el grupo de control no coinciden, significará que son muy diferentes y no podremos extrapolar el efecto de un grupo al otro. Tal extrapolación no es imposible (la hace la regresión), pero resulta muy peligrosa. Esto sería similar a probar un nuevo fármaco en un experimento en el que solo se administra a hombres y luego suponer que las mujeres responderán igual de bien a él. Formalmente, la puntuación de propensión se escribe de la forma que sigue:
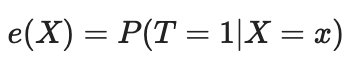
En un mundo perfecto, tendríamos una verdadera puntuación de propensión. En la práctica, sin embargo, desconocemos el mecanismo de asignación de tratamientos, y necesitamos sustituir la predisposición real por su estimación o expectativa. Una forma habitual de hacerlo es usando la regresión logística, pero también podemos emplear otras técnicas de aprendizaje automático, como la potenciación del gradiente (aunque esto requerirá algunos pasos adicionales para evitar el sobreentrenamiento).
Podemos usar esta ecuación para resolver el problema de la multidimensionalidad. Las puntuaciones de propensión son unidimensionales, por lo que ahora solo podremos emparejar dos valores en lugar de vectores multidimensionales.
Por lo tanto, si tenemos un independencia condicional de los resultados potenciales del tratamiento,
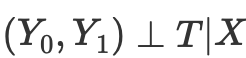
entonces podremos calcular el efecto causal medio para los casos continuos y discretos en los emparejamientos sin puntuación de propensión:
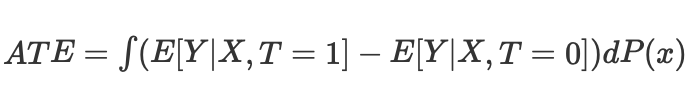
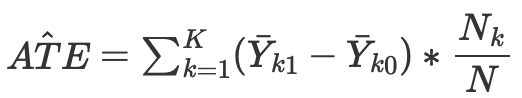
Donde Nk será el número de observaciones en cada celda. Una celda se refiere a un subconjunto de observaciones emparejadas según alguna métrica de proximidad.
No obstante, la puntuación de propensión surge de la constatación de que no necesitamos controlar directamente los factores de distorsión X para lograr la independencia condicional
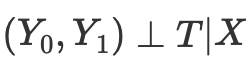
En su lugar, bastará con controlar el indicador de equilibrio
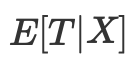
La puntuación de propensión permite ser independiente de X en su conjunto para conseguir la independencia de los resultados potenciales del tratamiento. Basta con condicionar esta única variable, que será una puntuación de propensión:
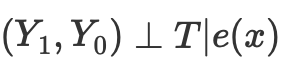
Diversa literatura sobre inferencia causal describe una serie de problemas con este enfoque:
- En primer lugar, las puntuaciones de propensión reducen la dimensionalidad de nuestros datos y, por definición, nos obligan a desechar una parte de la información.
- En segundo lugar, dos observaciones que sean muy diferentes en el espacio de características original pueden tener la misma puntuación de propensión. Esto puede dar lugar a solapamientos entre observaciones muy distintas y, por tanto, a resultados engañosos.
- En tercer lugar, el PSM (Propensity score modelling) conduce a una paradoja. En el caso binario, la puntuación de propensión óptima sería 0,5. ¿Y qué ocurre en un escenario ideal, cuando todas las observaciones tienen una puntuación de propensión óptima de 0,5? La posición de cada observación en el espacio de puntuación de propensión pasará a ser idéntica a la de cualquier otra observación. Esto se conoce en ocasiones como la paradoja PSM.
Emparejamiento mediante la puntuación de propensión y sus métodos relacionados
Existen varios métodos para comparar (emparejar) unidades basándose en la puntuación de propensión. El principal método generalmente aceptado es el método del vecino más próximo; este compara cada unidad i del grupo experimental con la unidad j del grupo de control con la distancia absoluta más próxima entre sus puntuaciones de propensión expresadas como
d(i, j) = minj{|e(Xi) – e(Xj)|}.
Alternativamente, el emparejamiento por umbral empareja cada unidad i del grupo de tratamiento con la unidad j del grupo de control dentro de un umbral predeterminado b; es decir.
d(i, j) = minj{|e(Xi) – e(Xj)| <b}.
Resulta recomendable que el umbral predeterminado b sea inferior o igual a 0,25 de la desviación estándar de las puntuaciones de propensión. Otros investigadores sostienen que lo óptimo sería b = 0,20 de la desviación estándar de las puntuaciones de propensión.
Otra variante del emparejamiento de umbrales sería el emparejamiento de radios, que es un emparejamiento de uno a muchos y empareja cada unidad i del grupo de tratamiento con múltiples unidades del grupo de control dentro de un rango predeterminado b; es decir.
d(i, j) = {|e(Xi) – e(Xj)| <b}.
Otros métodos de emparejamiento de puntuación de propensión incluyen la métrica de Mahalanobis. En el emparejamiento mediante la métrica de Mahalanobis, cada unidad i del grupo experimental se empareja con la unidad j del grupo de control, calculándose la distancia de Mahalanobis más próxima según la proximidad de las variables.
Los métodos de emparejamiento de puntuaciones de propensión analizados hasta ahora pueden aplicarse usando un algoritmo de emparejamiento codicioso o un algoritmo de emparejamiento óptimo.
- En el emparejamiento codicioso, una vez realizado el emparejamiento, las unidades emparejadas no pueden modificarse. Cada par de unidades emparejadas será el mejor par disponible en la actualidad.
- En el emparejamiento óptimo, las unidades emparejadas anteriores pueden cambiarse antes de efectuar el emparejamiento actual para lograr la distancia mínima u óptima global.
- Ambos algoritmos de emparejamiento suelen producir los mismos datos emparejados cuando el tamaño del grupo de control es grande; no obstante, el emparejamiento óptimo ofrece como resultado distancias globales más pequeñas dentro de las unidades emparejadas. Así, si el objetivo es simplemente encontrar grupos bien emparejados, podría bastar con el emparejamiento codicioso; si en cambio el objetivo es encontrar parejas bien emparejadas, podría resultar preferible el emparejamiento óptimo.
Existen métodos relacionados con el emparejamiento de puntuaciones de propensión que no comparan estrictamente unidades de muestreo individuales, por ejemplo, la subclasificación (o estratificación) clasifica todas las unidades de la muestra completa en varios estratos basándose en el número correspondiente de percentiles de puntuaciones de propensión y empareja las unidades según el estrato. Se ha observado que cinco estratos eliminan hasta el 90% de los errores de selección.
Un tipo especial de subclasificación es la asignación completa, en la que las subclases se crean de manera óptima. Una muestra totalmente emparejada consta de subconjuntos emparejados en los que cada conjunto contiene una unidad experimental y una o más unidades de control o una unidad de control y una o más unidades experimentales. El emparejamiento completo resulta óptimo en términos de minimización de la media ponderada de la medida de distancia estimada entre cada sujeto del tratamiento y cada sujeto de control en cada subclase.
Otro método relacionado con el emparejamiento de puntuación de propensión es el emparejamiento por núcleo (o emparejamiento de comparación local), que combina la comparación y el análisis de resultados en un único procedimiento con un emparejamiento uno a uno.
A pesar de la variedad de métodos de emparejamiento propuestos, su eficacia dependerá más de la correcta formulación del problema que del método específico.
El supuesto de negligencia fuerte
La "negligencia fuerte" es un supuesto crucial en la construcción de la puntuación de propensión, cuyo objetivo consiste en estimar los efectos causales en observaciones en las que la asignación al tratamiento es aleatoria. En esencia, esto significa que la asignación al tratamiento será independiente de los resultados potenciales dadas las covariables de referencia observadas (rasgos).
Veámoslo con más detalle:
- Asignación de tratamiento: se trata de si una unidad recibe tratamiento o no (por ejemplo, si toma un nuevo medicamento o participa en un programa).
- Resultados potenciales: son los resultados que una unidad habría experimentado tanto en condiciones de tratamiento como de control, pero solo podremos observar uno para cada unidad.
- Covariables básicas: son características de la unidad medidas antes de la asignación del tratamiento que pueden afectar tanto a la probabilidad de recibirlo como al resultado.
El supuesto "fuertemente ignorable" establece que:
- Sin factores de confusión no medidos: no hay variables no observadas que afecten tanto a la asignación del tratamiento como al resultado del mismo. Esto es importante porque los factores de confusión no observados pueden introducir sesgos en el efecto del tratamiento estimado.
- Positividad: cada unidad tiene una probabilidad distinta de cero de recibir tanto el tratamiento como el control, dadas sus covariables observadas. Esto garantiza que haya suficientes unidades en los grupos comparados para que la comparación resulte significativa.
Si se cumplen estas condiciones, el condicionamiento de la puntuación de propensión (probabilidad estimada de recibir tratamiento dadas las covariables) producirá estimaciones no sesgadas del efecto medio del tratamiento (ATE). El ATE representa la diferencia media en los resultados entre los grupos de tratamiento y control como si los tratamientos se hubieran asignado aleatoriamente.
Ponderación de probabilidad inversa
La ponderación de probabilidad inversa es un método para eliminar los factores de confusión o interferencia mediante el intento de reponderar las observaciones de un conjunto de datos basándose en la probabilidad inversa de asignación al tratamiento. La idea consiste en dar más peso a las observaciones que se evalúan como menos propensas a ser tratadas con una asignación de tratamiento, haciéndolas más representativas de la población general.
- En primer lugar, se estima la puntuación de propensión, que es la probabilidad de recibir el tratamiento dadas las covariables observadas.
- Luego se calcula la puntuación de propensión inversa para cada observación.
- A continuación, cada observación se multiplica por su peso correspondiente. Y esto significa que las observaciones con menor probabilidad de recibir el tratamiento observado se ponderan más.
- Después, el conjunto de datos ponderados se usa para el análisis. Se aplican ponderaciones tanto al grupo experimental como al de control, ajustando la posible influencia de las covariables observadas.
La ponderación de probabilidad inversa puede ayudar a equilibrar la distribución de las covariables entre los grupos procesados (tratamiento) y de control, reduciendo el error sistemático en la estimación de los efectos causales. Sin embargo, se basa en el supuesto de que todas las variables interferentes pertinentes se medirán e incluirán en el modelo utilizado para estimar la puntuación de propensión. Además, al igual que ocurre con cualquier método estadístico, el éxito de IPW dependerá de la calidad del modelo utilizado para estimar la puntuación de propensión.
La fórmula de la ponderación de probabilidad inversa es la siguiente:
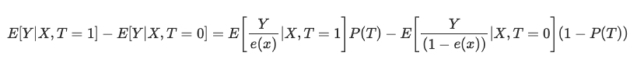
Sin entrar en detalles, podemos comparar los dos términos de la ecuación. El izquierdo para el grupo de tratamiento, el derecho para el grupo de control. La fórmula demuestra que una simple comparación de medias equivale a una comparación de medias ponderadas inversamente. Esto crea una población del mismo tamaño que la población original, pero en la que todos los del lado izquierdo reciben el tratamiento. Del mismo modo, el derecho se fija en los no tratados y da importancia a los que se parecen a los tratados.
Evaluación de los resultados tras el emparejamiento
En la inferencia causal, la estimación se construye como ATE o CATE, es decir, la diferencia de las medias ponderadas (considerando la puntuación de propensión) de los valores objetivo tratados y no tratados.
Una vez hemos obtenido una puntuación de propensión en forma de e(x), podemos utilizar estos valores, por ejemplo, para entrenar otro clasificador, en lugar de los valores originales de las características de X. Y también podemos emparejar muestras concretas de una muestra según su puntuación de propensión para dividir en estratos. Otra opción sería añadir e(x) como una característica independiente al entrenar el estimador final, lo cual ayudaría a eliminar las puntuaciones sesgadas debidas a la confusión, cuando diferentes ejemplos de la muestra tienen diferentes puntuaciones de propensión.
Nos interesa encontrar subgrupos que se presten bien o mal al tratamiento (aprendizaje de modelos). Y luego entrenar el clasificador final solo con los datos que estén bien entrenados (donde el error de clasificación sea mínimo). Los datos mal clasificados debe colocarse en el segundo subgrupo y el segundo clasificador debe entrenarse para distinguir entre estos dos subgrupos, es decir, para separar el grano de la paja, o para identificar los subgrupos que se prestan mejor al tratamiento. Por lo tanto, ahora no adoptaremos toda la metodología de la puntuación de propensión, sino que emparejaremos las muestras según las probabilidades obtenidas del clasificador entrenado, mientras que la puntuación global del ATE (efecto medio del tratamiento) nos interesará poco.
En otras palabras, basaremos nuestra evaluación en el rendimiento del algoritmo en datos nuevos que no han participado en el entrenamiento. Además, seguiremos interesados en la velocidad media del conjunto de modelos entrenados con datos aleatorios. Cuanto mayor sea la puntuación media de los modelos independientes, mayor será la confianza en cada modelo concreto.
Vamos a pasar a los experimentos
Al empezar este artículo me di cuenta de que para muchos tráders, especialmente los que no están familiarizados con el aprendizaje automático, este material en profundidad puede parecer muy poco intuitivo. Recordando mi primera introducción a la inferencia causal, este malentendido inicial afectó tanto a mi ego que no pude evitar entrar en más detalles. Es más, ni siquiera podía imaginar tomarme la libertad de adaptar las técnicas de inferencia causal a la clasificación de series temporales.
- archivo propensity without matching.py
Empezaremos con una forma de incorporar las puntuaciones de propensión directamente a nuestro estimador, o meta-aprendiz. Para ello, deberemos entrenarse dos modelos. En primer lugar, el propio PSM (propensity score model), que proporcionará las probabilidades de asignar los ejemplos de entrenamiento a los grupos de tratamiento o de prueba. Las probabilidades obtenidas, junto con los signos (covariables), se introducirán en la entrada del segundo modelo, que pronosticará los resultados (compra o venta).
La intuición de este enfoque es que ahora el meta-aprendiz podrá distinguir entre subgrupos de muestras en función de su propensión a la tratamiento. De este modo, obtendremos predicciones ponderadas de los resultados que deberían resultar más precisas. A continuación, dividiremos el conjunto de datos en casos bien predecibles y casos poco predecibles, como ya hemos hecho en artículos anteriores. En este caso, no necesitaremos un emparejamiento explícito de la muestra porque el meta-aprendiz tendrá en cuenta automáticamente la puntuación de propensión en sus estimaciones. Este enfoque me parece bastante práctico porque el aprendizaje automático hará todo el trabajo por nosotros.
En primer lugar, crearemos las submuestras train y val para entrenar el metamodelo. Dado que el meta-aprendiz se entrenará con la submuestra de entrenamiento, crearemos un par de objetivos y_T1, y_T0, y los rellenaremos con unos y ceros. Esto se corresponderá con el hecho de si las unidades han recibido o no tratamiento (entrenamiento del modelo). A continuación, volveremos a mezclar la submuestra, siendo las variables objetivo los tratamientos.
X_train, X_val, y_train, y_val = train_test_split( X, y, train_size = 0.5, test_size = 0.5, shuffle = True) # randomly assign treated and control y_T1 = pd.DataFrame(y_train) y_T1['T'] = 1 y_T1 = y_T1.drop(['labels'], axis=1) y_T0 = pd.DataFrame(y_val) y_T0['T'] = 0 y_T0 = y_T0.drop(['labels'], axis=1) y_TT = pd.concat([y_T1, y_T0]) y_TT = y_TT.sort_index() X_trainT, X_valT, y_trainT, y_valT = train_test_split( X, y_TT, train_size = 0.5, test_size = 0.5, shuffle = True)
El siguiente paso será entrenar el modelo PSM para predecir si las muestras pertenecen a las submuestras de tratamiento o de control.
# fit propensity model PSM = CatBoostClassifier(iterations = iterations, depth = depth, custom_loss = ['Accuracy'], eval_metric = 'Accuracy', use_best_model=True, early_stopping_rounds=15, verbose = False).fit(X_trainT, y_trainT, eval_set = (X_valT, y_valT), plot = False)
A continuación, tendremos que obtener predicciones de pertenencia a los grupos de tratamiento y control y añadirlas a las características de la meta-aprendiz, para después entrenarla.
# predict probabilities train_proba = PSM.predict_proba(X_train)[:, 1] val_proba = PSM.predict_proba(X_val)[:, 1] # fit meta-learner meta_m = CatBoostClassifier(iterations = iterations, depth = depth, custom_loss = ['Accuracy'], eval_metric = 'Accuracy', verbose = False, use_best_model = True, early_stopping_rounds=15).fit(X_train.assign(T=train_proba), y_train, eval_set = (X_val.assign(T=val_proba), y_val), plot = False)
En el último paso, obtendremos las predicciones del meta-aprendiz, compararemos las etiquetas predichas con las etiquetas reales y completaremos el libro de malos ejemplos.
# create daatset with predicted values predicted_PSM = PSM.predict_proba(X)[:,1] X_psm = X.assign(T=predicted_PSM) coreset = X.assign(T=predicted_PSM) coreset['labels'] = y coreset['labels_pred'] = meta_m.predict_proba(X_psm)[:,1] coreset['labels_pred'] = coreset['labels_pred'].apply(lambda x: 0 if x < 0.5 else 1) # add bad samples of this iteration (bad labels indices) coreset_b = coreset[coreset['labels']==0] coreset_s = coreset[coreset['labels']==1] diff_negatives_b = coreset_b['labels'] != coreset_b['labels_pred'] diff_negatives_s = coreset_s['labels'] != coreset_s['labels_pred'] BAD_BUY = BAD_BUY.append(diff_negatives_b[diff_negatives_b == True].index) BAD_SELL = BAD_SELL.append(diff_negatives_s[diff_negatives_s == True].index)
Ahora probaremos a entrenar 25 modelos y veremos los mejores resultados y la media.
options = [] best_res = 0.0 for i in range(25): print('Learn ' + str(i) + ' model') options.append(learn_final_models(meta_learners(models_number=5, iterations=25, depth=2, bad_samples_fraction=0.5))) if options[-1][0] > best_res: best_res = options[-1][0] print("BEST: " + str(best_res)) options.sort(key=lambda x: x[0]) test_model(options[-1][1:], plt=True) test_all_models(options)
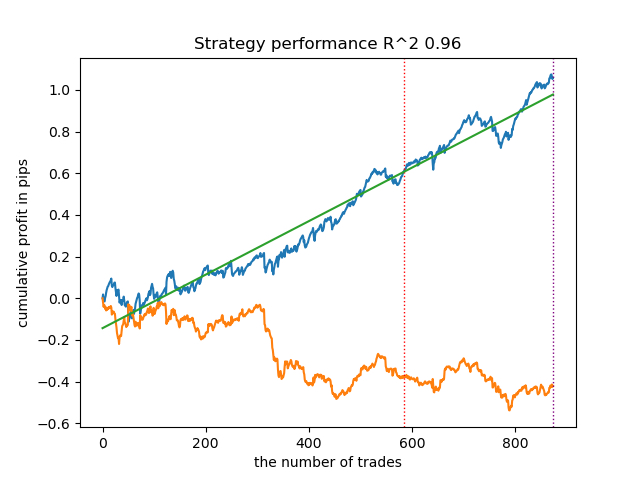
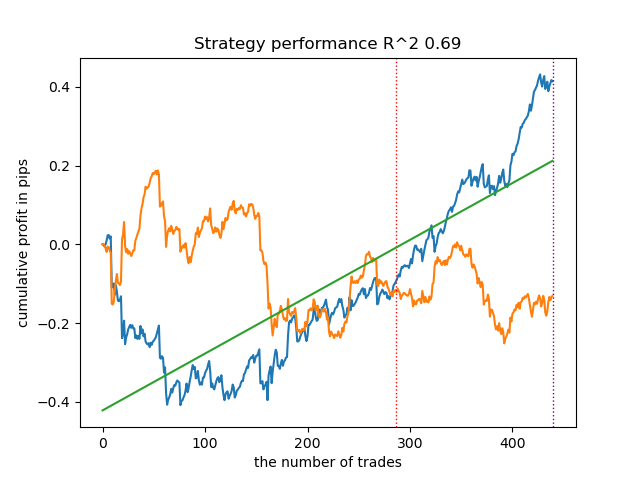
He llevado a cabo una serie de sesiones de entrenamiento de este tipo y he llegado a la conclusión de que este enfoque no difiere prácticamente en la calidad del modelo de mi enfoque en el artículo anterior. Ambos son capaces de generar buenos modelos que superan el OOS. Esto no debería sorprendernos porque hemos usado e(x) como característica adicional y el resto del algoritmo no cambia.
- archivo propensity matching naive.py
Al pensar en métodos de aplicación, nunca sabemos de antemano qué método funcionará mejor y cuál peor. A modo de experimento, he decidido hacer el emparejamiento no en función de la propensión a asignar el tratamiento, sino según la propensión a predecir etiquetas objetivo. Esto debería parecer más intuitivo al lector. La principal diferencia aquí es que solo se entrenará un modelo PSM, ahora denominado convencionalmente. A continuación, se pronosticarán las probabilidades y se creará una lista de bins según el número de estratos en los que queremos dividir las probabilidades resultantes. Para cada estrato se contará el número de resultados adivinados correcta/incorrectamente y, a continuación, para los estratos en los que el número de ejemplos adivinados incorrectamente (multiplicado por el coeficiente) supere el número de ejemplos adivinados correctamente, se activará la condición de adición de malos ejemplos al libro de malos ejemplos.
bins = np.linspace(lower_bound, upper_bound, num=bins_number) coreset['bin'] = pd.cut(coreset['propensity'], bins) for val in range(len(coreset['bin'].unique())): values = coreset.loc[coreset['bin'] == coreset['bin'].unique()[val]] diff_negatives = values['labels'] != values['labels_pred'] if len(diff_negatives[diff_negatives == False]) < (len(diff_negatives[diff_negatives == True]) * coefficient): B_S_B = B_S_B.append(diff_negatives[diff_negatives == True].index)
Este enfoque tiene parámetros adicionales:
- bins_number - número de bins
- lower_bound - límite inferior de probabilidad a partir del cual se cuentan los estratos
- upper_bound - límite superior de probabilidad hasta el que se cuentan los estratos
Dado que utilizaremos un meta-aprendiz con poca profundidad, las probabilidades tenderán a agruparse en torno a 0,5 y rara vez alcanzarán los límites marginales. Por lo tanto, es posible descartar los valores extremos por no resultar informativos estableciendo los límites superior e inferior.
Así, entrenaremos 25 modelos y veremos los resultados. Me gustaría señalar que todos los modelos se entrenarán con el mismo conjunto de datos, por lo que su comparación resultará bastante correcta.
options = [] best_res = 0.0 for i in range(25): print('Learn ' + str(i) + ' model') options.append(learn_final_models(meta_learners(models_number=5, iterations=25, depth=2, bad_samples_fraction=0.5, bins_number=25, lower_bound=0.3, upper_bound=0.7, coefficient=1.0))) if options[-1][0] > best_res: best_res = options[-1][0] print("BEST: " + str(best_res)) options.sort(key=lambda x: x[0]) test_model(options[-1][1:], plt=True)
Sorprendentemente, esta aplicación espontánea ha funcionado bastante bien con los nuevos datos. A continuación le mostramos los gráficos de negociación del mejor modelo y de los 25 modelos a la vez.
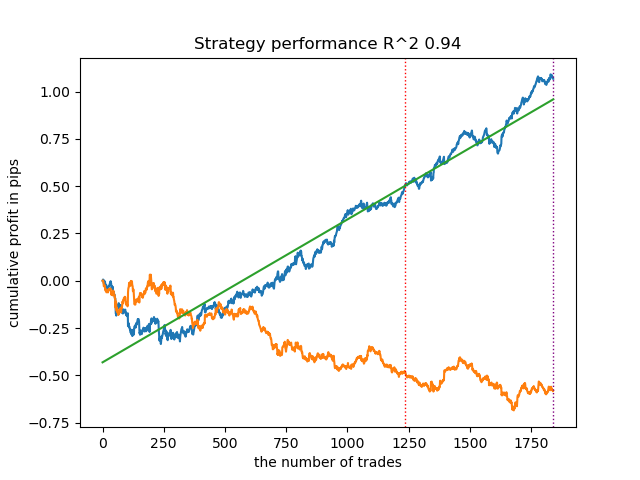
- archivo propensity matching original.py
Ahora procederemos a la aplicación del ejemplo más cercano a la teoría, en el que se cumplirá el supuesto «fuertemente ignorable». Recordemos que la asignación de tratamientos resulta fuertemente ignorable cuando no depende en modo alguno de los resultados potenciales, es decir, cuando es completamente aleatoria y no existen variables no contabilizadas que afecten al sesgo. Para ello, asignaremos aleatoriamente un tratamiento y entrenaremos un modelo PSM. A continuación, entrenaremos el meta-estimador para predecir los resultados de las transacciones (etiquetas de clase). Después estratificaremos la muestra según la puntuación de propensión y añadiremos al libro de malos ejemplos solo las muestras de aquellos intervalos en los que el número de predicciones fallidas supere al número de predicciones acertadas, dada la proporción.
También hemos añadido la posibilidad de aplicar IPW (inverse probability weighting, ponderación de probabilidad inversa), que se describe en la parte teórica.
Una vez entrenados los dos clasificadores, se ejecutará el siguiente código.
# create daatset with predicted values coreset = X.copy() coreset['labels'] = y coreset['propensity'] = PSM.predict_proba(X)[:, 1] if Use_IPW: coreset['propensity'] = coreset['propensity'].apply(lambda x: 1 / x if x > 0.5 else 1 / (1 - x)) coreset['propensity'] = coreset['propensity'].round(3) coreset['labels_pred'] = meta_m.predict_proba(X)[:, 1] coreset['labels_pred'] = coreset['labels_pred'].apply(lambda x: 0 if x < 0.5 else 1) bins = np.linspace(lower_bound, upper_bound, num=bins_number) coreset['bin'] = pd.cut(coreset['propensity'], bins) for val in range(len(coreset['bin'].unique())): values = coreset.loc[coreset['bin'] == coreset['bin'].unique()[val]] diff_negatives = values['labels'] != values['labels_pred'] if len(diff_negatives[diff_negatives == False]) < (len(diff_negatives[diff_negatives == True]) * coefficient): B_S_B = B_S_B.append(diff_negatives[diff_negatives == True].index)
Ahora entrenaremos 25 modelos sin IPW y observaremos el mejor gráfico de balance y la media de todos los modelos con estos ajustes:
options = [] best_res = 0.0 for i in range(25): print('Learn ' + str(i) + ' model') options.append(learn_final_models(meta_learners(models_number=5, iterations=25, depth=2, bad_samples_fraction=0.5, bins_number=25, lower_bound=0.3, upper_bound=0.7, coefficient=1.5, Use_IPW=False))) if options[-1][0] > best_res: best_res = options[-1][0] print("BEST: " + str(best_res))
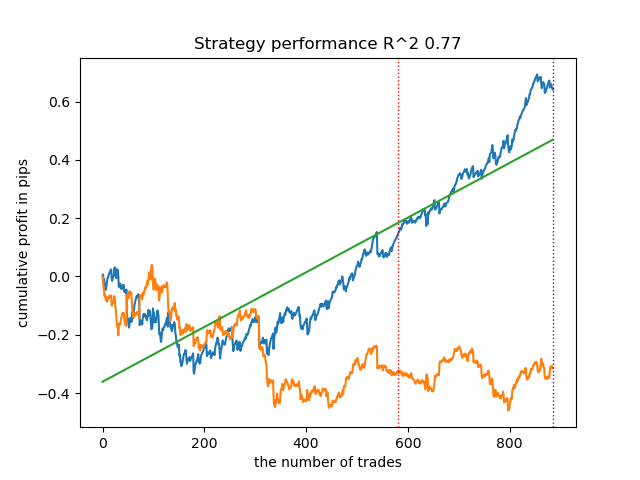
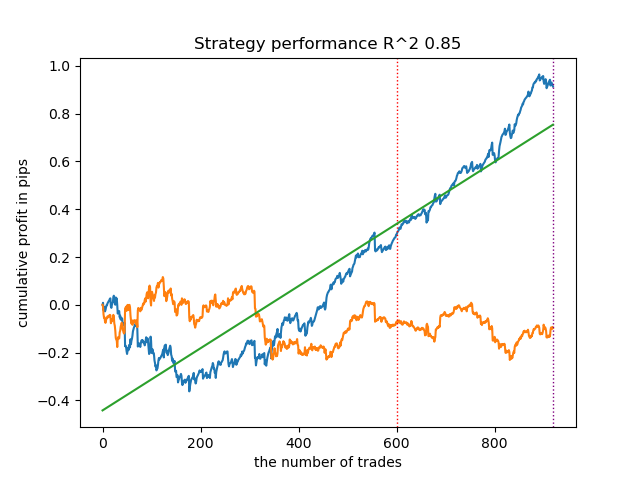
En general, los resultados son comparables a los de anteriores aplicaciones. Ahora haremos lo mismo con IPW activado.
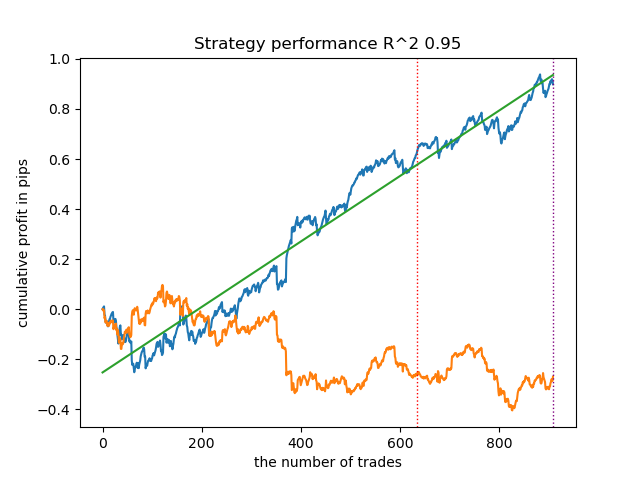
options = [] best_res = 0.0 for i in range(25): print('Learn ' + str(i) + ' model') options.append(learn_final_models(meta_learners(models_number=5, iterations=25, depth=2, bad_samples_fraction=0.5, bins_number=25, lower_bound=0.1, upper_bound=10.0, coefficient=1.5, Use_IPW=True))) if options[-1][0] > best_res: best_res = options[-1][0] print("BEST: " + str(best_res))

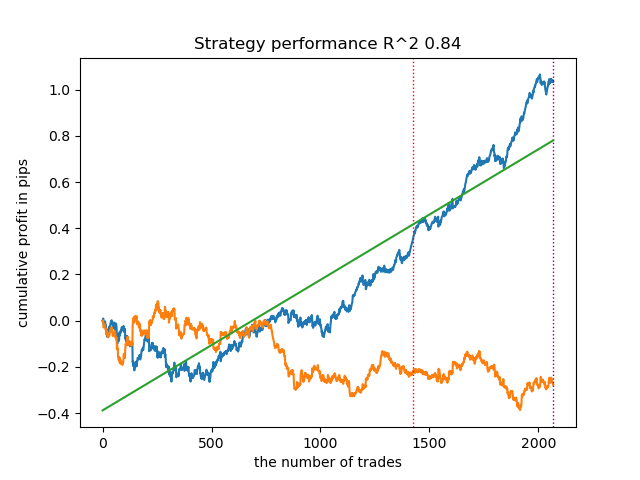
Los resultados han sido de los mejores. Por supuesto, para una comparación más detallada, deberíamos hacer múltiples pruebas con distintos símbolos, pero eso aumentaría demasiado la longitud de un artículo ya de por sí extenso. A continuación le presentamos un cuadro con los resultados obtenidos.
| Algoritmo | Mejor resultado | Resultado medio (25 modelos) |
|---|---|---|
| propensity without matching.py | 0.96 | 0.69 |
| propensity matching naive.py | 0.94 | 0.85 |
| propensity matching original.py | 0.95 | 0.77 |
| propensity matching original.py IPW | 0.97 | 0.84 |
Conclusión
Hoy hemos considerado la posibilidad de utilizar la puntuación de propensión para clasificar series temporales financieras. Este enfoque tiene una buena justificación teórica en cuanto a la ciencia de la inferencia causal, pero también tiene sus defectos. En general, ha producido modelos que conservan sus características con nuevos datos.
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/14360
 Desarrollamos un Asesor Experto multidivisas (Parte 5): Tamaños de posición variables
Desarrollamos un Asesor Experto multidivisas (Parte 5): Tamaños de posición variables
 Filtrado de estacionalidad y período de tiempo para modelos de Deep Learning ONNX con Python para EA
Filtrado de estacionalidad y período de tiempo para modelos de Deep Learning ONNX con Python para EA
 DoEasy. Funciones de servicio (Parte 1): Patrones de precios
DoEasy. Funciones de servicio (Parte 1): Patrones de precios
 Características del Wizard MQL5 que debe conocer (Parte 13): DBSCAN para la clase experta de señales
Características del Wizard MQL5 que debe conocer (Parte 13): DBSCAN para la clase experta de señales
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso