
Propensity Score in der Kausalinferenz
Einführung
Wir setzen unser Eintauchen in die Welt der kausalen Schlussfolgerungen und ihrer modernen Werkzeuge fort. Natürlich ist die Aufgabe etwas weiter gefasst - die Anwendung von Kausalschlussmethoden im Handel. Wir haben bereits mit dem Studium der Grundlagen begonnen und sogar unsere ersten Meta-Learner geschrieben, die sich übrigens als recht robust erwiesen haben. Oder besser gesagt, die Modelle, die mit ihrer Hilfe erstellt wurden, haben sich als robust erwiesen. Es ist erwähnenswert, dass sie die ersten nur für den Leser waren, da sie für den Autor nur ein weiteres Experiment darstellen. Daher gibt es kein Zurück mehr, und wir werden bis zum Ende gehen müssen, bis das gesamte Thema des Kausalschlusses im Handel behandelt ist. Schließlich können die Ansätze zum Kausalschluss unterschiedlich sein, und ich möchte dieses interessante Thema so umfassend wie möglich behandeln.
In diesem Artikel werde ich das Thema Abgleichen (matching) behandeln, das im vorherigen Artikel kurz angesprochen wurde, bzw. eine seiner Varianten, dem Propensity Score matching.
Dies ist wichtig, weil wir einen bestimmten Satz von gekennzeichneten Daten haben, der heterogen ist. Im Devisenhandel beispielsweise kann jedes einzelne Trainingsbeispiel zu einem Bereich mit hoher oder niedriger Volatilität gehören. Außerdem kann es sein, dass einige Beispiele häufiger in der Stichprobe vorkommen, während andere weniger häufig vorkommen. Bei dem Versuch, den durchschnittlichen kausalen Effekt (ATE) in einer solchen Stichprobe zu bestimmen, werden wir unweigerlich auf verzerrte Schätzungen stoßen, wenn wir davon ausgehen, dass alle Beispiele in der Stichprobe die gleiche Tendenz haben, eine Behandlung durchzuführen. Bei dem Versuch, einen konditionalen durchschnittlichen Behandlungseffekt (CATE) zu ermitteln, können wir auf ein Problem stoßen, das als „Fluch der Dimensionen“ bezeichnet wird.
Matching ist eine Familie von Methoden zur Schätzung kausaler Effekte durch den Abgleich ähnlicher Beobachtungen (oder Einheiten) in Behandlungs- und Kontrollgruppen. Der Zweck des Abgleichens besteht darin, Vergleiche zwischen ähnlichen Einheiten anzustellen, um eine möglichst genaue Schätzung der wahren kausalen Wirkung zu erhalten.
Einige Autoren der Kausalschlussliteratur schlagen vor, dass das Abgleichen als ein Schritt der Datenvorverarbeitung betrachtet werden sollte, auf den jeder beliebige Schätzer (z. B. Meta-Learner) aufgesetzt werden kann. Wenn wir genügend Daten haben, um möglicherweise einige Beobachtungen zu verwerfen, ist ein Abgleich als Vorverarbeitungsschritt in der Regel sinnvoll.
Stellen Sie sich vor, Sie haben einen zu analysierenden Datensatz. Diese Daten enthalten 1000 Beobachtungen. Wie hoch ist die Wahrscheinlichkeit, dass Sie für jede Zeile mindestens eine exakte Übereinstimmung finden, wenn Ihr Datensatz 18 Variablen enthält? Die Antwort hängt natürlich von einer Reihe von Faktoren ab. Wie viele Variablen sind binär? Wie viele davon sind kontinuierlich? Wie viele davon sind kategorisch? Wie viele Stufen haben kategoriale Variablen? Sind die Variablen unabhängig oder miteinander korreliert?
Aleksander Molak gibt ein gutes visuelles Beispiel in seinem Buch „Causal inference and discovery in Python“.
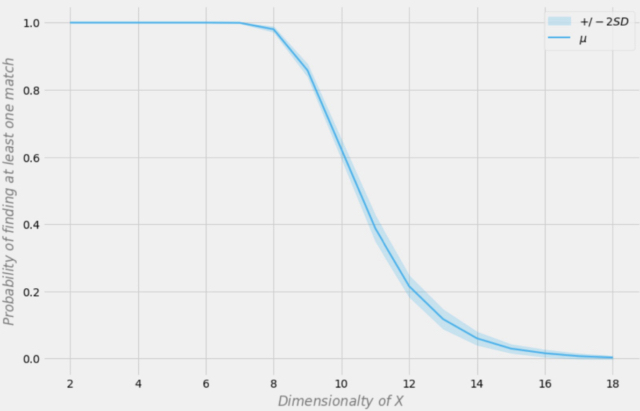
Wahrscheinlichkeit, eine exakte Übereinstimmung zu finden, abhängig von der Dimension des Datensatzes
Gehen wir davon aus, dass unsere Stichprobe 1000 Beobachtungen umfasst. In der obigen Abbildung steht die X-Achse für die Dimension des Datensatzes (die Anzahl der Variablen im Datensatz) und die Y-Achse für die Wahrscheinlichkeit, mindestens eine exakte Übereinstimmung in jeder Zeile zu finden.
Die blaue Linie ist die durchschnittliche Wahrscheinlichkeit und die schattierten Bereiche stellen +/- zwei Standardabweichungen dar. Der Datensatz wurde unter Verwendung unabhängiger Bernoulli-Verteilungen mit p = 0,5 erstellt. Daher ist jede Variable binär und unabhängig.
Ich beschloss, diese Aussage aus dem Buch zu testen und schrieb ein Python-Skript, das diese Wahrscheinlichkeit berechnet.
import numpy as np import matplotlib.pyplot as plt def calculate_exact_match_probability(dimensions): num_samples = 1000 num_trials = 1000 match_count = 0 for _ in range(num_trials): dataset = np.random.randint(2, size=(num_samples, dimensions)) row_sums = np.sum(dataset, axis=1) if any(row_sums == dimensions): match_count += 1 return match_count / num_trials def plot_probability_curve(max_dimensions): dimensions_range = list(range(1, max_dimensions + 1)) probabilities = [calculate_exact_match_probability(dim) for dim in dimensions_range] mean_probability = np.mean(probabilities) std_dev = np.std(probabilities) plt.plot(dimensions_range, probabilities, label='Exact Match Probability') plt.axhline(mean_probability, color='blue', linestyle='--', label='Mean Probability') plt.xlabel('Number of Variables') plt.ylabel('Probability') plt.title('Exact Match Probability in Binary Dataset') plt.legend() plt.show()
Bei 1000 Beobachtungen stimmten die Wahrscheinlichkeiten sogar überein. Versuchsweise können Sie sie für die Dimensionen Ihrer Datensätze selbst berechnen. Für unsere weitere Arbeit genügt es zu verstehen, dass die Fähigkeit zur Verallgemeinerung solcher Daten durch den Klassifikator eingeschränkt ist, wenn die Daten im Vergleich zur Anzahl der Trainingsbeispiele zu viele Merkmale (Kovariaten) enthalten. Je größer die Datenmenge ist, desto genauer sind die statistischen Schätzungen.
*In der Praxis ist dies nicht immer der Fall, da die Bedingung u.i.v (unabhängig und identisch verteilt) erfüllt sein sollte.
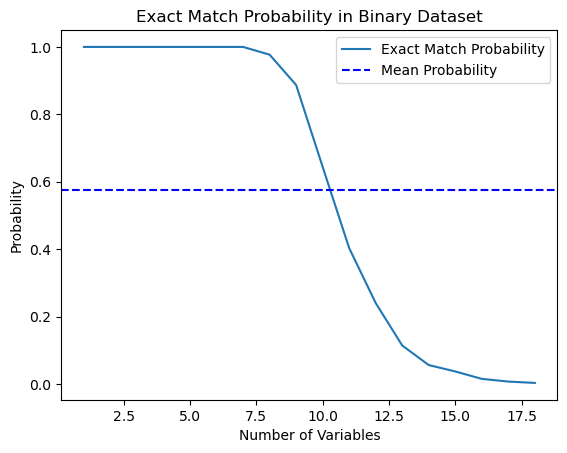
Wie Sie sehen, ist die Wahrscheinlichkeit, in einem 18-dimensionalen binären Zufallsdatensatz eine exakte Übereinstimmung zu finden, gleich Null. In der Praxis arbeiten wir selten mit rein binären Datensätzen, und bei kontinuierlichen Daten wird die mehrdimensionale Zuordnung noch schwieriger. Dies stellt ein ernsthaftes Problem für den Abgleich dar, selbst im ungefähren Fall. Wie können wir dieses Problem lösen?
Verringerung der Dimensionalität der Daten durch Propensity Score
Wir können das Problem des Fluches der Dimensionalität mit Hilfe von Propensity Score lösen. Propensity Scores sind Schätzungen der Wahrscheinlichkeit, dass eine bestimmte Einheit aufgrund ihrer Merkmale der Versuchsgruppe zugewiesen wird. Nach dem Theorem von Propensity Score (Rosenbaum und Rubin, 1983) gilt: Wenn wir für das Merkmal X unbestätigte Daten haben, dann haben wir auch keine Bestätigung aufgrund des Propensity Scores, wobei Positivität angenommen wird. Positiv bedeutet, dass sich behandelte und unbehandelte Verteilungen überschneiden sollten. Dies ist die Positivitätsannahme für kausale Schlüsse. Dies ist auch intuitiv sinnvoll. Wenn sich die Testgruppe und die Kontrollgruppe nicht überschneiden, bedeutet dies, dass sie sehr unterschiedlich sind und wir die Wirkung der einen Gruppe nicht auf die andere extrapolieren können. Diese Extrapolation ist nicht unmöglich (die Regression macht es möglich), aber sie ist sehr gefährlich. Dies ist vergleichbar mit der Erprobung eines neuen Medikaments in einem Experiment, bei dem nur Männer die Behandlung erhalten und dann davon ausgegangen wird, dass Frauen ebenso gut darauf ansprechen werden. Formal wird der Propensity Score wie folgt geschrieben:
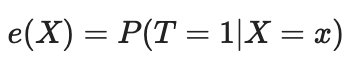
In einer perfekten Welt würden wir einen echten Propensity Score haben. In der Praxis ist der Mechanismus für die Zuweisung der Behandlung jedoch unbekannt, und wir müssen die tatsächliche Veranlagung durch ihre Bewertung oder Erwartung ersetzen. Eine gängige Methode ist die logistische Regression, aber auch andere Techniken des maschinellen Lernens, wie z. B. Gradient Boosting, können verwendet werden (obwohl dies einige zusätzliche Schritte erfordert, um eine Überanpassung zu vermeiden).
Wir können diese Gleichung verwenden, um das Problem der Mehrdimensionalität zu lösen. Propensity Scores sind univariat, sodass wir jetzt nur zwei Werte und keine multivariaten Vektoren abgleichen können.
Wenn wir also die bedingte Unabhängigkeit der potenziellen Ergebnisse von der Behandlung haben,
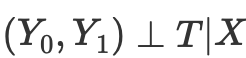
dann können wir den durchschnittlichen kausalen Effekt für kontinuierliche und diskrete Fälle beim Abgleich ohne Propensity Score berechnen:
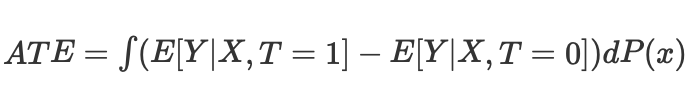
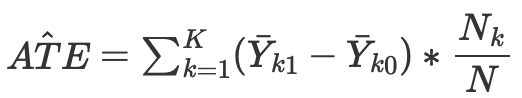
wobei Nk die Anzahl der Beobachtungen in jeder Zelle ist. Eine Zelle ist eine Untergruppe von Beobachtungen, die nach einer bestimmten Proximity-Metrik abgeglichen werden.
Der Propensity Score ergibt sich jedoch aus der Erkenntnis, dass wir nicht direkt X Störfaktoren kontrollieren müssen, um bedingte Unabhängigkeit zu erreichen
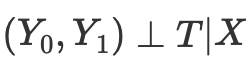
Stattdessen genügt es, den Balance-Indikator zu kontrollieren
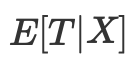
Der Propensity Score ermöglicht es, unabhängig von X als Ganzes zu sein, um sicherzustellen, dass die potenziellen Ergebnisse unabhängig von der Behandlung sind. Es reicht aus, diese eine Variable, den Propensity Score, zur Bedingung zu machen:
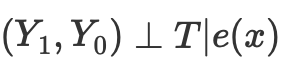
In den Quellen zum Kausalschluss werden eine Reihe von Problemen mit diesem Ansatz beschrieben:
- Erstens reduzieren Propensity Scores die Dimensionalität unserer Daten und zwingen uns per Definition, einige Informationen zu verwerfen.
- Zweitens können zwei Beobachtungen, die im ursprünglichen Merkmalsraum sehr unterschiedlich sind, denselben Propensity Score haben. Dies kann dazu führen, dass sehr unterschiedliche Beobachtungen übereinstimmen und somit die Ergebnisse verfälschen.
- Drittens führt das PSM (Propensity Score Modeling) zu einem Paradoxon. Im binären Fall würde der optimale Propensity Score 0,5 betragen. Was passiert in einem perfekten Szenario, in dem alle Beobachtungen einen optimalen Propensity Score von 0,5 haben? Die Position jeder Beobachtung im Propensity-Score-Raum wird mit jeder anderen Beobachtung identisch. Dies wird manchmal als PSM-Paradoxon bezeichnet.
Propensity Score Matching und verwandte Methoden
Es gibt eine Reihe von Methoden für das Abgleichen (matching) von Einheiten auf der Grundlage des Propensity Score. Die wichtigste Methode ist die Methode der nächsten Nachbarn, die jede Einheit i in der Versuchsgruppe mit der Einheit j in der Kontrollgruppe mit dem nächstgelegenen absoluten Abstand zwischen ihren Propensity Scores vergleicht, ausgedrückt als
d(i, j) = minj{|e(Xi) – e(Xj)|}.
Alternativ dazu wird beim Schwellenwert-Abgleichen jede Einheit i in der Behandlungsgruppe mit einer Einheit j in der Kontrollgruppe innerhalb eines vorgegebenen Schwellenwerts b; verglichen, d. h.
d(i, j) = minj{|e(Xi) – e(Xj)| <b}.
Es wird empfohlen, dass der vorgegebene Schwellenwert b weniger als oder gleich 0,25 Standardabweichungen des Propensity Scores beträgt. Andere Forscher vertreten die Auffassung, dass b = 0,20 der Standardabweichung der Propensity Scores optimal ist.
Eine andere Möglichkeit des Schwellenwert-Abgleichens ist der Radius-Abgleich, der ein Eins-zu-Vielen-Abgleich ist und jede Einheit i in der Behandlungsgruppe mit mehreren Einheiten in der Kontrollgruppe innerhalb des vordefinierten Bereichs b abgleicht; das heißt
d(i, j) = {|e(Xi) – e(Xj)| <b}.
Zu den weiteren Propensity-Score-Abgleichmethoden gehört die Mahalanobis-Metrik. Beim Abgleich mit der Mahalanobis-Metrik wird jede Einheit i in der Versuchsgruppe mit der Einheit j in der Kontrollgruppe verglichen, wobei der nächstgelegene Mahalanobis-Abstand auf der Grundlage der Nähe der Variablen berechnet wird.
Die bisher besprochenen Propensity-Score-Abgleichmethoden können entweder mit einem Algorithmus des gierigen Abgleichens oder einem optimalen Abgleichalgorithmus umgesetzt werden.
- Bei gierigem Abgleich können die übereinstimmenden Einheiten nicht mehr geändert werden, sobald der Abgleich erfolgt ist. Jedes Paar aufeinander abgestimmter Einheiten ist das beste derzeit verfügbare Paar.
- Bei optimalem Abgleich können frühere abgeglichene Einheiten geändert werden, bevor der aktuelle Abgleich durchgeführt wird, um einen minimalen oder optimalen Gesamtabstand zu erreichen.
- Beide Abgleichsalgorithmen liefern in der Regel die gleichen Abgleichsdaten, wenn die Kontrollgruppe groß ist. Ein optimaler Abgleich führt jedoch zu kleineren Gesamtabständen innerhalb der abgeglichenen Einheiten. Wenn das Ziel also einfach darin besteht, gut übereinstimmende Gruppen zu finden, kann ein gieriger Abgleich ausreichend sein. Wenn das Ziel stattdessen darin besteht, gut übereinstimmende Paare zu finden, ist ein optimaler Abgleich möglicherweise vorzuziehen.
Es gibt Methoden, die mit dem Propensity-Score-Abgleich verbunden sind und die nicht streng auf die einzelnen Stichprobeneinheiten abgestimmt sind. Bei der Subklassifizierung (oder Stratifizierung) werden beispielsweise alle Einheiten der gesamten Stichprobe auf der Grundlage der entsprechenden Anzahl von Propensity-Score-Perzentilen in mehrere Schichten eingeteilt und die Einheiten der jeweiligen Schicht zugeordnet. Es wurden fünf Schichten beobachtet, die bis zu 90 % der Selektionsverzerrungen beseitigen.
Eine besondere Art der Unterklassifizierung ist die vollständige Anpassung, bei der Unterklassen auf optimale Weise erstellt werden. Eine vollständig abgeglichene Stichprobe besteht aus abgeglichenen Teilmengen, wobei jede abgeglichene Menge eine Versuchseinheit und eine oder mehrere Kontrolleinheiten bzw. eine Kontrolleinheit und eine oder mehrere Versuchseinheiten enthält. Ein vollständiger Abgleich ist optimal im Hinblick auf die Minimierung des gewichteten Durchschnitts des geschätzten Abstandsmaßes zwischen jedem Behandlungsteilnehmer und jedem Kontrollteilnehmer in jeder Unterklasse.
Eine weitere Methode, die mit dem Propensity-Score-Abgleich verbunden ist, ist das Kernel-Matching (oder lokale lineare Matching), der Abgleich und Ergebnisanalyse in einem einzigen Verfahren mit einem One-to-All-Abgleich kombiniert.
Trotz der Vielfalt der vorgeschlagenen Vergleichsmethoden hängt die Effizienz ihrer Anwendung mehr von der richtigen Formulierung des Problems als von einer bestimmten Methode ab.
Starke Vernachlässigungsannahme
„Starke Vernachlässigung“ ist eine entscheidende Annahme bei der Konstruktion des Propensity Score, der darauf abzielt, kausale Effekte in Beobachtungen zu schätzen, bei denen die Behandlungszuweisung zufällig ist. Im Wesentlichen bedeutet dies, dass die Zuweisung der Behandlung unabhängig von den potenziellen Ergebnissen ist, die sich aus den beobachteten Basiskovariaten (Merkmalen) ergeben.
Schauen wir uns das einmal genauer an:
- Verteilung der Behandlung: Dies gilt unabhängig davon, ob eine Einheit eine Behandlung erhält oder nicht (z. B. die Einnahme eines neuen Medikaments oder die Teilnahme an einem Programm).
- Mögliche Ergebnisse: Dies sind die Ergebnisse, die eine Einheit sowohl unter den Behandlungs- als auch unter den Kontrollbedingungen erfahren würde, aber wir können nur eines für jede Einheit beobachten.
- Kovariate der Ausgangssituation: Dabei handelt es sich um Merkmale der Einheit, die vor der Zuteilung der Behandlung gemessen werden und die sowohl die Wahrscheinlichkeit, eine Behandlung zu erhalten, als auch das Ergebnis beeinflussen können.
Die „stark ignorierbare“ Annahme besagt, dass:
- Es gibt keine unbeobachteten Störfaktoren: Es gibt keine unbeobachteten Variablen, die die Zuweisung der Behandlung oder das Ergebnis beeinflussen. Dies ist wichtig, weil unbeobachtete Störfaktoren den geschätzten Behandlungseffekt verzerren können.
- Positivität: Jede Einheit hat eine von Null verschiedene Wahrscheinlichkeit, sowohl die Behandlung als auch die Kontrolle zu erhalten, wenn man die beobachteten Kovariaten berücksichtigt. Auf diese Weise wird sichergestellt, dass in den zu vergleichenden Gruppen genügend Einheiten vorhanden sind, um aussagekräftige Vergleiche anzustellen.
Wenn diese Bedingungen erfüllt sind, führt die Konditionierung auf den Propensity Score (die geschätzte Wahrscheinlichkeit bei gegebenen Kovariaten eine Behandlung zu erhalten) zu unverzerrten Schätzungen des durchschnittlichen Behandlungseffekts (ATE). Der ATE stellt den mittleren Unterschied zwischen den Ergebnissen der Behandlungs- und der Kontrollgruppe dar, als ob die Behandlung zufällig zugewiesen worden wäre.
Inverse Wahrscheinlichkeitsgewichtung
Die inverse Wahrscheinlichkeitsgewichtung ist einer der Ansätze, bei dem Rauschen oder Störfaktoren eliminiert werden, indem versucht wird, die Beobachtungen in einem Datensatz auf der Grundlage des Kehrwerts der Wahrscheinlichkeit der Behandlungszuweisung neu zu gewichten. Die Idee ist, Beobachtungen, die als weniger wahrscheinlich für eine Behandlung eingestuft werden, mehr Gewicht zu geben, sodass sie repräsentativer für die allgemeine Bevölkerung sind.
- Zunächst wird ein Propensity Score geschätzt, der die Wahrscheinlichkeit angibt, eine Behandlung zu erhalten, wenn die beobachteten Kovariaten berücksichtigt werden.
- Für jede Beobachtung wird ein inverser Propensity Score berechnet.
- Jede Beobachtung wird dann mit ihrem entsprechenden Gewicht multipliziert. Dies bedeutet, dass Beobachtungen mit einer geringeren Wahrscheinlichkeit, die beobachtete Behandlung zu erhalten, mehr Gewicht erhalten.
- Der gewichtete Datensatz wird dann für die Analyse verwendet. Die Gewichte werden sowohl auf die Behandlungs- als auch auf die Kontrollgruppe angewandt, um den potenziellen Einfluss der beobachteten Kovariaten zu berücksichtigen.
Eine inverse Wahrscheinlichkeitsgewichtung kann dazu beitragen, die Verteilung der Kovariaten zwischen Behandlungs- und Kontrollgruppen auszugleichen und so die Verzerrung bei der Schätzung der kausalen Effekte zu verringern. Sie basiert jedoch auf der Annahme, dass alle relevanten Störvariablen gemessen und in das zur Schätzung des Propensity Score verwendete Modell einbezogen werden. Darüber hinaus hängt der Erfolg von IPW wie bei jeder statistischen Methode von der Qualität des Modells ab, das zur Schätzung des Propensity Score verwendet wird.
Die inverse Propensity-Score-Gleichung lautet wie folgt:
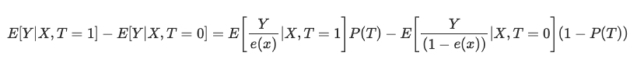
Ohne ins Detail zu gehen, können Sie die beiden Terme der Gleichung vergleichen. Das linke Bild ist für die Behandlungsgruppe, das rechte für die Kontrollgruppe. Die Gleichung zeigt, dass ein einfacher Vergleich von Durchschnittswerten einem Vergleich von invers gewichteten Durchschnittswerten entspricht. Auf diese Weise entsteht eine Population, die genauso groß ist wie die ursprüngliche Population, bei der aber alle auf der linken Seite die Behandlung erhalten. Aus denselben Gründen betrachtet der richtige die unbehandelten und legt Wert auf die, die wie behandelte aussehen.
Bewertung der Ergebnisse nach dem Abgleich
Bei der kausalen Inferenz wird die Schätzung als ATE oder CATE konstruiert, d. h. als die Differenz der gewichteten Mittelwerte (bereinigt um den Propensity Score) der behandelten und unbehandelten Zielwerte.
Sobald wir den Propensity Score als e(x) erhalten haben, können wir diese Werte z. B. für das Training eines anderen Klassifikators anstelle der ursprünglichen Werte der X-Merkmale verwenden. Wir können auch Stichproben anhand ihres Propensity Scores vergleichen, um sie in Schichten zu unterteilen. Eine weitere Möglichkeit besteht darin, e(x) als separates Merkmal beim Training des endgültigen Auswerters hinzuzufügen, was dazu beiträgt, verzerrte Schätzungen aufgrund von Störungen zu vermeiden, wenn verschiedene Beispiele in der Stichprobe unterschiedliche Schätzungen nach dem Propensity Score aufweisen.
Wir sind daran interessiert, Untergruppen zu finden, die gut oder schlecht auf die Behandlung ansprechen (Modelltraining), und dann den endgültigen Klassifikator nur auf Daten zu trainieren, die gut trainiert werden können (der Klassifikationsfehler ist minimal). Dann sollten wir die schlecht klassifizierten Daten in die zweite Untergruppe einordnen und den zweiten Klassifikator trainieren, um zwischen diesen beiden Untergruppen zu unterscheiden, d. h. um die Spreu vom Weizen zu trennen oder die Untergruppen zu ermitteln, die am ehesten einer Behandlung zugänglich sind. Daher werden wir jetzt nicht die gesamte Propensity-Score-Methode anwenden, sondern die Stichproben entsprechend den vom trainierten Klassifikator ermittelten Wahrscheinlichkeiten anpassen, während die Gesamtbewertung des ATE (durchschnittlicher Behandlungseffekt) für uns von geringem Interesse ist.
Mit anderen Worten: Wir stützen unsere Bewertung auf die Ergebnisse des Algorithmus, der auf neuen Daten läuft, die nicht am Training teilgenommen haben. Darüber hinaus sind wir an der durchschnittlichen Geschwindigkeit einer Reihe von Modellen interessiert, die auf randomisierten Daten trainiert wurden. Je höher die durchschnittliche Punktzahl der unabhängigen Modelle ist, desto mehr Vertrauen haben wir in jedes einzelne Modell.
Weiter zu den Experimenten
Als ich mit diesem Artikel begann, war mir bewusst, dass dieses ausführliche Material für viele Händler, insbesondere für diejenigen, die mit maschinellem Lernen nicht vertraut sind, sehr kontraintuitiv erscheinen mag. Als ich mich an meine erste Begegnung mit dem Kausalschluss erinnerte, verletzte dieses anfängliche Missverständnis meinen Stolz so sehr, dass ich einfach nicht anders konnte, als mich mit den Einzelheiten zu befassen. Außerdem konnte ich mir nicht vorstellen, dass ich mir die Freiheit nehmen würde, kausale Schlussfolgerungstechniken zur Klassifizierung von Zeitreihen anzuwenden.
- Die Datei propensity without matching.py
Beginnen wir mit einer Möglichkeit, wie wir Propensity Scores direkt in unseren Schätzer oder Meta-Learner einbauen können. Zu diesem Zweck müssen wir zwei Modelle trainieren. Erstens das PSM (Propensity Score Model) selbst, mit dem wir die Wahrscheinlichkeiten für die Zuordnung von Trainingsbeispielen zu Behandlungs- oder Testgruppen ermitteln können. Die ermittelten Wahrscheinlichkeiten werden zusammen mit den Merkmalen (Kovariaten) in das zweite Modell eingegeben, das die Ergebnisse (Kauf oder Verkauf) vorhersagen wird.
Die Intuition dieses Ansatzes besteht darin, dass der Meta-Learner nun in der Lage ist, Untergruppen von Stichproben auf der Grundlage ihrer Tendenz zur Behandlung zu unterscheiden. Auf diese Weise erhalten wir gewichtete Vorhersagen der Ergebnisse, die genauer sein dürften. Danach werden wir den Datensatz in gut vorhersehbare und schlecht vorhersehbare Fälle unterteilen, wie es in früheren Artikeln der Fall war. In diesem Fall ist ein explizites Stichprobenabgleich nicht erforderlich, da der Meta-Learner den Propensity Score automatisch in seine Schätzungen einbezieht. Dieser Ansatz scheint mir recht bequem zu sein, da das maschinelle Lernen die ganze Arbeit für uns erledigt.
Zunächst erstellen wir die Teilstichproben „train“ und „val“, um das Metamodell zu trainieren. Da der Meta-Learner auf der Unterstichprobe trainiert wird, erstellen wir ein Zielpaar y_T1, y_T0 und füllen es mit Einsen und Nullen. Dies hängt davon ab, ob die Einheiten eine Behandlung (Modellschulung) erhalten haben oder nicht. Dann wird die Unterauswahl, deren Zielvariablen Behandlungen sind, neu gemischt.
X_train, X_val, y_train, y_val = train_test_split( X, y, train_size = 0.5, test_size = 0.5, shuffle = True) # randomly assign treated and control y_T1 = pd.DataFrame(y_train) y_T1['T'] = 1 y_T1 = y_T1.drop(['labels'], axis=1) y_T0 = pd.DataFrame(y_val) y_T0['T'] = 0 y_T0 = y_T0.drop(['labels'], axis=1) y_TT = pd.concat([y_T1, y_T0]) y_TT = y_TT.sort_index() X_trainT, X_valT, y_trainT, y_valT = train_test_split( X, y_TT, train_size = 0.5, test_size = 0.5, shuffle = True)
Der nächste Schritt besteht darin, das PSM-Modell zu trainieren, um vorherzusagen, ob die Proben zu den Behandlungs- oder Kontrollteilstichproben gehören.
# fit propensity model PSM = CatBoostClassifier(iterations = iterations, depth = depth, custom_loss = ['Accuracy'], eval_metric = 'Accuracy', use_best_model=True, early_stopping_rounds=15, verbose = False).fit(X_trainT, y_trainT, eval_set = (X_valT, y_valT), plot = False)
Dann müssen wir Vorhersagen über die Zugehörigkeit zu den Behandlungs- und Kontrollgruppen erhalten und diese zu den Merkmalen des Meta-Learners hinzufügen, um ihn dann zu trainieren.
# predict probabilities train_proba = PSM.predict_proba(X_train)[:, 1] val_proba = PSM.predict_proba(X_val)[:, 1] # fit meta-learner meta_m = CatBoostClassifier(iterations = iterations, depth = depth, custom_loss = ['Accuracy'], eval_metric = 'Accuracy', verbose = False, use_best_model = True, early_stopping_rounds=15).fit(X_train.assign(T=train_proba), y_train, eval_set = (X_val.assign(T=val_proba), y_val), plot = False)
In der letzten Phase erhalten wir die Vorhersagen des Meta-Learners, vergleichen die vorhergesagten Bezeichnungen mit den tatsächlichen und füllen das Buch der schlechten Beispiele aus.
# create daatset with predicted values predicted_PSM = PSM.predict_proba(X)[:,1] X_psm = X.assign(T=predicted_PSM) coreset = X.assign(T=predicted_PSM) coreset['labels'] = y coreset['labels_pred'] = meta_m.predict_proba(X_psm)[:,1] coreset['labels_pred'] = coreset['labels_pred'].apply(lambda x: 0 if x < 0.5 else 1) # add bad samples of this iteration (bad labels indices) coreset_b = coreset[coreset['labels']==0] coreset_s = coreset[coreset['labels']==1] diff_negatives_b = coreset_b['labels'] != coreset_b['labels_pred'] diff_negatives_s = coreset_s['labels'] != coreset_s['labels_pred'] BAD_BUY = BAD_BUY.append(diff_negatives_b[diff_negatives_b == True].index) BAD_SELL = BAD_SELL.append(diff_negatives_s[diff_negatives_s == True].index)
Versuchen wir nun, 25 Modelle zu trainieren, und betrachten wir die besten und durchschnittlichen Ergebnisse.
options = [] best_res = 0.0 for i in range(25): print('Learn ' + str(i) + ' model') options.append(learn_final_models(meta_learners(models_number=5, iterations=25, depth=2, bad_samples_fraction=0.5))) if options[-1][0] > best_res: best_res = options[-1][0] print("BEST: " + str(best_res)) options.sort(key=lambda x: x[0]) test_model(options[-1][1:], plt=True) test_all_models(options)
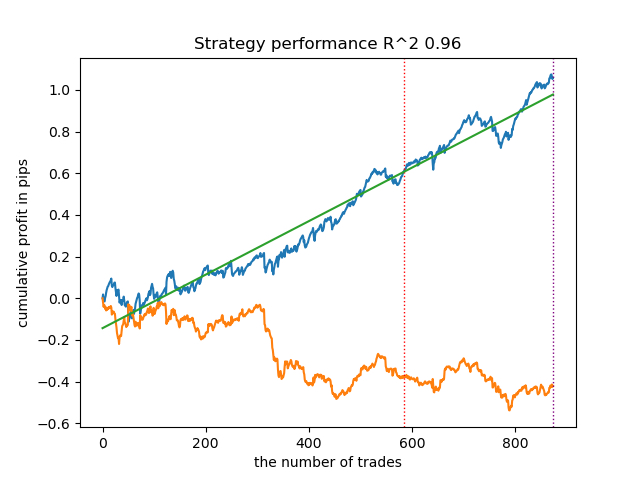
Ich habe eine Reihe solcher Schulungen durchgeführt und bin zu dem Schluss gekommen, dass sich dieser Ansatz in Bezug auf die Qualität der Modelle praktisch nicht von meinem im vorherigen Artikel beschriebenen Ansatz unterscheidet. Beide Optionen sind in der Lage, gute Modelle zu erstellen, die OOS bestehen. Dies sollte uns nicht überraschen, da wir e(x) als zusätzliches Merkmal verwendet haben und der Rest des Algorithmus unverändert blieb.
- Die Datei propensity matching naive.py
Bei der Erwägung von Umsetzungsmethoden können wir nie im Voraus wissen, welche Methode besser funktionieren wird. Als Experiment beschloss ich, einen Abgleich nicht nach der Tendenz, die Behandlung zuzuordnen, sondern nach der Tendenz, die Zielkennzeichnungen vorherzusagen, durchzuführen. Dies sollte für den Leser intuitiver erscheinen. Der Hauptunterschied besteht darin, dass wir nur ein (herkömmlich genanntes) PSM-Modell trainieren. Anschließend werden die Wahrscheinlichkeiten vorhergesagt und eine Liste von Bins entsprechend der Anzahl der Schichten erstellt, in die die ermittelten Wahrscheinlichkeiten unterteilt werden sollen. Für jede Schicht wird die Anzahl der richtig/falsch erratenen Ergebnisse gezählt. Danach wird für die Schichten, in denen die Anzahl der falsch erratenen Beispiele (multipliziert mit einem Verhältnis) die Anzahl der richtig erratenen Beispiele übersteigt, die Bedingung für die Aufnahme der schlechten Beispiele in das Buch der schlechten Beispiele ausgelöst.
bins = np.linspace(lower_bound, upper_bound, num=bins_number) coreset['bin'] = pd.cut(coreset['propensity'], bins) for val in range(len(coreset['bin'].unique())): values = coreset.loc[coreset['bin'] == coreset['bin'].unique()[val]] diff_negatives = values['labels'] != values['labels_pred'] if len(diff_negatives[diff_negatives == False]) < (len(diff_negatives[diff_negatives == True]) * coefficient): B_S_B = B_S_B.append(diff_negatives[diff_negatives == True].index)
Dieser Ansatz hat zusätzliche Parameter:
- bins_number - Anzahl der Bins
- lower_bound - untere Wahrscheinlichkeitsgrenze, aus der die Schichten berechnet werden
- upper_bound - obere Wahrscheinlichkeitsgrenze, bis zu der die Schichten berechnet werden
Da wir einen Meta-Lerner mit geringer Tiefe verwenden, gruppieren sich die Wahrscheinlichkeiten normalerweise um 0,5 und erreichen nur selten extreme Grenzen. Daher können wir Extremwerte als uninformativ verwerfen, indem wir Ober- und Untergrenzen festlegen.
Lassen Sie uns 25 Modelle trainieren und die Ergebnisse betrachten. Ich möchte anmerken, dass alle Modelle auf demselben Datensatz trainiert wurden, sodass ihr Vergleich durchaus sinnvoll ist.
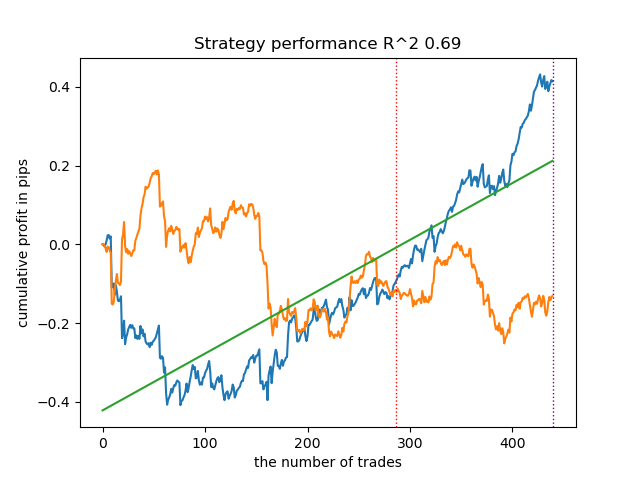
options = [] best_res = 0.0 for i in range(25): print('Learn ' + str(i) + ' model') options.append(learn_final_models(meta_learners(models_number=5, iterations=25, depth=2, bad_samples_fraction=0.5, bins_number=25, lower_bound=0.3, upper_bound=0.7, coefficient=1.0))) if options[-1][0] > best_res: best_res = options[-1][0] print("BEST: " + str(best_res)) options.sort(key=lambda x: x[0]) test_model(options[-1][1:], plt=True)
Überraschenderweise funktionierte diese spontane Implementierung bei den neuen Daten recht gut. Nachfolgend finden Sie Handelsdiagramme für das beste Modell und alle 25 Modelle auf einmal.
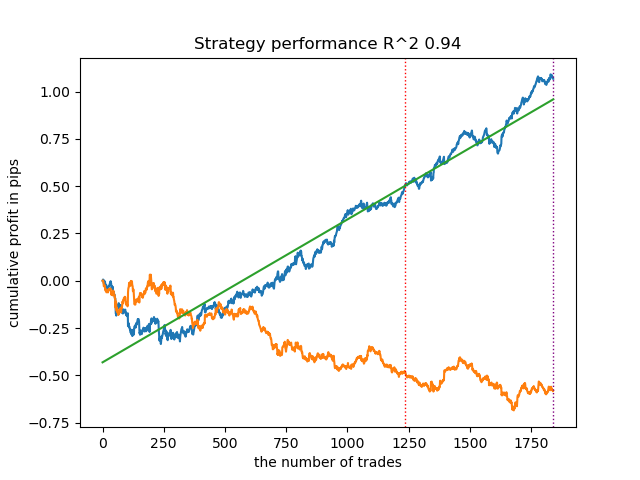
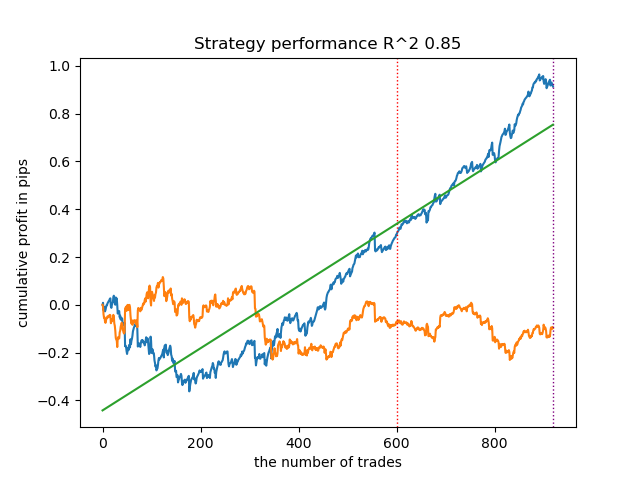
- Die Datei propensity matching original.py
Wir fahren fort mit der Umsetzung des Beispiels, das der Theorie am nächsten kommt und bei dem die Annahme der starken Vernachlässigung (stark ignorierbar) erfüllt ist. Ich möchte Sie daran erinnern, dass „stark ignorierbar“ bedeutet, dass die Zuteilung der Behandlung in keiner Weise von den möglichen Ergebnissen abhängt, was bedeutet, dass sie völlig zufällig ist und es keine nicht berücksichtigten Variablen gibt, die die Verzerrung beeinflussen. Zu diesem Zweck werden wir nach dem Zufallsprinzip eine Behandlung zuweisen und das PSM-Modell trainieren. Dann trainieren wir den Meta-Schätzer, um die Ergebnisse von Handelsgeschäften (Klassenkennzeichnungen) vorherzusagen. Danach schichten wir die Stichprobe nach dem Propensity Score und fügen dem Buch der schlechten Beispiele nur Stichproben aus den Bereichen hinzu, in denen die Zahl der erfolglosen Vorhersagen die Zahl der erfolgreichen übersteigt, wobei wir das Verhältnis berücksichtigen.
Ich habe auch die Möglichkeit hinzugefügt, die im theoretischen Teil beschriebene IPW (inverse Wahrscheinlichkeitsgewichtung) zu verwenden.
Nach dem Training von zwei Klassifikatoren wird der folgende Code ausgeführt.
# create daatset with predicted values coreset = X.copy() coreset['labels'] = y coreset['propensity'] = PSM.predict_proba(X)[:, 1] if Use_IPW: coreset['propensity'] = coreset['propensity'].apply(lambda x: 1 / x if x > 0.5 else 1 / (1 - x)) coreset['propensity'] = coreset['propensity'].round(3) coreset['labels_pred'] = meta_m.predict_proba(X)[:, 1] coreset['labels_pred'] = coreset['labels_pred'].apply(lambda x: 0 if x < 0.5 else 1) bins = np.linspace(lower_bound, upper_bound, num=bins_number) coreset['bin'] = pd.cut(coreset['propensity'], bins) for val in range(len(coreset['bin'].unique())): values = coreset.loc[coreset['bin'] == coreset['bin'].unique()[val]] diff_negatives = values['labels'] != values['labels_pred'] if len(diff_negatives[diff_negatives == False]) < (len(diff_negatives[diff_negatives == True]) * coefficient): B_S_B = B_S_B.append(diff_negatives[diff_negatives == True].index)
Lassen Sie uns 25 Modelle ohne IPW trainieren und schauen wir uns die beste Saldenkurve und die über alle Modelle gemittelte Kurve mit den folgenden Einstellungen an:
options = [] best_res = 0.0 for i in range(25): print('Learn ' + str(i) + ' model') options.append(learn_final_models(meta_learners(models_number=5, iterations=25, depth=2, bad_samples_fraction=0.5, bins_number=25, lower_bound=0.3, upper_bound=0.7, coefficient=1.5, Use_IPW=False))) if options[-1][0] > best_res: best_res = options[-1][0] print("BEST: " + str(best_res))
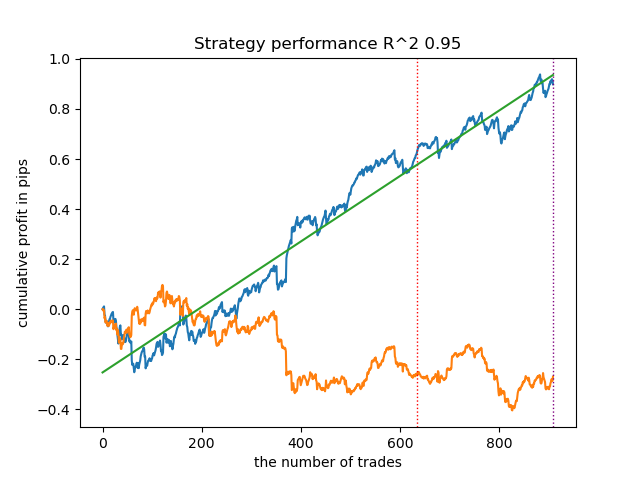
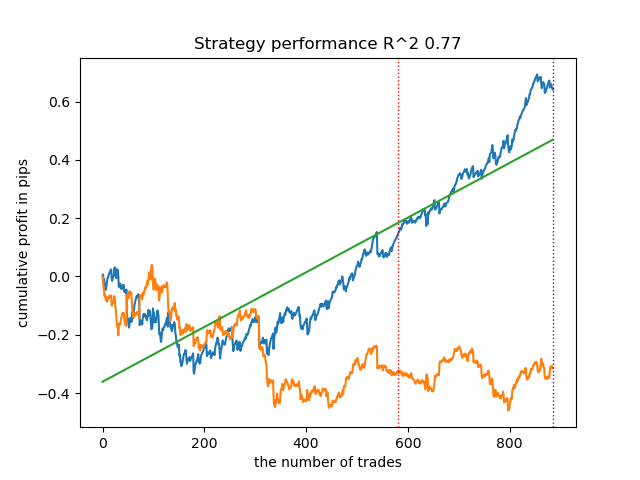
Im Allgemeinen sind die Ergebnisse mit denen früherer Umsetzungen vergleichbar. Nun wollen wir dasselbe mit aktiviertem IPW tun.
options = [] best_res = 0.0 for i in range(25): print('Learn ' + str(i) + ' model') options.append(learn_final_models(meta_learners(models_number=5, iterations=25, depth=2, bad_samples_fraction=0.5, bins_number=25, lower_bound=0.1, upper_bound=10.0, coefficient=1.5, Use_IPW=True))) if options[-1][0] > best_res: best_res = options[-1][0] print("BEST: " + str(best_res))

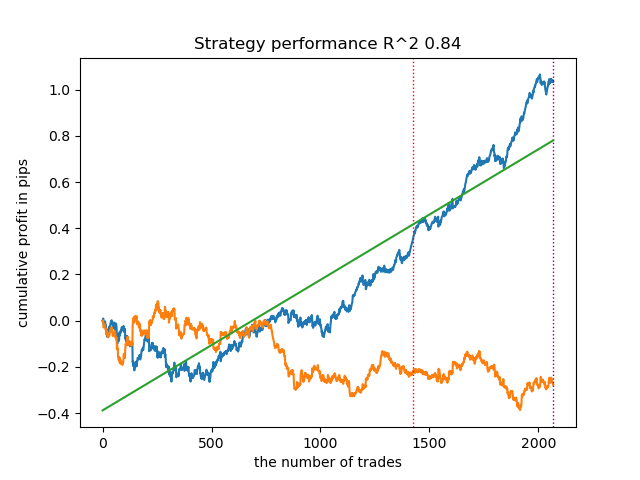
Die Ergebnisse waren mit die besten. Für einen detaillierteren Vergleich ist es natürlich notwendig, mehrere Tests mit verschiedenen Symbolen durchzuführen, aber dies würde den Umfang eines ohnehin schon umfangreichen Artikels erhöhen. Die Tabelle mit den Ergebnissen ist nachstehend aufgeführt.
| Der Algorithmus | Bestes Ergebnis | Durchschnittliches Ergebnis (25 Modelle) |
|---|---|---|
| propensity without matching.py | 0.96 | 0.69 |
| propensity matching naive.py | 0.94 | 0.85 |
| propensity matching original.py | 0.95 | 0.77 |
| propensity matching original.py IPW | 0.97 | 0.84 |
Schlussfolgerung
Wir haben die Möglichkeit in Betracht gezogen, den Propensity Score für das Problem der Klassifizierung von Finanzzeitreihen zu verwenden. Dieser Ansatz hat eine gute theoretische Rechtfertigung im Bereich der Kausalschlüsse, aber er hat auch seine Schwächen. Im Allgemeinen konnten wir so Modelle erhalten, die ihre Eigenschaften auch bei neuen Daten beibehalten.
Übersetzt aus dem Russischen von MetaQuotes Ltd.
Originalartikel: https://www.mql5.com/ru/articles/14360
 Neuronale Netze leicht gemacht (Teil 79): Feature Aggregated Queries (FAQ) im Kontext des Staates
Neuronale Netze leicht gemacht (Teil 79): Feature Aggregated Queries (FAQ) im Kontext des Staates
 DoEasy. Dienst-Funktionen (Teil 1): Preismuster
DoEasy. Dienst-Funktionen (Teil 1): Preismuster
 Eine alternative Log-datei mit der Verwendung der HTML und CSS
Eine alternative Log-datei mit der Verwendung der HTML und CSS
 Algorithmen zur Optimierung mit Populationen: Widerstand gegen das Steckenbleiben in lokalen Extremen (Teil I)
Algorithmen zur Optimierung mit Populationen: Widerstand gegen das Steckenbleiben in lokalen Extremen (Teil I)
- Freie Handelsapplikationen
- Über 8.000 Signale zum Kopieren
- Wirtschaftsnachrichten für die Lage an den Finanzmärkte
Sie stimmen der Website-Richtlinie und den Nutzungsbedingungen zu.