Algoritmos de optimización de la población: Algoritmo genético binario (Binary Genetic Algorithm, BGA). Parte II
Contenido:
1. Introducción
2. Descripción del algoritmo
3. Resultados de las pruebas
1. Introducción
En el artículo anterior, introdujimos todos los conceptos y métodos básicos importantes utilizados no solo en los algoritmos genéticos sino, de un modo u otro, en cualquier algoritmo de optimización de la población. Ahora, basándonos en estos conocimientos, podremos examinar con detalle el tema principal del presente "tratado" en dos volúmenes: el algoritmo genético binario (BGA). Empezando por la información general, pasaremos al código de esta notable estrategia de búsqueda, y concluiremos con los resultados de la prueba.
El algoritmo genético (GA) pertenece a los algoritmos evolutivos que incluyen varios enfoques como algoritmos genéticos, programación genética, estrategias evolutivas y otros. Se basan en las ideas de evolución y herencia, donde las soluciones se representan como una población y se aplican operadores genéticos como el cruce y la mutación para crear nuevas generaciones de soluciones.
Los algoritmos genéticos usan los principios de la selección natural y la genética para resolver problemas de optimización. El algoritmo genético binario (BGA) analizado en este trabajo apareció en primer lugar entre todos los tipos de algoritmos genéticos. Así, el BGA pertenece a la clase de los algoritmos evolutivos y supone una variante particular del algoritmo genético que utiliza una representación binaria de los datos.
Los algoritmos genéticos empezaron a desarrollarse a mediados del siglo XX. Uno de sus fundadores es John Holland, que en 1975 publicó el libro "Adaptation in Natural and Artificial Systems" (Adaptación en sistemas naturales y artificiales), donde introdujo por primera vez el algoritmo genético como toda una dirección de aproximación a la solución de los problemas de optimización.
El desarrollo del algoritmo binario genético se inspiró en varios factores e ideas, las más importantes de las cuales son:
- La selección natural y los principios de la evolución: La BGA se basa en los principios de la selección natural y evolución propuestos por Charles Darwin. La idea dice que en una población hay diversidad de soluciones, y las que estén mejor adaptadas al entorno tendrán más posibilidades de sobrevivir y transmitir sus características a la siguiente generación.
- Genética y herencia: El BGA también utiliza conceptos genéticos como gen, cromosoma y herencia. Las soluciones en el BGA se representan como cadenas binarias, donde los grupos individuales de bits representan genes específicos, mientras que el gen a su vez representa el parámetro a optimizar. Los operadores genéticos, como el cruce y la mutación, se aplican a cadenas binarias para crear nuevas generaciones de soluciones.
En general, el desarrollo del BGA fue resultado de una combinación de ideas procedentes de los campos de los algoritmos evolutivos, la genética y la optimización. Fue creado para resolver problemas de optimización usando los principios de la selección natural y la genética, y su desarrollo prosigue hasta nuestros días; se han creado un gran número de variantes de GA, así como un amplio uso de ideas y enfoques en algoritmos genéticos como parte de algoritmos híbridos, incluyendo algoritmos muy complejos y llenos de distintos componentes.
El algoritmo genético binario, el BGA, utiliza una representación binaria de los datos. Y esto significa que cada individuo (solución) se representa como una cadena de bits (0 y 1). Asimismo, los operadores genéticos como el cruce y la mutación se aplican a cadenas de bits para crear nuevas generaciones de soluciones.
Un dato interesante: los algoritmos genéticos, incluido el BGA, pueden utilizarse para crear y mejorar la inteligencia artificial. Por ejemplo, pueden aplicarse a la evolución de las redes neuronales, posibilitando el desarrollo de modelos de aprendizaje automático más eficientes.
Los algoritmos genéticos en general, y los BGA en particular, representan una poderosa herramienta para resolver problemas de optimización complejos en los que los métodos tradicionales pueden no ser lo suficientemente eficaces debido a la falta de una solución analítica. MetaTrader 5 usa un GA binario y por lo tanto resulta doblemente interesante estudiar los principios de este maravilloso algoritmo.
2. Descripción del algoritmo
El algoritmo genético binario incluye los siguientes pasos:
- Inicialización de la población: se crea una población inicial formada por cromosomas con valores binarios aleatorios.
- Evaluación de la aptitud: se evalúa la aptitud de cada individuo (cromosoma) en la población hija.
- Cría: se seleccionan los padres para crear la descendencia mediante el método de la ruleta.
- Cruce: se rompen los cromosomas parentales en secciones y se crean nuevos cromosomas hijos con secciones de ambos progenitores.
- Inversión: se rompe el cromosoma hijo en un punto elegido al azar e se intercambian las regiones resultantes.
- Mutación: se cambian aleatoriamente bits en los genes de la descendencia con una probabilidad de mutación determinada.
- Evaluación de la aptitud de la descendencia: se evalúa la aptitud de cada nueva descendencia.
- Formación de una nueva población: se coloca la población descendiente al final de la población total y se clasifica según el valor de aptitud.
- Criterio de parada: se repite el proceso desde el paso 3 hasta alcanzar el criterio de parada.
Para garantizar que el BGA funcione solo con números positivos, simplificaremos las operaciones con cadenas binarias y aumentaremos la velocidad de los cálculos; así, utilizaremos la distancia entre "max" y "min" de los parámetros optimizados. Los valores positivos de las distancias que hayamos obtenido de este modo, los representaremos como código binario de Gray colocados secuencialmente en un array común de caracteres de cromosomas como se presenta en la figura 1. Debemos considerar que los puntos de ruptura del cromosoma al realizar el método de cruce se sitúan en lugares aleatorios del cromosoma y no necesariamente en los puntos de unión de los genes, los puntos de ruptura pueden estar dentro del espacio de los genes.
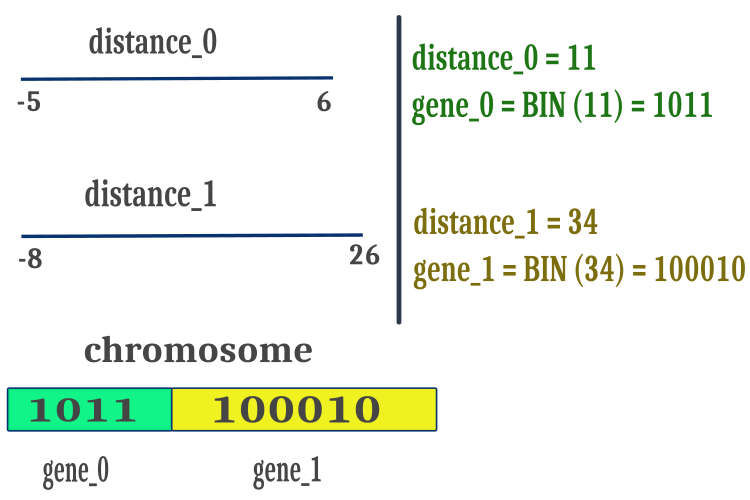
Figura 1. Colocación de las características de un individuo (parámetros optimizados) en un cromosoma.
Existe número considerable de parámetros externos en un algoritmo genético, por lo que resulta razonable considerarlos con más detalle. Los ajustes por defecto son casi exactamente los recomendados por muchos autores de publicaciones científicas. Los he probado y seleccionado para garantizar la máxima eficacia en la mayoría de los tipos de tareas. Sin embargo, desviarse de estos parámetros en cualquier dirección puede llevarnos a alcanzar una convergencia del 100% en pruebas con 10 o incluso 50 parámetros optimizados en funciones individuales, pero al mismo tiempo reducir significativamente el rendimiento en otras tareas. Por ello, los parámetros por defecto resultan óptimos para la mayoría de los casos prácticos.
- Population_P (tamaño de la población = 50): el parámetro define el número de descendientes en cada generación de la población. Este tamaño de población se usa en la mayoría de los algoritmos considerados en las pruebas y ofrece equilibrio entre la diversidad de soluciones y la tasa de convergencia.
- ParentPopulation_P (tamaño de la población parental = 50): el parámetro define el número de padres seleccionados para reproducirse y crear la siguiente generación. Disminuir el valor de este parámetro mejora la convergencia en funciones suaves (aumenta la precisión) y aumenta el valor en las discretas (aumenta la diversidad del material genético).
- CrossoverProbab_P (probabilidad de cruce = 1,0): el parámetro determina la probabilidad de realizar una operación de cruce entre dos padres seleccionados. Una probabilidad de cruce alta aumentará la capacidad combinatoria del algoritmo; un valor decreciente aumentará la tasa de convergencia pero aumentará la probabilidad de atascarse.
- CrossoverPoints_P (número de puntos de cruce = 3): el parámetro define el número de puntos en los que se producirá el cruce entre dos cromosomas parentales. El aumento de los puntos provoca una mezcla más intensa de los parámetros entre sí y, en el límite, se reduce a un comportamiento aleatorio del algoritmo.
- MutationProbab_P (probabilidad de mutación = 0,001): el parámetro determina la probabilidad de mutación de cada bit en el genotipo del cromosoma. La mutación permite la introducción de cambios aleatorios en el material genético y añade nuevas soluciones a la población. Una probabilidad baja aumenta la tasa de convergencia del algoritmo pero reducirá la diversidad, mientras que una probabilidad demasiado alta provoca la pérdida de información útil.
- InversionProbab_P (probabilidad de inversión = 0,7): el parámetro define la probabilidad de realizar una operación de inversión en un cromosoma. Una alta probabilidad de inversión aumenta la diversidad del material genético, pero una probabilidad demasiado alta provoca un comportamiento aleatorio del algoritmo.
- DoubleDigitsInChromo_P (número de decimales en el gen): este parámetro determina el número de decimales de un número real (parámetro optimizado) representado en forma binaria en el cromosoma. El aumento del valor provoca una mayor complejidad de los cálculos y el aumento de tiempo de optimización (sin utilizar la forma binaria directamente en la solución del problema no tiene sentido establecer más de 16, en la salida al convertir a double se perderán).
Vamos a ver ahora el código.
Al inicializar los agentes, debemos determinar el número de bits de la representación binaria del parámetro que se va a optimizar. Supongamos que necesitamos analizar cinco decimales, lo cual se corresponde con una determinada longitud del código de Gray. Sin embargo, en este código puede codificarse un número superior al exigido. Por ello, necesitamos determinar el número real máximo que se puede codificar en forma binaria. A continuación, podemos escalar el número codificado al rango deseado del parámetro optimizado en la salida. Para la parte fraccionaria de un número real usamos el número de dígitos especificado (especificado en los parámetros), mientras que para la parte entera utilizamos tantos como sean necesarios en forma binaria.
Por ejemplo, si el parámetro de entrada de la función "digitsInGrayCode" es 3, la función retornará el valor máximo del tipo "ulong" utilizando el código de Gray para 3 bits, es decir, 7 (111 en binario).
Para lograr este objetivo de determinar el número máximo posible que puede codificarse usando un número determinado de bits para las partes fraccionaria y entera de un número real, utilizaremos la función "GetMaxDecimalFromGray".
//—————————————————————————————————————————————————————————————————————————————— //Calculation of the maximum possible ulong number using the Gray code for a given number of bits ulong GetMaxDecimalFromGray (int digitsInGrayCode) { ulong maxValue = 1; for (int i = 1; i < digitsInGrayCode; i++) { maxValue <<= 1; maxValue |= 1; } return maxValue; } //——————————————————————————————————————————————————————————————————————————————
Para representar cada gen en el cromosoma (posición del gen en el cromosoma) usaremos la estructura "S_BinaryGene", que contiene los campos y métodos necesarios para trabajar con genes en formato binario:
- "gen": array de caracteres que representa un gen.
- "integPart": array de caracteres de la parte entera del gen.
- "fractPart": array de caracteres para la parte fraccionaria del gen.
- "integGrayDigits": número de dígitos del código Gray para la parte entera del gen.
- "fractGrayDigits": número de dígitos del código Gray para la parte fraccionaria del gen.
- "length": longitud total del gen.
- "minDoubleRange": valor mínimo del rango de números reales.
- "maxDoubleRange": valor máximo del rango de números reales.
- "maxDoubleNumber": número real máximo que se puede representar utilizando el gen.
- "doubleFract": valor para convertir la parte fraccionaria del gen en un número real.
Métodos de estructura:
- "Init": inicializa los campos de la estructura. Admite los valores mínimo y máximo de un rango de números reales, así como el número de dígitos de la parte fraccionaria del gen. Dentro del método, se calculan los valores para el número real máximo que codifica partes del gen utilizando el código de Gray.
- "ToDouble": convierte un gen en un número real. Acepta un array de caracteres del código de Gray "chromo" (cromosoma) y el índice de inicio del gen "indChr". El método lee una sección del cromosoma y convierte el gen leído en un valor decimal y después lo escala a un rango dado de números reales.
- "Scale": escala el valor de entrada "In" del rango "InMIN" e "InMAX" al rango de salida "OutMIN" y "OutMAX". Se trata de un método auxiliar que se usa en "ToDouble".
//—————————————————————————————————————————————————————————————————————————————— struct S_BinaryGene { char gene []; char integPart []; char fractPart []; uint integGrayDigits; uint fractGrayDigits; uint length; double minDoubleRange; double maxDoubleRange; double maxDoubleNumber; double doubleFract; //---------------------------------------------------------------------------- void Init (double min, double max, int doubleDigitsInChromo) { doubleFract = pow (0.1, doubleDigitsInChromo); minDoubleRange = min; maxDoubleRange = max - min; ulong decInfr = 0; for (int i = 0; i < doubleDigitsInChromo; i++) { decInfr += 9 * (ulong)pow (10, i); } //---------------------------------------- DecimalToGray (decInfr, fractPart); ulong maxDecInFr = GetMaxDecimalFromGray (ArraySize (fractPart)); double maxDoubFr = maxDecInFr * doubleFract; //---------------------------------------- DecimalToGray ((ulong)maxDoubleRange, integPart); ulong maxDecInInteg = GetMaxDecimalFromGray (ArraySize (integPart)); double maxDoubInteg = (double)maxDecInInteg + maxDoubFr; maxDoubleNumber = maxDoubInteg; ArrayResize (gene, 0, 1000); integGrayDigits = ArraySize (integPart); fractGrayDigits = ArraySize (fractPart); length = integGrayDigits + fractGrayDigits; ArrayCopy (gene, integPart, 0, 0, WHOLE_ARRAY); ArrayCopy (gene, fractPart, ArraySize (gene), 0, WHOLE_ARRAY); } //---------------------------------------------------------------------------- double ToDouble (const char &chromo [], const int indChr) { double d; if(integGrayDigits > 0)d = (double)GrayToDecimal(chromo, indChr, indChr + integGrayDigits - 1); else d = 0.0; d +=(double)GrayToDecimal(chromo, indChr + integGrayDigits, indChr + integGrayDigits + fractGrayDigits - 1) * doubleFract; return minDoubleRange + Scale(d, 0.0, maxDoubleNumber, 0.0, maxDoubleRange); } //---------------------------------------------------------------------------- double Scale (double In, double InMIN, double InMAX, double OutMIN, double OutMAX) { if (OutMIN == OutMAX) return (OutMIN); if (InMIN == InMAX) return (double((OutMIN + OutMAX) / 2.0)); else { if (In < InMIN) return OutMIN; if (In > InMAX) return OutMAX; return (((In - InMIN) * (OutMAX - OutMIN) / (InMAX - InMIN)) + OutMIN); } } }; //——————————————————————————————————————————————————————————————————————————————
Para describir un agente, necesitamos la estructura "S_Agent", que representa al agente y contiene los siguientes datos además del cromosoma:
- "c": array de valores de coordenadas del agente en forma de número real.
- "f": valor de aptitud del agente.
- "genes": array de estructuras "S_BinaryGene" que describen la posición de cada gen en el cromosoma y las reglas para convertir el código binario en un número real.
- "chromosome": array de caracteres del cromosoma del agente.
- "calculated": valor booleano que indica si se han calculado los valores del agente (el campo está presente pero no interviene en el código).
Métodos de estructura:
- "Init": inicializa los campos de la estructura. Acepta el número de coordenadas "coords", los arrays "min" y "max" con los valores mínimo y máximo de cada parámetro optimizado, y "doubleDigitsInChromo", el número de dígitos de números reales en la parte fraccionaria del gen. El método crea e inicializa los genes para cada coordenada, y establece el valor inicial de aptitud y la bandera "calculado".
- "ExtractGenes": extrae los valores de los genes del cromosoma y los almacena en el array "c", utiliza el método "ToDouble" de la estructura "S_BinaryGene" para convertir los genes del cromosoma en números reales.
//—————————————————————————————————————————————————————————————————————————————— struct S_Agent { void Init (const int coords, const double &min [], const double &max [], int doubleDigitsInChromo) { ArrayResize(c, coords); f = -DBL_MAX; ArrayResize(genes, coords); ArrayResize(chromosome, 0, 1000); for(int i = 0; i < coords; i++) { genes [i].Init(min [i], max [i], doubleDigitsInChromo); ArrayCopy(chromosome, genes [i].gene, ArraySize(chromosome), 0, WHOLE_ARRAY); } calculated = false; } void ExtractGenes () { uint pos = 0; for (int i = 0; i < ArraySize (genes); i++) { c [i] = genes [i].ToDouble (chromosome, pos); pos += genes [i].length; } } double c []; //coordinates double f; //fitness S_BinaryGene genes []; char chromosome []; bool calculated; }; //——————————————————————————————————————————————————————————————————————————————
A continuación, el código presenta la definición de la estructura "S_Roulette", que representa un segmento de ruleta.
Campos de la estructura:
- "start": valor del punto inicial del segmento de la ruleta.
- "end": valor del punto final del segmento de la ruleta.
//—————————————————————————————————————————————————————————————————————————————— struct S_Roulette { double start; double end; }; //——————————————————————————————————————————————————————————————————————————————
Declaramos la clase "C_AO_BGA" que implementa el algoritmo genético:
- "cB": array de los mejores valores de coordenadas.
- "fB": valor de aptitud de las mejores coordenadas.
- "a": array de estructuras "S_Agent" que representan agentes.
- "rangeMax": array de los valores máximos del rango de búsqueda.
- "rangeMin": array de los valores mínimos del rango de búsqueda.
- "rangeStep": array de valores que representa el paso de búsqueda.
Métodos de clase:
- "Init": inicializa los campos de la clase. Admite los parámetros: número de coordenadas "coordsP", tamaño de la población "popSizeP", tamaño de la población padre "parentPopSizeP", probabilidad de cruce "crossoverProbabP", número de puntos de cruce "crossoverPointsP", probabilidad de mutación "mutationProbabP", probabilidad de inversión "inversionProbabP", número de decimales en el gen "doubleDigitsInChromoP". El método inicializa las variables internas y los arrays necesarios para el funcionamiento del algoritmo genético.
- "Moving": método principal del algoritmo genético, realiza operaciones poblacionales como el cruce, la mutación, la inversión y la estimación de la aptitud.
- "Revision": el método realiza una revisión de la población, clasificando los agentes y seleccionando los mejores.
Los campos privados y los métodos de clase se usan para la implementación interna del algoritmo genético, incluyendo operaciones de ruleta, escalado de valores, clasificación y otras operaciones.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BGA { //---------------------------------------------------------------------------- public: double cB []; //best coordinates public: double fB; //FF of the best coordinates public: S_Agent a []; //agent public: double rangeMax []; //maximum search range public: double rangeMin []; //manimum search range public: double rangeStep []; //step search public: void Init (const int coordsP, //coordinates number const int popSizeP, //population size const int parentPopSizeP, //parent population size const double crossoverProbabP, //crossover probability const int crossoverPointsP, //crossover points const double mutationProbabP, //mutation probability const double inversionProbabP, //inversion probability const int doubleDigitsInChromoP);//number of decimal places in the gene public: void Moving (); public: void Revision (); //---------------------------------------------------------------------------- private: int coords; //coordinates number private: int popSize; //population size private: int parentPopSize; //parent population size private: double crossoverProbab; //crossover probability private: int crossoverPoints; //crossover points private: double mutationProbab; //mutation probability private: double inversionProbab; //inversion probability private: int doubleDigitsInChromo; //number of decimal places in the gene private: bool revision; private: S_Agent parents []; //parents private: int ind []; //temporary array for sorting the population private: double val []; //temporary array for sorting the population private: S_Agent pTemp []; //temporary array for sorting the population private: char tempChrome []; //temporary chromosome for inversion surgery private: uint lengthChrome; //length of the chromosome (the length of the string of characters according to the Gray code) private: int pCount; //indices of chromosome break points private: uint poRND []; //temporal indices of chromosome break points private: uint points []; //final indices of chromosome break points private: S_Roulette roulette []; //roulette private: void PreCalcRoulette (); private: int SpinRoulette (); private: double SeInDiSp (double In, double InMin, double InMax, double Step); private: double RNDfromCI (double min, double max); private: void Sorting (S_Agent &p [], int size); private: double Scale (double In, double InMIN, double InMAX, double OutMIN, double OutMAX); }; //——————————————————————————————————————————————————————————————————————————————
El siguiente código representa la implementación del método "Init" de la clase "C_AO_BGA". Este método inicializa los campos de la clase y los arrays necesarios para que funcione el algoritmo genético.
Parámetros de entrada del método:
- "coordsP": número de coordenadas.
- "popSizeP": tamaño de la población.
- "parentPopSizeP": tamaño de la población padre.
- "crossoverProbabP": probabilidad de cruce.
- "crossoverPointsP": número de puntos de cruce.
- "mutationProbabP": probabilidad de mutación.
- "inversionProbabP": probabilidad de inversión.
- "doubleDigitsInChromoP": número de decimales del gen.
El método "Init" se usa para inicializar los campos y los arrays de la clase antes de ejecutar el algoritmo genético. Establece los valores de los campos de clase, comprueba y corrige los valores de algunos parámetros y redimensiona los arrays asignándoles memoria.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BGA::Init (const int coordsP, //coordinates number const int popSizeP, //population size const int parentPopSizeP, //parent population size const double crossoverProbabP, //crossover probability const int crossoverPointsP, //crossover points const double mutationProbabP, //mutation probability const double inversionProbabP, //inversion probability const int doubleDigitsInChromoP) //number of decimal places in the gene { MathSrand ((int)GetMicrosecondCount ()); // reset of the generator fB = -DBL_MAX; revision = false; coords = coordsP; popSize = popSizeP; parentPopSize = parentPopSizeP; crossoverProbab = crossoverProbabP; crossoverPoints = crossoverPointsP; pCount = crossoverPointsP; mutationProbab = mutationProbabP; inversionProbab = inversionProbabP; doubleDigitsInChromo = doubleDigitsInChromoP; if (crossoverPoints < 1) crossoverPoints = 1; if (pCount < 1) pCount = 1; ArrayResize (poRND, pCount); ArrayResize (points, pCount + 2); ArrayResize (ind, parentPopSize + popSize); ArrayResize (val, parentPopSize + popSize); ArrayResize (pTemp, parentPopSize + popSize); ArrayResize (a, popSize); ArrayResize (parents, parentPopSize + popSize); ArrayResize (roulette, parentPopSize); ArrayResize (rangeMax, coords); ArrayResize (rangeMin, coords); ArrayResize (rangeStep, coords); ArrayResize (cB, coords); } //——————————————————————————————————————————————————————————————————————————————
Todas las operaciones básicas de trabajo con el material genético de los agentes se realizan usando el método "Moving" de la clase "C_AO_BGA". El método Moving ejecuta un paso del algoritmo genético que incluye el muestreo parental, el cruce, la inversión y la mutación de cromosomas, y la aplicación de operaciones a los genes y las coordenadas de los individuos.
El método se divide de forma lógica en varias partes:
1. "if (!revision)" - si "revision" es "false", se realizará la inicialización de los individuos de la población:
- Primero se llama al método "Init" para inicializar el individuo con los parámetros dados.
- Luego se genera un cromosoma aleatorio rellenando cada gen con un valor aleatorio de 0 o 1.
- Después se llama al método "ExtractGenes" para extraer genes del cromosoma.
- Las coordenadas del individuo "c" se llevan al rango usando la función "SeInDiSp".
- El valor de aptitud de cada individuo "f" se establece en "-DBL_MAX".
- "lengthChrome = ArraySize (a [0].chromosome)" - se define la longitud del cromosoma de un individuo (todos los individuos tienen la misma longitud).
- "ArrayResize (tempChrome, lengthChrome)" - cambia el tamaño del array temporal "tempChrome" a "lengthChrome".
2. Para cada individuo de la población:
- Se realiza un cálculo previo de la ruleta de selección parental mediante el método "PreCalcRoulette".
- La selección parental se realiza usando el método "SpinRoulette".
- Se copia el cromosoma del individuo parental seleccionado en el cromosoma del individuo actual.
- Se efectúa una operación de cruce con probabilidad "crossoverProbab".
- Se selecciona un segundo individuo parental.
- Se identifican los puntos de rotura cromosómica.
- Se realiza un cruce entre los cromosomas de los individuos parentales.
- Se realiza una operación de inversión con probabilidad "inversionProbab".
- Se determina un punto de rotura cromosómico aleatorio.
- Las partes del cromosoma cambian de lugar.
- Se efectúa una operación de mutación para cada gen del cromosoma con una probabilidad de "mutationProbab".
- Después se llama al método "ExtractGenes" para extraer genes del cromosoma.
- Las coordenadas del individuo "c" se llevan al rango usando la función "SeInDiSp".
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BGA::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { a [i].Init (coords, rangeMin, rangeMax, doubleDigitsInChromo); int r = 0; for (int len = 0; len < ArraySize (a [i].chromosome); len++) { r = MathRand (); //[0,32767] if (r > 16384) a [i].chromosome [len] = 1; else a [i].chromosome [len] = 0; } a [i].ExtractGenes (); for (int c = 0; c < coords; c++) a [i].c [c] = SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].f = -DBL_MAX; a [i].calculated = true; } lengthChrome = ArraySize (a [0].chromosome); ArrayResize (tempChrome, lengthChrome); for (int i = 0; i < parentPopSize + popSize; i++) { parents [i].Init (coords, rangeMin, rangeMax, doubleDigitsInChromo); parents [i].f = -DBL_MAX; } revision = true; return; } //---------------------------------------------------------------------------- int pos = 0; double r = 0; uint p1 = 0; uint p2 = 0; uint p3 = 0; uint temp = 0; for (int i = 0; i < popSize; i++) { PreCalcRoulette (); //selection, select and copy the parent to the child------------------------ pos = SpinRoulette (); ArrayCopy (a [i].chromosome, parents [pos].chromosome, 0, 0, WHOLE_ARRAY); //crossover----------------------------------------------------------------- r = RNDfromCI (0.0, 1.0); if (r < crossoverProbab) { //choose a second parent to breed with------------------------------------ pos = SpinRoulette (); //determination of chromosome break points-------------------------------- for (int p = 0; p < pCount; p++) { poRND [p] = (int)RNDfromCI (0.0, lengthChrome); if (poRND [p] >= lengthChrome) poRND [p] = lengthChrome - 1; } ArraySort (poRND); ArrayCopy (points, poRND, 1, 0, WHOLE_ARRAY); points [0] = 0; points [pCount + 1] = lengthChrome - 1; r = RNDfromCI (0.0, 1.0); int startPoint = r > 0.5 ? 0 : 1; for (int p = startPoint; p < pCount + 2; p += 2) { if (p < pCount + 1) { for (uint len = points [p]; len < points [p + 1]; len++) a [i].chromosome [len] = parents [pos].chromosome [len]; } } } //perform an inversion------------------------------------------------------ //(break the chromosome, swap the received parts, connect them together) r = RNDfromCI (0.0, 1.0); if (r < inversionProbab) { p1 = (int)RNDfromCI (0.0, lengthChrome); if (p1 >= lengthChrome) p1 = lengthChrome - 1; //copying the second part to the beginning of the temporary array for (uint len = p1; len < lengthChrome; len++) tempChrome [len - p1] = a [i].chromosome [len]; //copying the first part to the end of the temporary array for (uint len = 0; len < p1; len++) tempChrome [lengthChrome - p1 + len] = a [i].chromosome [len]; //copying a temporary array back for (uint len = 0; len < lengthChrome; len++) a [i].chromosome [len] = tempChrome [len]; } //выполнить мутацию--------------------------------------------------------- for (uint len = 0; len < lengthChrome; len++) { r = RNDfromCI (0.0, 1.0); if (r < mutationProbab) a [i].chromosome [len] = a [i].chromosome [len] == 1 ? 0 : 1; } a [i].ExtractGenes (); for (int c = 0; c < coords; c++) a [i].c [c] = SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].calculated = true; } } //——————————————————————————————————————————————————————————————————————————————
El método Revision realiza una revisión de la población, clasifica los individuos según el valor de su función de aptitud y actualiza la aptitud de la mejor solución "fB" y las coordenadas de la mejor solución "cB". Antes de clasificar la población, la población hija se copiará al final de la población total.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BGA::Revision () { //---------------------------------------------------------------------------- for (int i = parentPopSize; i < parentPopSize + popSize; i++) { parents [i] = a [i - parentPopSize]; } Sorting (parents, parentPopSize + popSize); if (parents [0].f > fB) { fB = parents [0].f; ArrayCopy (cB, parents [0].c, 0, 0, WHOLE_ARRAY); } } //——————————————————————————————————————————————————————————————————————————————
El código de pre-marcado de la ruleta representa el método "PreCalcRoulette". Este método calcula previamente los valores de rango para la ruleta de selección de individuos usando como base su función de aptitud.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BGA::PreCalcRoulette () { roulette [0].start = parents [0].f; roulette [0].end = roulette [0].start + (parents [0].f - parents [parentPopSize - 1].f); for (int s = 1; s < parentPopSize; s++) { if (s != parentPopSize - 1) { roulette [s].start = roulette [s - 1].end; roulette [s].end = roulette [s].start + (parents [s].f - parents [parentPopSize - 1].f); } else { roulette [s].start = roulette [s - 1].end; roulette [s].end = roulette [s].start + (parents [s - 1].f - parents [s].f) * 0.1; } } } //——————————————————————————————————————————————————————————————————————————————
El método "SpinRoulette" se usa para imponer la probabilidad de selección de la posición del padre.
//—————————————————————————————————————————————————————————————————————————————— int C_AO_BGA::SpinRoulette () { double r = RNDfromCI (roulette [0].start, roulette [parentPopSize - 1].end); for (int s = 0; s < parentPopSize; s++) { if (roulette [s].start <= r && r < roulette [s].end) return s; } return 0; } //——————————————————————————————————————————————————————————————————————————————
3. Resultados de las pruebas
Impresión del funcionamiento del algoritmo genético binario BGA en el banco de pruebas:
C_AO_BGA:50:50:1.0:3:0.001:0.7:3
=============================
5 Hilly's; Func runs: 10000; result: 0.9999191151339382
25 Hilly's; Func runs: 10000; result: 0.994841435673127
500 Hilly's; Func runs: 10000; result: 0.5048331764136147
=============================
5 Forest's; Func runs: 10000; result: 1.0
25 Forest's; Func runs: 10000; result: 0.9997457419655973
500 Forest's; Func runs: 10000; result: 0.32054251149158375
=============================
5 Megacity's; Func runs: 10000; result: 0.9066666666666668
25 Megacity's; Func runs: 10000; result: 0.9640000000000001
500 Megacity's; Func runs: 10000; result: 0.23034999999999997
=============================
All score: 6.92090 (76.90%)
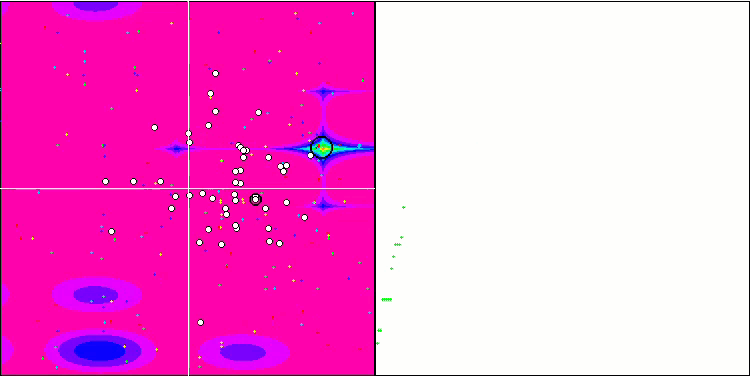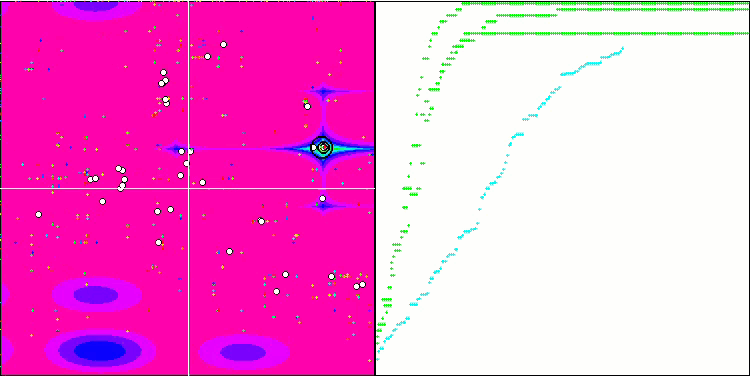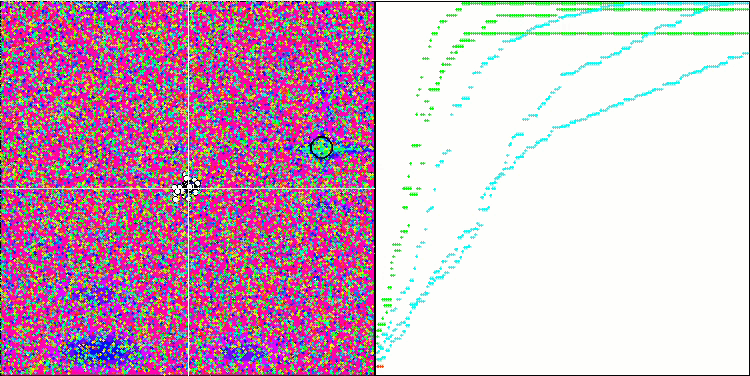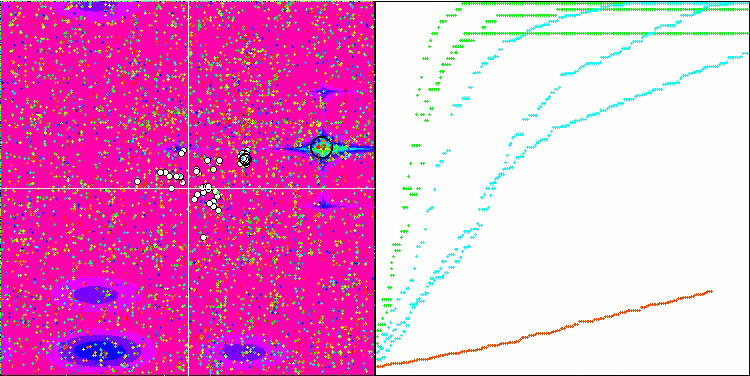
La visualización del proceso de optimización BGA demuestra claramente la elevada convergencia del algoritmo. Resulta interesante observar que, durante el proceso de optimización, una parte de la población se mantiene alejada del extremo global, lo cual indica la exploración de nuevas regiones desconocidas del espacio de búsqueda, al tiempo que se mantiene la diversidad de soluciones en la población. No obstante, el algoritmo se enfrenta a algunas dificultades al tratar con la función discreta "Megacity", que resulta problemática para la mayoría de los algoritmos. A pesar de existir cierta dispersión en los resultados al trabajar con esta compleja función, el BGA se comporta significativamente mejor que otros algoritmos.
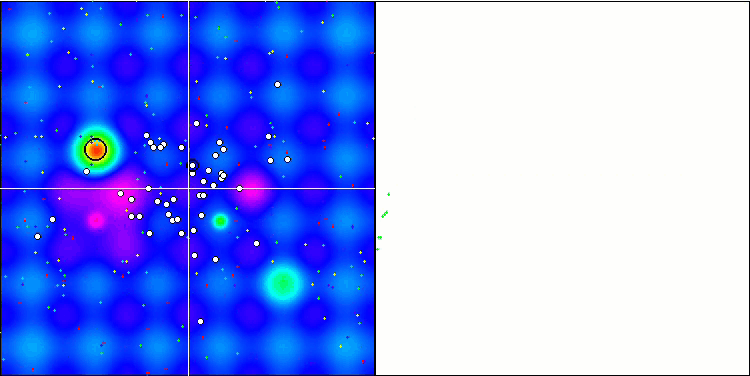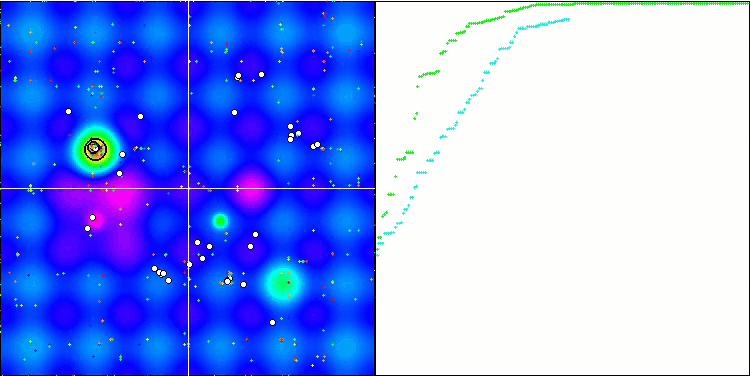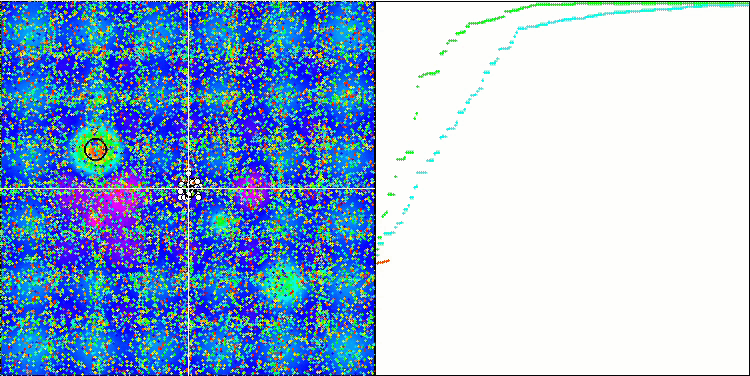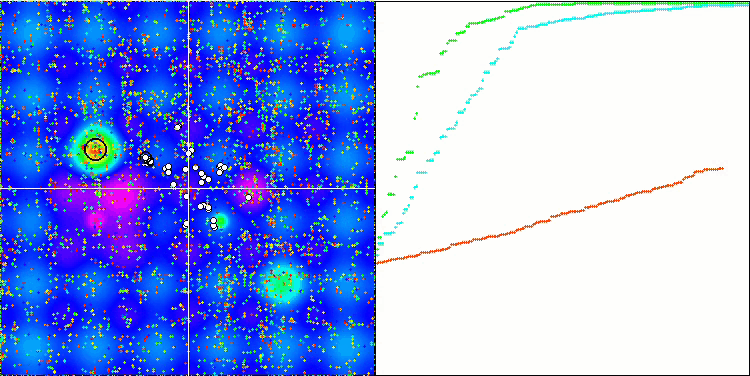
BGA en la función de prueba Hilly.
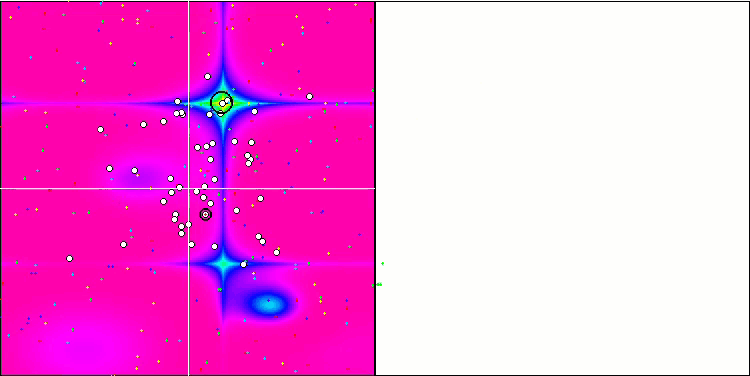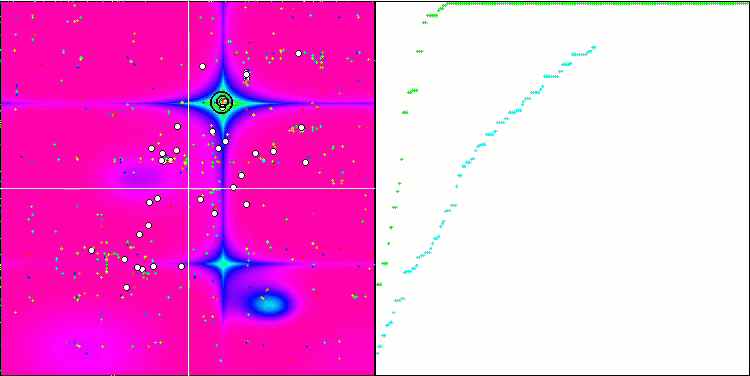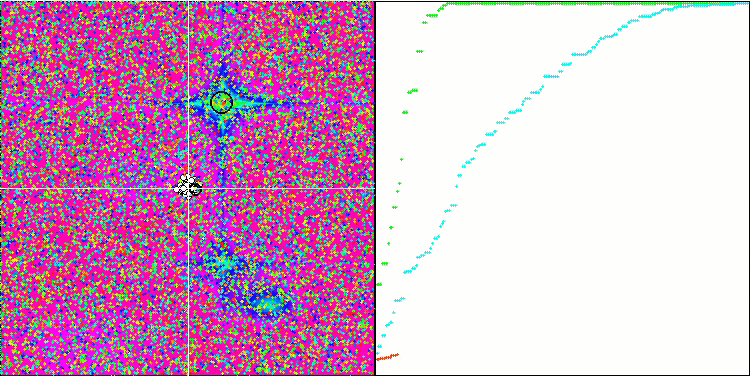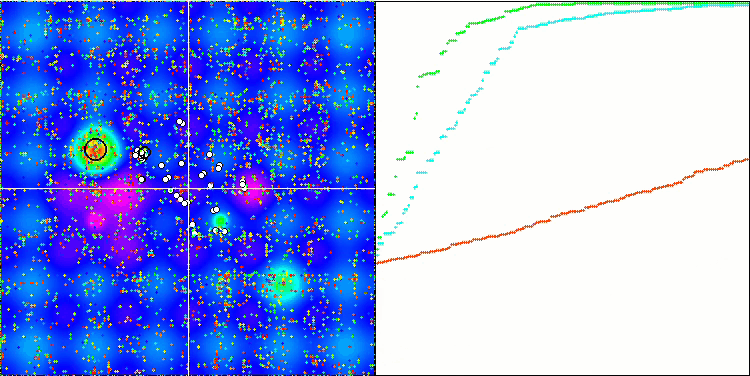
BGA en la función de prueba Forest.
BGA en la función de prueba Megacity.
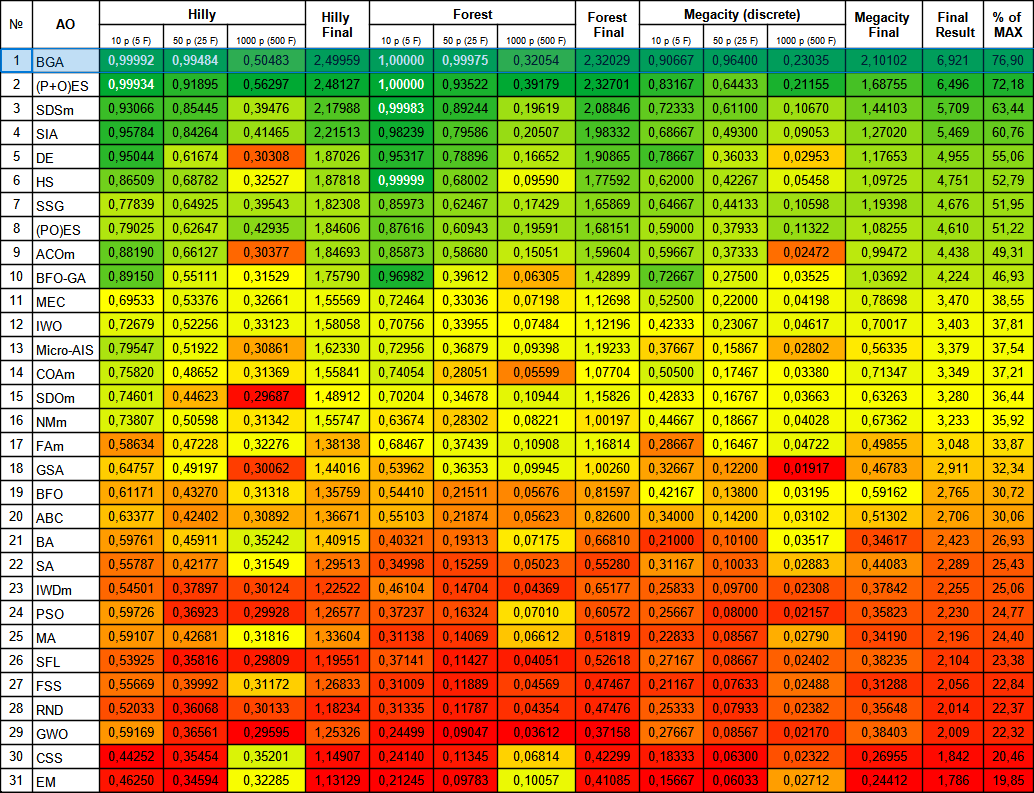
El BGA ha ocupado el primer puesto de la tabla de clasificación con los mejores resultados en la mayoría de las pruebas para las tres funciones de prueba. Asimismo, ha mostrado unos resultados especialmente impresionantes en la función Megacity discreta, superando a otros algoritmos.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Conclusiones
En este artículo hemos analizado la variante clásica de BGA, un caso especial de la clase general de algoritmos genéticos GA y todas las conclusiones son aplicables a esta. A pesar de que la idea de la representación binaria de las soluciones viene de lejos, el enfoque del código binario sigue resultando pertinente hoy en día, pues combina dimensiones espaciales independientes del problema de optimización en una sola entidad usando la codificación de la información en un único cromosoma, lo cual resulta difícil de aplicar utilizando la codificación convencional de características del mundo real, lo que hace que este algoritmo destaque entre otros algoritmos de optimización.
Aunque entiendo perfectamente el aparato matemático y la lógica de la estrategia BGA, me sigue fascinando lo que sucede con el cromosoma. Es como compararlo con un caleidoscopio mágico. Cuando giramos un caleidoscopio, toda una variedad de formas y colores se unen en patrones únicos para crear una imagen impresionante. Del mismo modo, un operador de cruce en un BGA corta aleatoriamente el cromosoma en múltiples partes, incluidas las regiones de los parámetros internos. Acto seguido, estas piezas se unen, como si se barajaran los fragmentos de un caleidoscopio. Este proceso permite combinar las mejores características de distintas soluciones y crear una nueva combinación, aún más óptima. Como en un caleidoscopio, los resultados del cruce de BGA pueden ser sorprendentes e inesperados, convirtiendo simples cromosomas en verdaderos "diamantes" de soluciones óptimas.
Estoy seguro de que la información de las dos partes de este artículo sobre los métodos y herramientas usados en los algoritmos genéticos le ayudará a ampliar sus conocimientos y alcanzar nuevas cotas en su trabajo e investigación. La naturaleza, la tecnología y la mente humana demuestran continuamente el poder de la evolución, y el BGA es uno de los muchos algoritmos asombrosos que nos ayudarán a alcanzar nuevas metas y logros.
El BGA resuelve eficazmente una gran variedad de problemas, ya hablemos de superficies lisas, problemas discretos o incluso problemas de alta dimensionalidad, incluso utilizando un enfoque estocástico, lo cual abre nuevas posibilidades allí donde las soluciones analíticas están limitadas.
 <
<
Figura 2. Gradación por colores de los algoritmos según sus respectivas pruebas. Los resultados superiores o iguales a 0,99 aparecen resaltados en blanco.
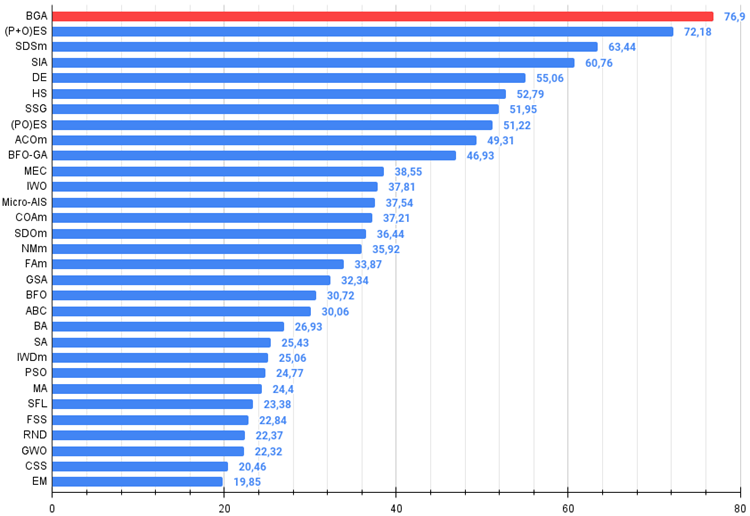
Figura 3. Histograma con los resultados de las pruebas de los algoritmos (en una escala de 0 a 100, cuanto mayor, mejor),
donde 100 es el máximo resultado teórico posible; hay un script para calcular la tabla de puntuación en el archivo).
Los pros y los contras del Algoritmo Genético Binario (BGA) se refieren únicamente a la aplicación presentada:
Ventajas:
- Alta eficiencia en una gran variedad de tareas.
- Resistencia al atascamiento.
- Buenos resultados en funciones discretas tanto suaves como complejas.
- Alta convergencia.
Desventajas:
- Gran número de parámetros externos.
- Aplicación bastante compleja del algoritmo.
- Alta complejidad computacional.
Adjuntamos al artículo un archivo con las versiones reales actualizadas de los códigos de los algoritmos descritos en los artículos anteriores. El autor de este artículo no se responsabiliza de la exactitud absoluta de la descripción de los algoritmos canónicos, muchos de ellos han sido modificados para mejorar las capacidades de búsqueda. Las conclusiones y juicios presentados en los artículos se basan en los resultados de los experimentos realizados.
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/14040
 Desarrollo de un sistema de repetición (Parte 46): Proyecto Chart Trade (V)
Desarrollo de un sistema de repetición (Parte 46): Proyecto Chart Trade (V)
 Desarrollamos de un asesor multidivisa (Parte 1): Funcionamiento conjunto de varias estrategias comerciales
Desarrollamos de un asesor multidivisa (Parte 1): Funcionamiento conjunto de varias estrategias comerciales
 Desarrollo de un sistema de repetición (Parte 47): Proyecto Chart Trade (VI)
Desarrollo de un sistema de repetición (Parte 47): Proyecto Chart Trade (VI)
 Desarrollo de un sistema de repetición (Parte 45): Proyecto Chart Trade (IV)
Desarrollo de un sistema de repetición (Parte 45): Proyecto Chart Trade (IV)
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso