Algoritmos de optimización de la población: Algoritmo híbrido de optimización de forrajeo bacteriano con algoritmo genético (Bacterial Foraging Optimization - Genetic Algorithm, BFO-GA)
Contenido:
1. Introducción
2. Descripción del algoritmo
3. Resultados de las pruebas
1. Introducción
El algoritmo de optimización híbrido BFO-GA combina dos algoritmos de optimización diferentes: el algoritmo de optimización de forrajeo bacteriano (BFO) y el algoritmo genético (GA). Este algoritmo híbrido se creó para mejorar la eficacia de la optimización y superar algunas de las deficiencias de cada uno de los algoritmos por separado.
El BFO es un algoritmo de optimización inspirado en el comportamiento de las bacterias cuando buscan comida, propuesto en 2002 por Rahul K. Kujur. El BFO modela el movimiento bacteriano usando tres mecanismos principales: transiciones, difusión y actualización de la posición. Cada bacteria del algoritmo representa una solución al problema de optimización, y el alimento corresponde a la solución óptima. Así, las bacterias se mueven por el espacio de búsqueda para encontrar el mejor alimento.
El algoritmo genético (AG) es un algoritmo de optimización inspirado en los principios de la selección natural y la genética, desarrollado por John Holland en los años setenta. El GA trabaja con una población de individuos que representan soluciones a un problema de optimización. Para crear nuevas generaciones, los individuos se someten a operaciones de cruce (combinación de información genética) y mutación (cambios aleatorios en la información genética). Tras varias generaciones, el GA se esfuerza por encontrar la mejor solución.
El algoritmo híbrido BFO-GA combina las ventajas de ambos algoritmos. El BFO tiene buena capacidad de búsqueda local y convergencia rápida, pero puede tener problemas en la búsqueda global. Por otro lado, el AG tiene una buena capacidad de búsqueda global, pero puede resultar lento en la convergencia y propenso a quedarse atascado en óptimos locales. El algoritmo híbrido BFO-GA intenta superar dichas limitaciones utilizando el BFO para la búsqueda global y el GA para refinar los óptimos locales.
En 2007, D. N. Kim, A. Abraham y Chou propusieron el algoritmo híbrido BFO-GA (Bacterial Foraging Optimisation - Genetic Algorithm). Este algoritmo combina los principios de optimización basados en el comportamiento de los enjambres de bacterias con operadores genéticos de selección, cruce y mutación.
El BFO-GA utiliza el algoritmo bacteriano como base, pero lo amplía con operadores genéticos de selección, cruce y mutación. Este algoritmo usa el enjambre bacteriano para encontrar la solución óptima.
El BFO-GA utiliza un algoritmo basado en el método de la ruleta como operador de selección. Este método selecciona las bacterias progenitoras con una probabilidad proporcional a su aptitud. Así, las bacterias mejor adaptadas tienen más probabilidades de ser seleccionadas para el cruce.
El cruce en el algoritmo BFO-GA se implementa usando el operador de cruce aritmético. Este combina la información genética de dos bacterias progenitoras, creando una nueva descendencia con nuevas combinaciones de genes.
La mutación en BFO-GA se realiza mediante un mutador de Michalewicz no uniforme. Este mutador altera la información genética de la bacteria mediante cambios aleatorios. La no uniformidad de la mutación implica que la probabilidad de mutación puede variar en función de diversos factores.
Los operadores modificadores anteriores se realizan una vez finalizados los procedimientos de quimiotaxis y reproducción del algoritmo bacteriano, antes de los procedimientos de liquidación y dispersión de este algoritmo. Es decir, después de que las bacterias hayan realizado un movimiento hacia una solución óptima y producido descendencia, se aplicarán operadores genéticos para diversificar y mejorar las soluciones.
2. Descripción del algoritmo
El algoritmo BFO-GA usa la modelización del comportamiento de los enjambres bacterianos para encontrar la mejor solución a un problema de optimización, simulando el movimiento de bacterias en el espacio de parámetros, donde cada bacteria representa una solución potencial al problema. Las bacterias se mueven en el espacio paramétrico realizando dos tipos básicos de movimientos: movimientos en la dirección del gradiente alimentario y movimientos en una dirección aleatoria.
El algoritmo BFO-GA sigue los siguientes pasos:
- Inicialización: Se crea una población inicial de bacterias, cada una de las cuales representa una solución potencial al problema. Cada bacteria tiene sus propios parámetros que pueden modificarse durante el proceso de optimización.
- Se evalúa el gradiente alimentario: Se calcular el valor de la función de aptitud para cada bacteria de la población y se comparan además los dos últimos valores; esta diferencia de valores determinará la calidad de la solución presentada por cada bacteria.
- Movimiento en la dirección del gradiente: Cada bacteria se mueve en la dirección del gradiente alimentario, lo cual le permite avanzar hacia soluciones más óptimas con un vector constante de cambio de posición.
- Movimiento en una dirección aleatoria: Algunas de las bacterias también realizan movimientos aleatorios para evitar quedarse atascadas en óptimos localizados. Este movimiento se basa en un cambio aleatorio de los parámetros de la bacteria si el movimiento anterior ha sido fallido.
- Operadores genéticos: Aplicamos los operadores genéticos (selección, cruzamiento y mutación) a una población de bacterias. Esto permite crear nuevas combinaciones de parámetros y mejorar la calidad de las soluciones.
- Repetimos los pasos 2-5: Repetimos los pasos del movimiento en la dirección del gradiente, del movimiento en una dirección aleatoria y aplicamos los operadores genéticos hasta que se alcance un criterio de parada, como alcanzar un número máximo de iteraciones o alcanzar un determinado valor de la función de aptitud.
En teoría, el algoritmo híbrido BFO-GA debería tener una serie de ventajas sobre el BFO que merece la pena mencionar:
- Exploración y explotación: El BFO-GA tiene la capacidad de explorar el espacio de parámetros, gracias al movimiento aleatorio de las bacterias, y también de realizar la explotación, debido al movimiento en la dirección del gradiente alimentario. Esto permite al algoritmo encontrar soluciones óptimas evitando los óptimos locales y convergiendo a uno global.
- Adaptabilidad: El BFO-GA tiene la capacidad de adaptarse a condiciones de optimización cambiantes. Durante el funcionamiento del algoritmo, los parámetros de la bacteria pueden cambiar, lo cual le permite adaptarse a nuevas condiciones y encontrar mejores soluciones.
- No es necesario ajustar los parámetros: Como cualquier algoritmo de optimización, el BFO-GA tiene una serie de parámetros que pueden ajustarse para obtener mejores resultados. Esto incluye los parámetros relacionados con el tamaño de la población bacteriana, los pasos del movimiento en la dirección del gradiente y el movimiento aleatorio, las probabilidades de los operadores genéticos y otros. La modificación de los parámetros no tiene ningún efecto significativo sobre el resultado.

Figura 1. Herencia de los "genes" parentales por parte de la bacteria hija.
Vamos a pasar ahora de los fundamentos teóricos a la aplicación práctica.
En primer lugar, hemos abandonado la selección de ruleta (el método utilizado en el algoritmo de la colonia artificial de abejas), experimentalmente hemos obtenido mejores resultados en el BFO-GA utilizando la selección aleatoria simple entre la población parental.
En segundo lugar, hemos decidido sustituir el mutador de Michalewicz no uniforme por una mutación de ley escalonada (algunas fuentes se refieren a ella como una especie de mutador escalonado), que es esencialmente la generación de un número aleatorio con una distribución escalonada mencionada en el artículo sobre el Cefalópodo Inteligente.
En tercer lugar, el operador de cruce aritmético ha sido sustituido por la simple herencia aleatoria de genes de diferentes individuos parentales elegidos al azar según una ley de distribución uniforme.
Para describir una bacteria, escribiremos la estructura S_Bacteria, que contiene los siguientes campos:
- c: array de coordenadas de la bacteria, representa las coordenadas actuales de la bacteria en el espacio de parámetros, el tamaño del array "c" dependerá del número de variables del problema de optimización.
- cLast: array de coordenadas anteriores de la bacteria, utilizado para almacenar la posición anterior de la bacteria en el espacio de parámetros a partir del cual se calcula la nueva posición.
- v: array de vectores bacterianos, representa la dirección actual del movimiento bacteriano en el espacio de parámetros, el tamaño de la array "v" dependerá del número de variables del problema de optimización.
- f: valor actual de salud (aptitud) de la bacteria, define la calidad de la posición actual de la bacteria en el espacio de parámetros, cuanto mayor sea el valor de "f", mejor.
- fLast: valor anterior de salud bacteriana, utilizado para rastrear la calidad anterior de la posición bacteriana y usado en la determinación del gradiente alimentario.
- lifeCNT: contador de vida de la bacteria, lleva la cuenta del número de iteraciones que la bacteria ha pasado en la posición actual; se usa para limitar el número de pasos que la bacteria se mueve en una dirección.
//—————————————————————————————————————————————————————————————————————————————— struct S_Bacteria { double c []; //coordinates double cLast []; //coordinates double v []; //vector double f; //current health double fLast; //previous health double lifeCNT; //life counter }; //——————————————————————————————————————————————————————————————————————————————
Ahora declararemos la clase C_AO_BFO_GA, que implementa el algoritmo BFO-GA (Bacterial Foraging Optimisation - Genetic Algorithm). Vamos a desglosar cada campo y método de la clase:
Campos de clase:
- b: array de bacterias, cada elemento del array será un objeto de tipo "S_Bacteria" que representará una bacteria en el algoritmo.
- rangeMax: array de los valores máximos del rango de búsqueda para cada variable del problema de optimización.
- rangeMin: array de los valores mínimos del rango de búsqueda para cada variable.
- rangeStep: array de pasos de búsqueda para cada variable.
- cB: array de las mejores coordenadas encontradas por el algoritmo.
- fB: valor de la función de aptitud para las mejores coordenadas.
Métodos de clase:
- Init: Inicializa los parámetros del algoritmo BFO-GA, como el número de parámetros a optimizar "paramsP"; el tamaño de la población "populationSizeP"; el parámetro "lambda", la longitud del movimiento bacteriano; la probabilidad de reproducción "reproductionP"; el contador de vidas "lifeCounterP", cuando se completa el número de vidas, las bacterias realizan una voltereta; y la potencia de mutación "powerMutP".
- Swimming: realiza el movimiento de las bacterias en el espacio de los parámetros.
- Evaluation: realiza la revisión de la población y actualiza la solución global.
Métodos privados de clase:
- NewVector: genera un nuevo vector para el parámetro "paramInd" especificado de la bacteria.
- PowerDistribution: aplica una distribución escalonada de potencia al valor de entrada "In" con los parámetros establecidos "outMin", "outMax", "power".
- Sorting: clasifica las bacterias de una población según sus valores de función de aptitud en orden descendente.
- SeInDiSp: normaliza el valor de entrada "In" en el rango [InMin, InMax] con el paso "Step".
- RNDfromCI: genera un número aleatorio en el intervalo especificado [min, max].
- Scale: escala el valor de entrada "In" del intervalo [InMIN, InMAX] al intervalo [OutMIN, OutMAX].
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BFO_GA { //---------------------------------------------------------------------------- public: S_Bacteria b []; //bacteria public: double rangeMax []; //maximum search range public: double rangeMin []; //manimum search range public: double rangeStep []; //step search public: double cB []; //best coordinates public: double fB; //FF of the best coordinates public: void Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP, //life counter const double powerMutP); //mutation power public: void Swimming (); public: void Evaluation (); //---------------------------------------------------------------------------- private: double NewVector (int paramInd); private: S_Bacteria bT []; //bacteria private: double v []; private: int ind []; private: double val []; private: int populationSize; //population size private: int parameters; //number of optimized parameters private: double lambda; //lambda private: double reproduction; //probability of reproduction private: int lifeCounter; //life counter private: double powerMut; //mutation power private: bool evaluation; private: double PowerDistribution (const double In, const double outMin, const double outMax, const double power); private: void Sorting (); private: double SeInDiSp (double In, double InMin, double InMax, double Step); private: double RNDfromCI (double min, double max); private: double Scale (double In, double InMIN, double InMAX, double OutMIN, double OutMAX, bool revers); }; //——————————————————————————————————————————————————————————————————————————————
El método "Init" se utiliza para inicializar la clase, que inicializará los parámetros del algoritmo BFO-GA.
Parámetros de entrada del método:
- paramsP: número de parámetros a optimizar.
- populationSizeP: tamaño de la población.
- lambdaP: parámetro lambda que define el rango de movimiento de la bacteria en cada paso.
- reproductionP: probabilidad de reproducción.
- lifeCounterP: contador de vida.
- powerMutP: poder de mutación.
Dentro del método los campos de la clase C_AO_BFO_GA se inicializarán con los valores iniciales y se establecerán los tamaños de los arrays.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO_GA::Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP, //life counter const double powerMutP) //mutation power { fB = -DBL_MAX; evaluation = false; parameters = paramsP; populationSize = populationSizeP; lambda = lambdaP; reproduction = reproductionP; lifeCounter = lifeCounterP; powerMut = powerMutP; ArrayResize (rangeMax, parameters); ArrayResize (rangeMin, parameters); ArrayResize (rangeStep, parameters); ArrayResize (v, parameters); ArrayResize (ind, populationSize); ArrayResize (val, populationSize); ArrayResize (b, populationSize); ArrayResize (bT, populationSize); for (int i = 0; i < populationSize; i++) { ArrayResize (b [i].c, parameters); ArrayResize (b [i].cLast, parameters); ArrayResize (b [i].v, parameters); b [i].f = -DBL_MAX; b [i].fLast = -DBL_MAX; ArrayResize (bT [i].c, parameters); ArrayResize (bT [i].cLast, parameters); ArrayResize (bT [i].v, parameters); } ArrayResize (cB, parameters); } //——————————————————————————————————————————————————————————————————————————————
Para realizar la quimiotaxis bacteriana, la replicación, la selección, la herencia de los rasgos y la mutación, escribiremos el método Swimming:
void Swimming ()
{
} En la primera iteración, la bandera de "evaluación" será igual a "false"; distribuiremos las bacterias por el espacio de búsqueda aleatoriamente con una distribución uniforme. En esta fase, la salud actual y la salud anterior se desconocen, por lo que asignaremos "-DBL_MAX" a las variables correspondientes, además de añadir un vector de movimiento aleatorio a la bacteria recién creada.
Asimismo, comprobaremos si las coordenadas obtenidas de la posición de la bacteria se encuentran fuera de los límites y aplicaremos un cambio de paso en los parámetros optimizados. El vector de movimiento de cada bacteria les permite nadar de forma uniforme y progresiva.
if (!evaluation) { fB = -DBL_MAX; for (int s = 0; s < populationSize; s++) { for (int k = 0; k < parameters; k++) { b [s].c [k] = RNDfromCI (rangeMin [k], rangeMax [k]); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); v [k] = rangeMax [k] - rangeMin [k]; b [s].v [k] = NewVector (k); b [s].f = -DBL_MAX; b [s].fLast = -DBL_MAX; b [s].lifeCNT = 0; } } evaluation = true; return; }
En la segunda iteración y en las siguientes, primero se comprobará el cumplimiento de la probabilidad de reproducción bacteriana (replicación). Si se cumple la probabilidad de reproducción, ocurrirá lo siguiente:
- "bacterium original": la mejor mitad de la población bacteriana clasificada continuará su movimiento en la misma dirección, para ello añadiremos el vector de movimiento individual de cada bacteria a las coordenadas de la posición anterior.
- "bacterium clone": la otra mitad de la población estará llena de bacterias hijas, cada una de las cuales se obtendrá de los "genes" (coordenadas) de diferentes bacterias progenitoras, heredando así rasgos y cumpliendo la capacidad combinatoria del algoritmo; figura 1 anterior.
- Las bacterias clonadas que heredan genes de diferentes progenitores heredan, además, un componente del vector de movimiento del progenitor, y el gen heredado sufre una mutación según la ley de distribución escalonada.
Así, las bacterias clonadas heredan los rasgos de diferentes progenitores. En este caso, los genes se alterarán con alta probabilidad en sus proximidades y con una probabilidad distinta de cero lejos de los valores de los genes de sus progenitores. Esto permitirá combinar los rasgos exitosos de los progenitores, ya que los genes solo pueden transmitir a los herederos los mejores miembros de la población. Además, los componentes heredados del vector de movimiento hacen que las bacterias hijas empiecen a moverse en la siguiente iteración según los mejores componentes de los vectores de sus progenitores.
De forma ideal, esto debería parecerse al movimiento de un banco de peces, pero esta analogía no siempre se cumple debido a la influencia de muchos factores aleatorios en el movimiento de las bacterias. Estos factores incluyen el paisaje de la función de aptitud, la sustitución por miembros más exitosos de la población y el cambio inevitable del vector de movimiento debido a la limitación del número de vidas.
El contador de vidas de las bacterias de la mejor mitad de la colonia aumentará en uno, mientras que las clonadas se reiniciarán.
double r = RNDfromCI (0.0, 1.0); if (r < reproduction) { int st = populationSize / 2; //bacterium original-------------------------------------------------------- for (int s = 0; s < st; s++) { for (int k = 0; k < parameters; k++) { b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++; } //bacterium clone----------------------------------------------------------- for (int s = st; s < populationSize; s++) { for (int k = 0; k < parameters; k++) { int i = (int)RNDfromCI (0, st); if (i >= st) i = st - 1; b [s].c [k] = b [i].cLast [k]; b [s].c [k] = PowerDistribution (b [s].c [k], rangeMin [k], rangeMax [k], powerMut); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT = 0; } }
Luego, si no se cumple la probabilidad reproductiva, en un ciclo para toda la población:
for (int s = 0; s < populationSize; s++)
En primer lugar, se comprobará si se ha alcanzado el límite de vida de la bacteria. Las bacterias que hayan alcanzado su límite vital darán una voltereta y comenzarán a moverse en una nueva dirección, cambiando su vector de movimiento. En este caso, el contador de vidas se pondrá a cero.
Tras comprobar si las bacterias han alcanzado su límite de vida, las que lo hayan alcanzado mutarán, y en la siguiente iteración comenzarán a moverse en una nueva dirección cambiando su vector de movimiento, con el contador de vida a cero.
if (b [s].lifeCNT >= lifeCounter) { for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = PowerDistribution (b [s].cLast [k], rangeMin [k], rangeMax [k], powerMut); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT = 0; }
Si la bacteria aún no ha agotado su límite vital, comprobaremos la presencia de quimiotaxis positiva, es decir, si la bacteria se desplaza hacia un gradiente alimentario creciente. El movimiento de la bacteria se considerará correcto si los valores de aptitud en las dos iteraciones anteriores son iguales. ¿Por qué son precisamente iguales? Porque en el siguiente paso, en el método de evaluación, comprobaremos si se ha producido un cambio positivo en la salud bacteriana. Si los cambios son positivos, el estado actual se asignará al estado anterior. Por eso, si los estados de salud son los mismos en las dos últimas iteraciones, esto indicará que la dirección de desplazamiento de la bacteria en busca de mayor alimento es correcta. De este modo, la bacteria solo podrá avanzar hacia una cantidad de alimento mayor. De lo contrario, la bacteria empezará a dar tumbos por el lugar. Sin embargo, esto no es un problema, porque tarde o temprano la bacteria atascada mutará a un nuevo estado o heredará los genes de progenitores más exitosos y su vector de desplazamiento.
El contador de vidas bacterianas que se mueven en la dirección correcta aumenta gradualmente. Esto significa que, tarde o temprano, incluso las bacterias exitosas sufrirán mutaciones, como hemos mencionado en el bloque de código anterior.
if (b [s].f == b [s].fLast) { for (int k = 0; k < parameters; k++) { b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++; }
Y, si no se observa quimiotaxis positiva en una bacteria, dicha bacteria cambiará su vector de movimiento dando una voltereta. El contador de la vida de tales bacterias también continuará funcionando; aquí se me ocurrió la idea de aumentar el valor del contador de la vida de tales bacterias de bajo éxito con mayor intensidad, aunque no he probado esta conjetura.
Si la bacteria carece de quimiotaxis positiva, cambiará su vector de movimiento dando una voltereta. El contador de vida de tales bacterias seguirá contando; al momento de escribir estas líneas se me ocurrió la idea de aumentar más intensamente el valor del contador de vida de tales bacterias poco exitosas, pero no he probado más a fondo esta suposición.
for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++;
Para realizar la voltereta de la bacteria, es decir, para cambiar el vector de movimiento, se utiliza el método "NewVector", que se ejecutará para el parámetro optimizado con el índice "paramInd":
- luego se generará un número aleatorio "r" con distribución uniforme en el intervalo [-1,0, 1,0] utilizando la función "RNDfromCI".
- el nuevo valor del componente vectorial se calculará multiplicando el intervalo aceptable de valores "v" del parámetro optimizado por el coeficiente "lambda" y un número aleatorio "r".
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BFO_GA::NewVector (int paramInd) { double r = RNDfromCI (-1.0, 1.0); return lambda * v [paramInd] * r; } //——————————————————————————————————————————————————————————————————————————————El método de evaluación se utilizará para organizar el proceso de competición bacteriana y actualizar la solución global.
Al principio de este método, cada bacteria de la población se someterá a una prueba de quimiotaxis positiva: si el valor actual de la función de aptitud "b[s].f" es mayor que el valor anterior "b[s].fLast", se actualizará la aptitud anterior y se actualizarán las coordenadas anteriores desde las que la bacteria se moverá en el futuro.
A continuación, la población se clasificará con la ayuda del método Sorting en un orden descendente de los valores de la función de aptitud utilizando el valor "fLast" de la bacteria.
A continuación, se actualizará la solución global.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO_GA::Evaluation () { for (int s = 0; s < populationSize; s++) { if (b [s].f > b [s].fLast) { b [s].fLast = b [s].f; ArrayCopy (b [s].cLast, b [s].c, 0, 0, WHOLE_ARRAY); } } Sorting (); if (b [0].fLast > fB) { fB = b [0].fLast; ArrayCopy (cB, b [0].cLast, 0, 0, WHOLE_ARRAY); } } //——————————————————————————————————————————————————————————————————————————————
3. Resultados de las pruebas
Impresión del rendimiento del algoritmo híbrido BFO-GA en el banco de pruebas:
C_AO_BFO_GA:50;0.01;0.8;50;10.0
=============================
5 Hilly's; Func runs: 10000; result: 0.8914957961234459
25 Hilly's; Func runs: 10000; result: 0.5511088047221568
500 Hilly's; Func runs: 10000; result: 0.31529201642392934
=============================
5 Forest's; Func runs: 10000; result: 0.9698155977381523
25 Forest's; Func runs: 10000; result: 0.39612322805995287
500 Forest's; Func runs: 10000; result: 0.06304955092899359
=============================
5 Megacity's; Func runs: 10000; result: 0.7266666666666667
25 Megacity's; Func runs: 10000; result: 0.275
500 Megacity's; Func runs: 10000; result: 0.035250000000000004
=============================
All score: 4.22380 (46.93%)
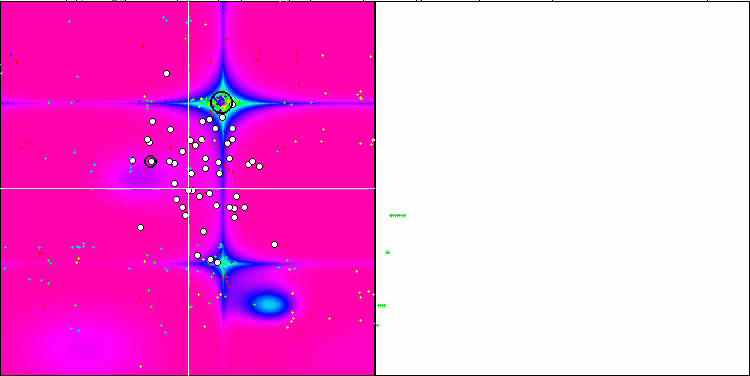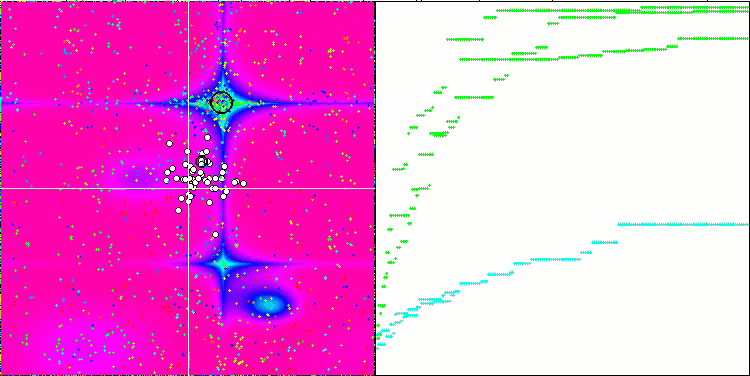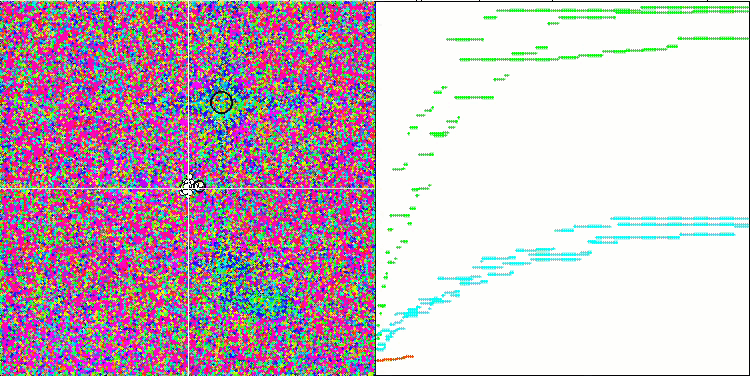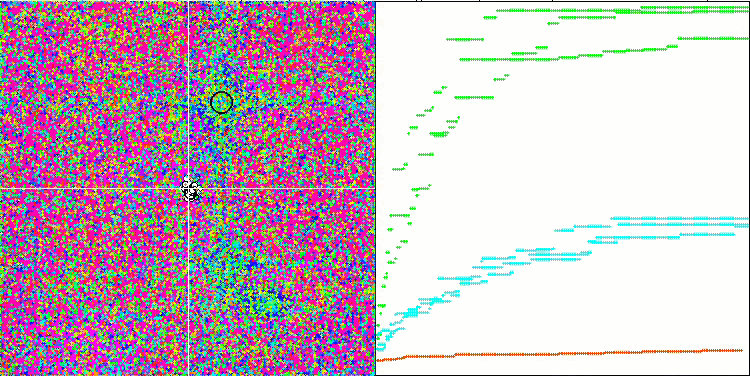
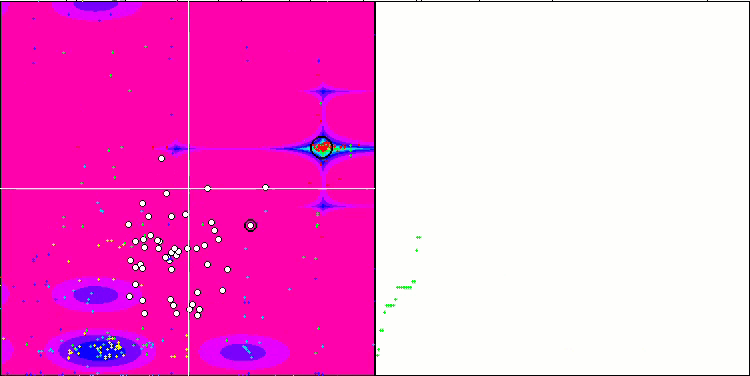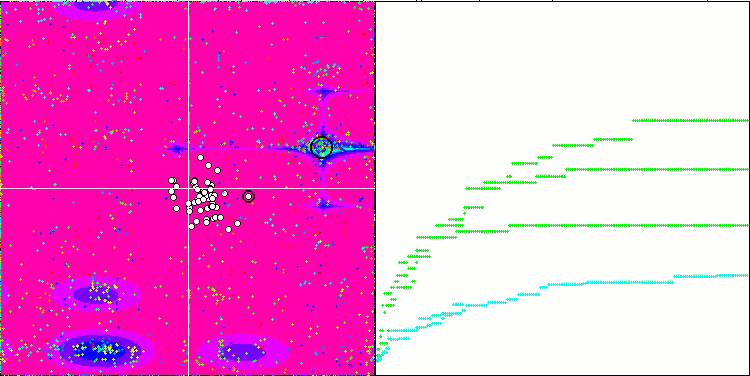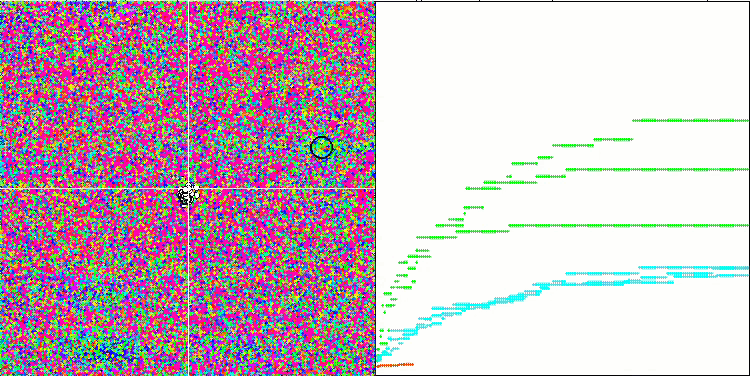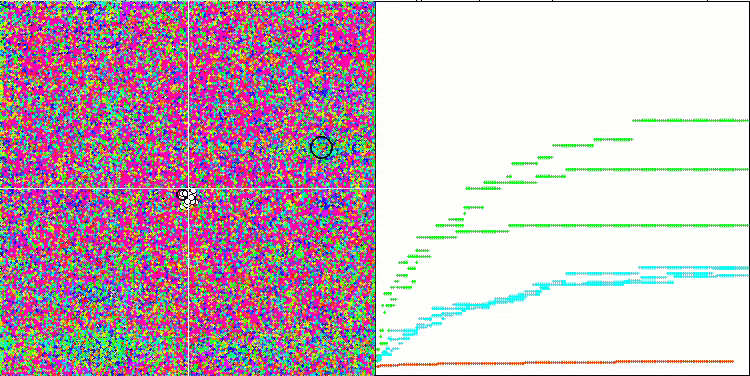
El análisis visual del funcionamiento del banco de pruebas permite observar que el algoritmo BFO-GA detecta rápidamente la región del extremo global, mientras que presta menos atención a la elaboración de los extremos locales, lo que potencialmente puede llevar al atoramiento si el extremo global resulta ser imaginario (erróneo). En general, el algoritmo converge con bastante seguridad, aunque con cierta lentitud. Podemos observar cierto "enjambre", lo cual es una buena señal, sin que exista una asociación completa entre las bacterias, que se comportan como unidades de población independientes.
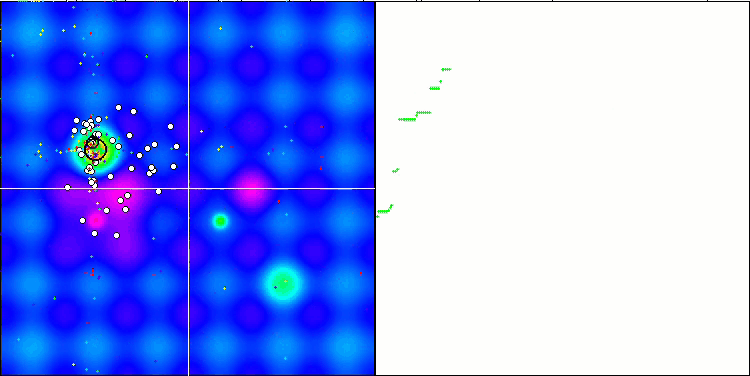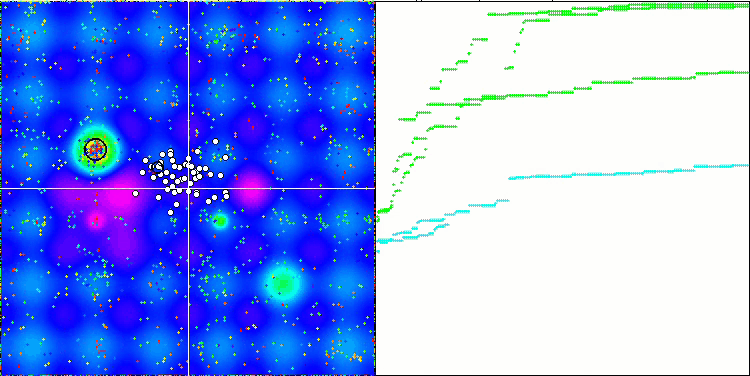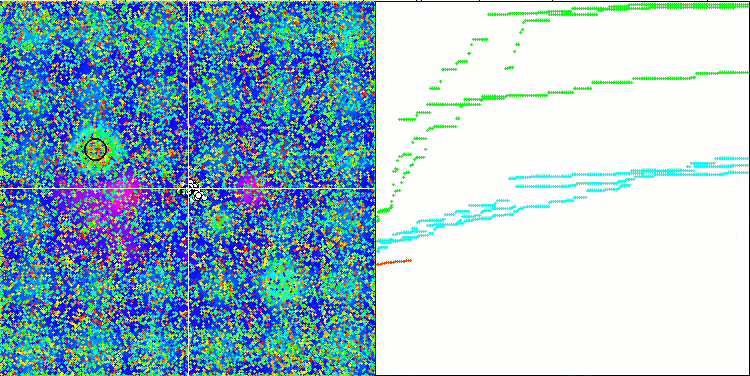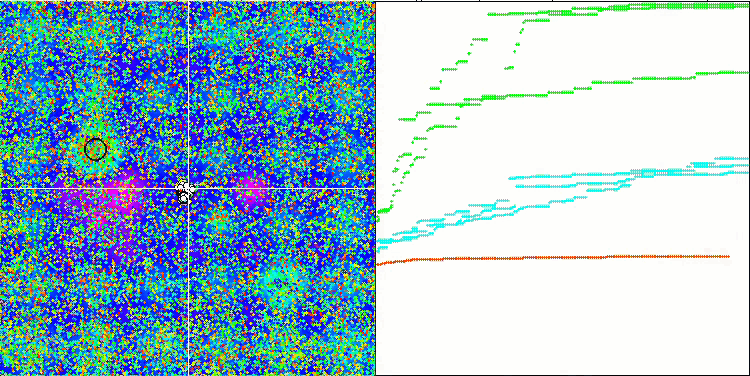
BFO-GA en la función de prueba Hilly.
BFO-GA en la función de prueba Forest.
BFO-GA en la función de prueba Megacity.
En la tabla de clasificación general, podemos ver el algoritmo BFO-GA en noveno lugar, lo cual indica un resultado bastante alto. Esto indica que las transformaciones realizadas al añadir los operadores del algoritmo genético al algoritmo BFO original no han sido en vano y han dado los resultados correspondientes. Predominantemente, este algoritmo ha obtenido los mejores resultados en funciones de prueba con un número reducido de variables, superando a la mayoría de los demás algoritmos.
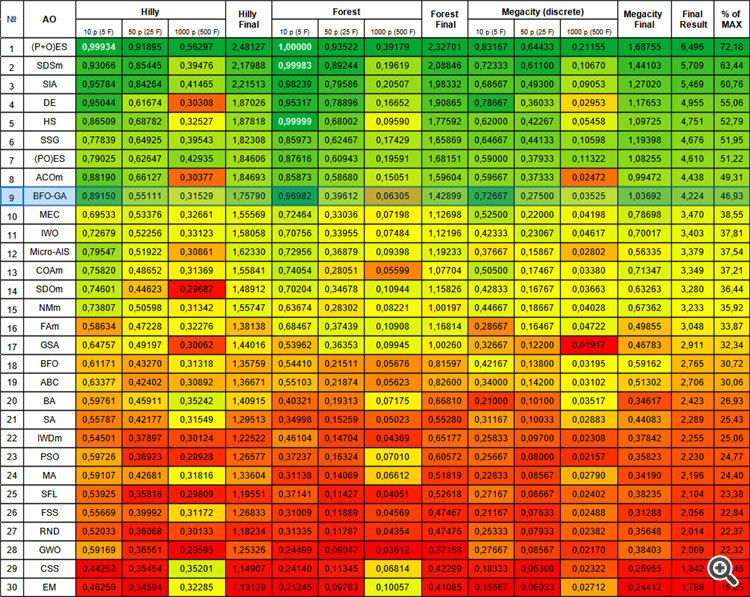
| № | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % de MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | (P+O)ES | (P+O) evolution strategies | 0,99934 | 0,91895 | 0,56297 | 2,48127 | 1,00000 | 0,93522 | 0,39179 | 2,32701 | 0,83167 | 0,64433 | 0,21155 | 1,68755 | 6,496 | 72,18 |
| 2 | SDSm | stochastic diffusion search M | 0,93066 | 0,85445 | 0,39476 | 2,17988 | 0,99983 | 0,89244 | 0,19619 | 2,08846 | 0,72333 | 0,61100 | 0,10670 | 1,44103 | 5,709 | 63,44 |
| 3 | SIA | simulated isotropic annealing | 0,95784 | 0,84264 | 0,41465 | 2,21513 | 0,98239 | 0,79586 | 0,20507 | 1,98332 | 0,68667 | 0,49300 | 0,09053 | 1,27020 | 5,469 | 60,76 |
| 4 | DE | differential evolution | 0,95044 | 0,61674 | 0,30308 | 1,87026 | 0,95317 | 0,78896 | 0,16652 | 1,90865 | 0,78667 | 0,36033 | 0,02953 | 1,17653 | 4,955 | 55,06 |
| 5 | HS | harmony search | 0,86509 | 0,68782 | 0,32527 | 1,87818 | 0,99999 | 0,68002 | 0,09590 | 1,77592 | 0,62000 | 0,42267 | 0,05458 | 1,09725 | 4,751 | 52,79 |
| 6 | SSG | saplings sowing and growing | 0,77839 | 0,64925 | 0,39543 | 1,82308 | 0,85973 | 0,62467 | 0,17429 | 1,65869 | 0,64667 | 0,44133 | 0,10598 | 1,19398 | 4,676 | 51,95 |
| 7 | (PO)ES | (PO) evolution strategies | 0,79025 | 0,62647 | 0,42935 | 1,84606 | 0,87616 | 0,60943 | 0,19591 | 1,68151 | 0,59000 | 0,37933 | 0,11322 | 1,08255 | 4,610 | 51,22 |
| 8 | ACOm | ant colony optimization M | 0,88190 | 0,66127 | 0,30377 | 1,84693 | 0,85873 | 0,58680 | 0,15051 | 1,59604 | 0,59667 | 0,37333 | 0,02472 | 0,99472 | 4,438 | 49,31 |
| 9 | BFO-GA | bacterial foraging optimization - ga | 0,89150 | 0,55111 | 0,31529 | 1,75790 | 0,96982 | 0,39612 | 0,06305 | 1,42899 | 0,72667 | 0,27500 | 0,03525 | 1,03692 | 4,224 | 46,93 |
| 10 | MEC | mind evolutionary computation | 0,69533 | 0,53376 | 0,32661 | 1,55569 | 0,72464 | 0,33036 | 0,07198 | 1,12698 | 0,52500 | 0,22000 | 0,04198 | 0,78698 | 3,470 | 38,55 |
| 11 | IWO | invasive weed optimization | 0,72679 | 0,52256 | 0,33123 | 1,58058 | 0,70756 | 0,33955 | 0,07484 | 1,12196 | 0,42333 | 0,23067 | 0,04617 | 0,70017 | 3,403 | 37,81 |
| 12 | Micro-AIS | micro artificial immune system | 0,79547 | 0,51922 | 0,30861 | 1,62330 | 0,72956 | 0,36879 | 0,09398 | 1,19233 | 0,37667 | 0,15867 | 0,02802 | 0,56335 | 3,379 | 37,54 |
| 13 | COAm | cuckoo optimization algorithm M | 0,75820 | 0,48652 | 0,31369 | 1,55841 | 0,74054 | 0,28051 | 0,05599 | 1,07704 | 0,50500 | 0,17467 | 0,03380 | 0,71347 | 3,349 | 37,21 |
| 14 | SDOm | spiral dynamics optimization M | 0,74601 | 0,44623 | 0,29687 | 1,48912 | 0,70204 | 0,34678 | 0,10944 | 1,15826 | 0,42833 | 0,16767 | 0,03663 | 0,63263 | 3,280 | 36,44 |
| 15 | NMm | Nelder-Mead method M | 0,73807 | 0,50598 | 0,31342 | 1,55747 | 0,63674 | 0,28302 | 0,08221 | 1,00197 | 0,44667 | 0,18667 | 0,04028 | 0,67362 | 3,233 | 35,92 |
| 16 | FAm | firefly algorithm M | 0,58634 | 0,47228 | 0,32276 | 1,38138 | 0,68467 | 0,37439 | 0,10908 | 1,16814 | 0,28667 | 0,16467 | 0,04722 | 0,49855 | 3,048 | 33,87 |
| 17 | GSA | gravitational search algorithm | 0,64757 | 0,49197 | 0,30062 | 1,44016 | 0,53962 | 0,36353 | 0,09945 | 1,00260 | 0,32667 | 0,12200 | 0,01917 | 0,46783 | 2,911 | 32,34 |
| 18 | BFO | bacterial foraging optimization | 0,61171 | 0,43270 | 0,31318 | 1,35759 | 0,54410 | 0,21511 | 0,05676 | 0,81597 | 0,42167 | 0,13800 | 0,03195 | 0,59162 | 2,765 | 30,72 |
| 19 | ABC | artificial bee colony | 0,63377 | 0,42402 | 0,30892 | 1,36671 | 0,55103 | 0,21874 | 0,05623 | 0,82600 | 0,34000 | 0,14200 | 0,03102 | 0,51302 | 2,706 | 30,06 |
| 20 | BA | bat algorithm | 0,59761 | 0,45911 | 0,35242 | 1,40915 | 0,40321 | 0,19313 | 0,07175 | 0,66810 | 0,21000 | 0,10100 | 0,03517 | 0,34617 | 2,423 | 26,93 |
| 21 | SA | simulated annealing | 0,55787 | 0,42177 | 0,31549 | 1,29513 | 0,34998 | 0,15259 | 0,05023 | 0,55280 | 0,31167 | 0,10033 | 0,02883 | 0,44083 | 2,289 | 25,43 |
| 22 | IWDm | intelligent water drops M | 0,54501 | 0,37897 | 0,30124 | 1,22522 | 0,46104 | 0,14704 | 0,04369 | 0,65177 | 0,25833 | 0,09700 | 0,02308 | 0,37842 | 2,255 | 25,06 |
| 23 | PSO | particle swarm optimisation | 0,59726 | 0,36923 | 0,29928 | 1,26577 | 0,37237 | 0,16324 | 0,07010 | 0,60572 | 0,25667 | 0,08000 | 0,02157 | 0,35823 | 2,230 | 24,77 |
| 24 | MA | monkey algorithm | 0,59107 | 0,42681 | 0,31816 | 1,33604 | 0,31138 | 0,14069 | 0,06612 | 0,51819 | 0,22833 | 0,08567 | 0,02790 | 0,34190 | 2,196 | 24,40 |
| 25 | SFL | shuffled frog-leaping | 0,53925 | 0,35816 | 0,29809 | 1,19551 | 0,37141 | 0,11427 | 0,04051 | 0,52618 | 0,27167 | 0,08667 | 0,02402 | 0,38235 | 2,104 | 23,38 |
| 26 | FSS | fish school search | 0,55669 | 0,39992 | 0,31172 | 1,26833 | 0,31009 | 0,11889 | 0,04569 | 0,47467 | 0,21167 | 0,07633 | 0,02488 | 0,31288 | 2,056 | 22,84 |
| 27 | RND | random | 0,52033 | 0,36068 | 0,30133 | 1,18234 | 0,31335 | 0,11787 | 0,04354 | 0,47476 | 0,25333 | 0,07933 | 0,02382 | 0,35648 | 2,014 | 22,37 |
| 28 | GWO | grey wolf optimizer | 0,59169 | 0,36561 | 0,29595 | 1,25326 | 0,24499 | 0,09047 | 0,03612 | 0,37158 | 0,27667 | 0,08567 | 0,02170 | 0,38403 | 2,009 | 22,32 |
| 29 | CSS | charged system search | 0,44252 | 0,35454 | 0,35201 | 1,14907 | 0,24140 | 0,11345 | 0,06814 | 0,42299 | 0,18333 | 0,06300 | 0,02322 | 0,26955 | 1,842 | 20,46 |
| 30 | EM | electroMagnetism-like algorithm | 0,46250 | 0,34594 | 0,32285 | 1,13129 | 0,21245 | 0,09783 | 0,10057 | 0,41085 | 0,15667 | 0,06033 | 0,02712 | 0,24412 | 1,786 | 19,85 |
Conclusiones
Podemos observar claras mejoras en los resultados de BFO-GA en comparación con el algoritmo BFO original, lo cual se debe al uso de los operadores del algoritmo genético: selección, mutación y herencia. El BFO antes no disponía de mecanismos para salir de los extremos locales, y este problema ha desaparecido. El algoritmo presenta enormes oportunidades para la experimentación, combinando el orden de los componentes de la estrategia de búsqueda bacteriana y los operadores GA, muchos de los cuales aún no hemos probado.
En el algoritmo BFO, antes habíamos cometido un error al calcular el número de vidas bacterianas, pero este error ha sido corregido. El BFO ha mejorado ligeramente y subido un puesto por encima del ABC. Me gustaría señalar que, al escribir algoritmos de optimización, resulta difícil detectar errores en el código y en la lógica de la estrategia de búsqueda. Incluso si hay errores, el algoritmo casi siempre continúa la búsqueda. Los errores no siempre empeoran los resultados; hay veces en que los resultados mejoran significativamente y el "error" se convierte en parte de la estrategia. Siempre conviene recordar que, en los algoritmos de optimización, los grandes cambios en la lógica no siempre provocan mejoras significativas en los resultados, pero los pequeños cambios a veces pueden dar lugar a mejoras espectaculares. La versión corregida del BFO se encuentra en el archivo del artículo.
Figura 2. Gradación por colores de los algoritmos según sus respectivas pruebas.
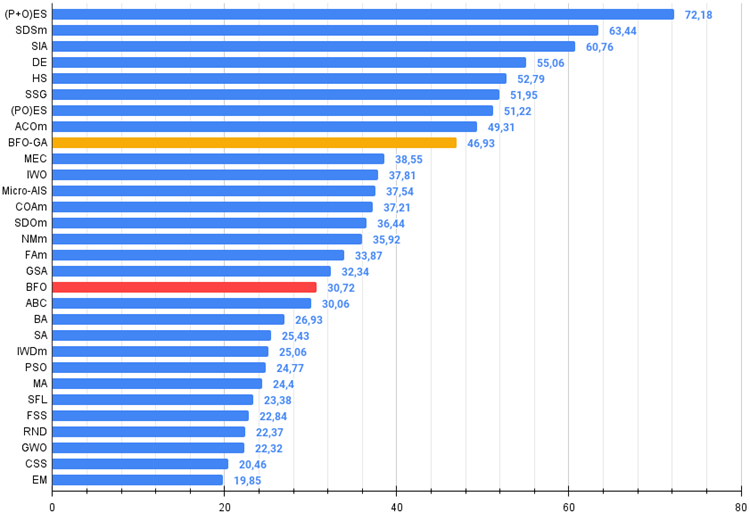
Figura 3. Histograma con los resultados de las pruebas de los algoritmos (en una escala de 0 a 100, cuanto mayor, mejor),
donde 100 es el máximo resultado teórico posible; hay un script para calcular la tabla de puntuación en el archivo).
Ventajas y desventajas del algoritmo híbrido de optimización de forrajeo bacteriano con algoritmo genético (BFO-GA):
- Alta velocidad del algoritmo.
- Implementación sencilla.
- Buena convergencia en funciones con pocos parámetros.
- Baja convergencia en problemas con gran espacio de búsqueda.
Adjuntamos al artículo un archivo con las versiones reales actualizadas de los códigos de los algoritmos descritos en los artículos anteriores. El autor de este artículo no se responsabiliza de la exactitud absoluta de la descripción de los algoritmos canónicos, muchos de ellos han sido modificados para mejorar las capacidades de búsqueda. Las conclusiones y juicios presentados en los artículos se basan en los resultados de los experimentos realizados.
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/14011
 Aprendiendo MQL5 de principiante a profesional (Parte II): Tipos de datos básicos y uso de variables
Aprendiendo MQL5 de principiante a profesional (Parte II): Tipos de datos básicos y uso de variables
 Redes neuronales: así de sencillo (Parte 71): Previsión de estados futuros basada en objetivos (GCPC)
Redes neuronales: así de sencillo (Parte 71): Previsión de estados futuros basada en objetivos (GCPC)
 Algoritmos de optimización de la población: microsistema inmune artificial (Micro Artificial immune system, Micro-AIS)
Algoritmos de optimización de la población: microsistema inmune artificial (Micro Artificial immune system, Micro-AIS)
 Redes neuronales: así de sencillo (Parte 70): Mejoramos las políticas usando operadores de forma cerrada (CFPI)
Redes neuronales: así de sencillo (Parte 70): Mejoramos las políticas usando operadores de forma cerrada (CFPI)
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso