El método de agrupamiento para el manejo de datos: Implementación del algoritmo iterativo multicapa en MQL5
Introducción
El método de agrupamiento para el manejo de datos (GMDH, Group Method of Data Handling) es una familia de algoritmos inductivos utilizados para el modelado informático de datos. Los algoritmos funcionan construyendo y optimizando automáticamente modelos de redes neuronales polinómicas a partir de datos, lo que ofrece un enfoque único para descubrir las relaciones entre las variables de entrada y de salida.Tradicionalmente, el marco de trabajo GMDH constaba de cuatro algoritmos principales: el algoritmo combinatorio (COMBI), el algoritmo combinatorio selectivo (MULTI), el algoritmo iterativo multicapa (MIA) y el algoritmo iterativo de relajación (RIA). En este artículo exploraremos la implementación del algoritmo iterativo multicapa en MQL5. Discutiremos su funcionamiento interno y también demostraremos cómo se puede aplicar para construir modelos predictivos a partir de conjuntos de datos.
Comprender GMDH: El método de agrupamiento para el manejo de datos
El método de agrupamiento para el manejo de datos es un tipo de algoritmo utilizado para el análisis y la predicción de datos. Es una técnica de aprendizaje automático cuyo objetivo es encontrar el mejor modelo matemático para describir un conjunto de datos determinado. El GMDH fue desarrollado por el matemático soviético Alexey Ivakhnenko en la década de 1960. Se desarrolló para abordar los retos asociados a la modelización de sistemas complejos a partir de datos empíricos. Los algoritmos GMDH emplean un enfoque de modelización basado en los datos, en el que los modelos se generan y perfeccionan a partir de los datos observados y no de nociones preconcebidas o supuestos teóricos.
Una de las principales ventajas de GMDH es que automatiza el proceso de construcción de modelos mediante la generación y evaluación iterativa de modelos candidatos. Seleccionando los modelos más eficaces y perfeccionarlos a partir de los datos obtenidos. Esta automatización reduce la necesidad de intervención manual y de conocimientos especializados en la construcción del modelo.
La idea clave del GMDH es construir una serie de modelos de complejidad y precisión crecientes seleccionando y combinando variables de forma iterativa. El algoritmo comienza con un conjunto de modelos sencillos (normalmente modelos lineales) y aumenta gradualmente su complejidad añadiendo variables y términos adicionales. En cada paso, el algoritmo evalúa el rendimiento de los modelos y selecciona los más eficaces como base para la siguiente iteración. Este proceso continúa hasta que se obtiene un modelo satisfactorio o hasta que se cumple un criterio de parada.
GMDH es especialmente adecuado para modelizar conjuntos de datos con un gran número de variables de entrada y relaciones complejas entre ellas. Las técnicas GMDH dan lugar a modelos que relacionan las entradas con una salida que puede representarse mediante un polinomio infinito Volterra-Kolmogorov-Gabor (VKG). Un polinomio Volterra-Kolmogorov-Gabor (VKG) es un tipo específico de polinomio utilizado para modelar sistemas no lineales y aproximar datos complejos. Un polinomio VKG tiene la siguiente forma:
Donde:
- Yn es la salida del sistema.
- Xi, Xj y Xk son las variables de entrada en los tiempos i, j y k, respectivamente.
- ai, aj, ak, etc. son los coeficientes del polinomio.
Un polinomio de este tipo puede considerarse una red neuronal polinómica (PNN). La estructura de una red neuronal polinómica es similar a la de otras redes neuronales, con nodos de entrada, capas ocultas y nodos de salida. Sin embargo, las funciones de activación aplicadas a las neuronas en las PNN son funciones polinómicas. Los algoritmos paramétricos GMDH se desarrollaron específicamente para manejar variables continuas. Cuando el objeto que se modela se caracteriza por propiedades que carecen de ambigüedad en su representación o definición. El algoritmo iterativo multicapa es un ejemplo de algoritmo GMDH paramétrico.
Algoritmo iterativo multicapa
MIA es una variante del marco GMDH para construir modelos de redes neuronales polinómicas. Su estructura es casi idéntica a la de una red neuronal multicapa. La información fluye desde la capa de entrada a través de capas intermedias hasta la salida final. Cada capa realiza transformaciones específicas de los datos. En relación con el método general del GMDH, la característica diferenciadora clave del MIA reside en la selección de las subfunciones óptimas del polinomio final que mejor describen los datos. Significa que parte de la información obtenida a través de la formación se descarta de acuerdo con un criterio predefinido.
Para construir un modelo utilizando MIA, empezamos particionando el conjunto de datos que queremos estudiar, en conjuntos de entrenamiento y de prueba. Queremos tener la mayor variedad posible en el conjunto de entrenamiento para captar adecuadamente las características del proceso subyacente. Comenzamos con la construcción de capas una vez que está hecho.
Construcción de capas
Al igual que en una red neuronal multicapa 'feedforward', comenzamos con la capa de entrada, que es la colección de predictores o variables independientes. Estas entradas se toman de dos en dos y se envían a la primera capa de la red. Por tanto, la primera capa estará formada por "M combinaciones de 2" nodos, donde M es el número de predictores.
La ilustración anterior muestra un ejemplo de cómo serían la capa de entrada y la primera capa si se tratara de 4 entradas (denominadas x1..x4). En la primera capa, se construirán modelos parciales basados en las entradas de un nodo utilizando el conjunto de datos de entrenamiento y el modelo parcial resultante se evaluará frente al conjunto de datos de prueba. A continuación, se compara el error de predicción de todos los modelos parciales de la capa. Los N mejores modelos se anotan y se utilizan para generar las entradas de la capa siguiente. El error de predicción de los N modelos superiores de una capa se combinan de alguna manera para obtener una única medida que dé una indicación del progreso general en la generación de modelos. Que se compara con la cifra de la capa anterior. Si es menor, se crea una nueva capa y se repite el proceso. De lo contrario, si no hay mejora. La generación del modelo se detiene y los datos de la capa actual se descartan, lo que indica que el entrenamiento del modelo habría finalizado.
Nodos y modelos parciales
En cada nodo de una capa, se calcula un polinomio que estima las observaciones en el conjunto de datos de entrenamiento dado el par de entradas de salida de la capa anterior. Es lo que se denomina un modelo parcial. A continuación se muestra un ejemplo de ecuación utilizada para modelar las salidas del conjunto de entrenamiento dadas las entradas del nodo.
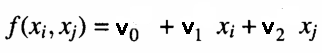
Donde "v" son los coeficientes del modelo lineal ajustado. La bondad del ajuste se comprueba determinando el error cuadrático medio de las predicciones frente a los valores reales, en el conjunto de datos de prueba. A continuación, estas medidas de error se combinan de alguna manera. Ya sea calculando su media o simplemente seleccionando el nodo con el menor error cuadrático medio. Esta medida final da una indicación de si las aproximaciones están mejorando o no en relación con otras capas. Al mismo tiempo, se anotan los N mejores nodos con el menor error de predicción. Y los coeficientes correspondientes se utilizan para generar los valores de un conjunto de nuevas entradas para la capa siguiente. Si las aproximaciones de la capa actual son mejores (en este caso menores) que las de la capa anterior, se construirá una nueva capa.
Una vez completada la red, sólo se conservan los coeficientes de los nodos que tuvieron el mejor error de predicción en cada capa y se utilizan para definir el modelo final que mejor describe los datos. En la siguiente sección, nos adentramos en el código que implementa el procedimiento que acabamos de describir. El código está adaptado de una implementación en C++ del GMDH disponible en GitHub.
Implementación en MQL5
La implementación en C++ entrena modelos y los guarda en formato JSON en un archivo de texto para su uso posterior. Aprovecha el multihilo para acelerar el entrenamiento y está construido utilizando las bibliotecas Boost y Eigen. Para nuestra implementación en MQL5, la mayoría de las características se mantendrán excepto el entrenamiento multihilo y la disponibilidad de opciones alternativas para la Factorización QR para resolver ecuaciones lineales.
Nuestra implementación constará de tres archivos de cabecera. El primero es gmdh_internal.mqh. Este archivo contiene definiciones para varios tipos de datos personalizados. Comienza definiendo tres enumeraciones:
- PolynomialType - especifica el tipo de polinomio utilizado para transformar las variables existentes antes de emprender otra ronda de entrenamiento.
//+---------------------------------------------------------------------------------------------------------+ //| Enumeration for specifying the polynomial type to be used to construct new variables from existing ones| //+---------------------------------------------------------------------------------------------------------+ enum PolynomialType { linear, linear_cov, quadratic };
"PolynomialType" expone tres opciones que representan las funciones polimíticas a continuación, aquí x1 y x2 son las entradas a la función f(x1,x2) y v0...vN son los coeficientes a encontrar. La enumeración representa el tipo de ecuaciones a partir de las cuales se generará el conjunto de soluciones:
| Opción | Función f(x1,x2) |
|---|---|
| linear | Ecuación lineal: v0 + v1*x1 + v2*x2 |
| linear_cov | Ecuación lineal con covariación: v0 + v1*x1 + v2*x2 + v3*x1*x2 |
| quadratic | Ecuación cuadrática: v0 + v1*x1 + v2*x2 + v3*x1*x2 + v4*x1^2 + v5*x2^2 |
- Solver - determina el método de factorización QR utilizado para resolver ecuaciones lineales. Nuestra aplicación sólo tendrá una opción utilizable. La versión C++ emplea variaciones del método Householder para la factorización QR utilizando la biblioteca Eigen.
//+-----------------------------------------------------------------------------------------------+ //| Enum for specifying the QR decomposition method for linear equations solving in models. | //+-----------------------------------------------------------------------------------------------+ enum Solver { fast, accurate, balanced };
- CriterionType - permite a los usuarios seleccionar un criterio externo específico que se utilizará como base para evaluar los modelos candidatos. La enumeración recoge las opciones que se pueden utilizar como criterios de parada al entrenar un modelo.
//+------------------------------------------------------------------+ //|Enum for specifying the external criterion | //+------------------------------------------------------------------+ enum CriterionType { reg, symReg, stab, symStab, unbiasedOut, symUnbiasedOut, unbiasedCoef, absoluteNoiseImmun, symAbsoluteNoiseImmun };
Las opciones disponibles se explican con más detalle en la tabla siguiente:
| CriterionType | Descripción |
|---|---|
| reg | Regularidad: Aplica la suma regular de errores al cuadrado (SSE) basada en la diferencia entre los objetivos del conjunto de datos de prueba y las predicciones realizadas con coeficientes calculados utilizando el conjunto de datos de entrenamiento en combinación con los predictores del conjunto de datos de prueba. |
| symReg | Regularidad simétrica: Es la suma de la SSE basada en la diferencia entre los objetivos del conjunto de datos de prueba y las predicciones realizadas con coeficientes calculados utilizando el conjunto de datos de entrenamiento en combinación con los predictores del conjunto de datos de prueba y la SSE basada en la diferencia entre los objetivos del conjunto de datos de entrenamiento y las predicciones realizadas con coeficientes calculados utilizando el conjunto de datos de prueba en combinación con los predictores del conjunto de datos de entrenamiento. |
| stab | Estabilidad: Utiliza el SSE basado en la diferencia entre todos los objetivos y las predicciones realizadas con coeficientes calculados utilizando el conjunto de datos de entrenamiento en combinación con todos los predictores. |
| symStab | Estabilidad simétrica: Este criterio combina la SSE calculada de forma similar al criterio de "estabilidad", así como la SSE basada en la diferencia entre todos los objetivos y las predicciones realizadas con coeficientes calculados utilizando el conjunto de datos de prueba en combinación con todos los predictores del conjunto de datos. |
| unbiasedOut | Resultados imparciales: El SSE se basa en la diferencia entre las predicciones realizadas con los coeficientes calculados utilizando el conjunto de datos de entrenamiento y las predicciones realizadas con los coeficientes calculados utilizando el conjunto de datos de prueba, ambos utilizando los predictores del conjunto de datos de prueba. |
| symUnbiasedOut | Resultados imparciales simétricos: Calcula el SSE de la misma manera que el criterio de "resultados imparciales", pero en esta ocasión utilizamos todos los predictores. |
| unbiasedCoef | Coeficientes imparciales: La suma de las diferencias cuadradas entre los coeficientes calculados utilizando los datos de entrenamiento y los coeficientes calculados utilizando los datos de prueba. |
| absoluteNoiseImmun | Inmunidad al ruido absoluto: Al utilizar esta opción, el criterio se calcula como el producto punto entre las predicciones del modelo entrenado con el conjunto de datos completo y las predicciones del modelo entrenado con el conjunto de datos de entrenamiento, cuando se aplican al conjunto de datos de prueba, y las predicciones del modelo entrenado con el conjunto de datos de prueba y las predicciones del modelo entrenado con el conjunto de datos de entrenamiento, cuando se aplican al conjunto de datos de prueba. |
| symAbsoluteNoiseImmun | Inmunidad absoluta al ruido simétrica: Aquí el criterio es el producto punto entre las predicciones del modelo entrenado con el conjunto de datos completo menos las predicciones del modelo entrenado solo con el conjunto de datos de entrenamiento, cuando se aplican al conjunto de datos de entrenamiento, y las predicciones del modelo entrenado con el conjunto de datos completo menos las predicciones del modelo entrenado con el conjunto de datos de prueba, cuando se aplican a todas las observaciones. |
Las enumeraciones son seguidas por cuatro estructuras personalizadas:
- BufferValues - Es una estructura de vectores utilizada para almacenar coeficientes y valores predichos calculados de varias formas utilizando los conjuntos de datos de prueba y de entrenamiento.
//+-------------------------------------------------------------------------------------+ //| Structure for storing coefficients and predicted values calculated in different ways| //+--------------------------------------------------------------------------------------+ struct BufferValues { vector coeffsTrain; // Coefficients vector calculated using training data vector coeffsTest; // Coefficients vector calculated using testing data vector coeffsAll; // Coefficients vector calculated using learning data vector yPredTrainByTrain; // Predicted values for *training* data calculated using coefficients vector calculated on *training* data vector yPredTrainByTest; // Predicted values for *training* data calculated using coefficients vector calculated on *testing* data vector yPredTestByTrain; // Predicted values for *testing* data calculated using coefficients vector calculated on *training* data vector yPredTestByTest; //Predicted values for *testing* data calculated using coefficients vector calculated on *testing* data BufferValues(void) { } BufferValues(BufferValues &other) { coeffsTrain = other.coeffsTrain; coeffsTest = other.coeffsTest; coeffsAll = other.coeffsAll; yPredTrainByTrain = other.yPredTrainByTrain; yPredTrainByTest = other.yPredTrainByTest; yPredTestByTrain = other.yPredTestByTrain; yPredTestByTest = other.yPredTestByTest; } BufferValues operator=(BufferValues &other) { coeffsTrain = other.coeffsTrain; coeffsTest = other.coeffsTest; coeffsAll = other.coeffsAll; yPredTrainByTrain = other.yPredTrainByTrain; yPredTrainByTest = other.yPredTrainByTest; yPredTestByTrain = other.yPredTestByTrain; yPredTestByTest = other.yPredTestByTest; return this; } };
- PairDVXd - Encapsula una estructura de datos que combina un escalar y un vector correspondiente.
//+------------------------------------------------------------------+ //| struct PairDV | //+------------------------------------------------------------------+ struct PairDVXd { double first; vector second; PairDVXd(void) { first = 0.0; second = vector::Zeros(10); } PairDVXd(double &_f, vector &_s) { first = _f; second.Copy(_s); } PairDVXd(PairDVXd &other) { first = other.first; second = other.second; } PairDVXd operator=(PairDVXd& other) { first = other.first; second = other.second; return this; } };
- PairMVXd - Es una estructura que combina una matriz y un vector. Juntos almacenan las entradas y las correspondientes salidas o valores objetivo. Las entradas se guardan en la matriz y el vector es la colección de salidas. Cada fila de la matriz corresponde a un valor del vector.
//+------------------------------------------------------------------+ //| structure PairMVXd | //+------------------------------------------------------------------+ struct PairMVXd { matrix first; vector second; PairMVXd(void) { first = matrix::Zeros(10,10); second = vector::Zeros(10); } PairMVXd(matrix &_f, vector& _s) { first = _f; second = _s; } PairMVXd(PairMVXd &other) { first = other.first; second = other.second; } PairMVXd operator=(PairMVXd &other) { first = other.first; second = other.second; return this; } };
- SplittedData - Esta estructura de datos almacena los conjuntos de datos particionados para entrenamiento y prueba.
//+------------------------------------------------------------------+ //| Structure for storing parts of a split dataset | //+------------------------------------------------------------------+ struct SplittedData { matrix xTrain; matrix xTest; vector yTrain; vector yTest; SplittedData(void) { xTrain = matrix::Zeros(10,10); xTest = matrix::Zeros(10,10); yTrain = vector::Zeros(10); yTest = vector::Zeros(10); } SplittedData(SplittedData &other) { xTrain = other.xTrain; xTest = other.xTest; yTrain = other.yTrain; yTest = other.yTest; } SplittedData operator=(SplittedData &other) { xTrain = other.xTrain; xTest = other.xTest; yTrain = other.yTrain; yTest = other.yTest; return this; } };
Después de las estructuras, pasamos a las definiciones de las clases:
- La clase 'Combination' representa un modelo candidato. Almacena los criterios de evaluación, la combinación de entradas y los coeficientes calculados para un modelo.
//+------------------------------------------------------------------+ //| Сlass representing the candidate model of the GMDH algorithm | //+------------------------------------------------------------------+ class Combination { vector _combination,_bestCoeffs; double _evaluation; public: Combination(void) { _combination = vector::Zeros(10); _bestCoeffs.Copy(_combination); _evaluation = DBL_MAX; } Combination(vector &comb) : _combination(comb) { _bestCoeffs=vector::Zeros(_combination.Size()); _evaluation = DBL_MAX;} Combination(vector &comb, vector &coeffs) : _combination(comb),_bestCoeffs(coeffs) { _evaluation = DBL_MAX; } Combination(Combination &other) { _combination = other.combination(); _bestCoeffs=other.bestCoeffs(); _evaluation = other.evaluation();} vector combination(void) { return _combination;} vector bestCoeffs(void) { return _bestCoeffs; } double evaluation(void) { return _evaluation; } void setCombination(vector &combination) { _combination = combination; } void setBestCoeffs(vector &bestcoeffs) { _bestCoeffs = bestcoeffs; } void setEvaluation(double evaluation) { _evaluation = evaluation; } bool operator<(Combination &combi) { return _evaluation<combi.evaluation();} Combination operator=(Combination &combi) { _combination = combi.combination(); _bestCoeffs = combi.bestCoeffs(); _evaluation = combi.evaluation(); return this; } };
- CVector - Define un contenedor personalizado de tipo vectorial que almacena una colección de instancias de 'Combination'. Convirtiéndolo en un contenedor de modelos candidatos.
//+------------------------------------------------------------------+ //| collection of Combination instances | //+------------------------------------------------------------------+ class CVector { protected: Combination m_array[]; int m_size; int m_reserve; public: //+------------------------------------------------------------------+ //| default constructor | //+------------------------------------------------------------------+ CVector(void) :m_size(0),m_reserve(1000) { } //+------------------------------------------------------------------+ //| parametric constructor specifying initial size | //+------------------------------------------------------------------+ CVector(int size, int mem_reserve = 1000) :m_size(size),m_reserve(mem_reserve) { ArrayResize(m_array,m_size,m_reserve); } //+------------------------------------------------------------------+ //| Copy constructor | //+------------------------------------------------------------------+ CVector(CVector &other) { m_size = other.size(); m_reserve = other.reserve(); ArrayResize(m_array,m_size,m_reserve); for(int i=0; i<m_size; ++i) m_array[i]=other[i]; } //+------------------------------------------------------------------+ //| destructor | //+------------------------------------------------------------------+ ~CVector(void) { } //+------------------------------------------------------------------+ //| Add element to end of array | //+------------------------------------------------------------------+ bool push_back(Combination &value) { ResetLastError(); if(ArrayResize(m_array,int(m_array.Size()+1),m_reserve)<m_size+1) { Print(__FUNCTION__," Critical error: failed to resize underlying array ", GetLastError()); return false; } m_array[m_size++]=value; return true; } //+------------------------------------------------------------------+ //| set value at specified index | //+------------------------------------------------------------------+ bool setAt(int index, Combination &value) { ResetLastError(); if(index < 0 || index >= m_size) { Print(__FUNCTION__," index out of bounds "); return false; } m_array[index]=value; return true; } //+------------------------------------------------------------------+ //|access by index | //+------------------------------------------------------------------+ Combination* operator[](int index) { return GetPointer(m_array[uint(index)]); } //+------------------------------------------------------------------+ //|overload assignment operator | //+------------------------------------------------------------------+ CVector operator=(CVector &other) { clear(); m_size = other.size(); m_reserve = other.reserve(); ArrayResize(m_array,m_size,m_reserve); for(int i=0; i<m_size; ++i) m_array[i]= other[i]; return this; } //+------------------------------------------------------------------+ //|access last element | //+------------------------------------------------------------------+ Combination* back(void) { return GetPointer(m_array[m_size-1]); } //+-------------------------------------------------------------------+ //|access by first index | //+------------------------------------------------------------------+ Combination* front(void) { return GetPointer(m_array[0]); } //+------------------------------------------------------------------+ //| Get current size of collection ,the number of elements | //+------------------------------------------------------------------+ int size(void) { return ArraySize(m_array); } //+------------------------------------------------------------------+ //|Get the reserved memory size | //+------------------------------------------------------------------+ int reserve(void) { return m_reserve; } //+------------------------------------------------------------------+ //|set the reserved memory size | //+------------------------------------------------------------------+ void reserve(int new_reserve) { if(new_reserve > 0) m_reserve = new_reserve; } //+------------------------------------------------------------------+ //| clear | //+------------------------------------------------------------------+ void clear(void) { ArrayFree(m_array); m_size = 0; } };
- CVector2d - Es otro contenedor personalizado de tipo vectorial, que almacena una colección de instancias de CVector.
//+------------------------------------------------------------------+ //| Collection of CVector instances | //+------------------------------------------------------------------+ class CVector2d { protected: CVector m_array[]; int m_size; int m_reserve; public: //+------------------------------------------------------------------+ //| default constructor | //+------------------------------------------------------------------+ CVector2d(void) :m_size(0),m_reserve(1000) { } //+------------------------------------------------------------------+ //| parametric constructor specifying initial size | //+------------------------------------------------------------------+ CVector2d(int size, int mem_reserve = 1000) :m_size(size),m_reserve(mem_reserve) { ArrayResize(m_array,m_size,m_reserve); } //+------------------------------------------------------------------+ //| Copy constructor | //+------------------------------------------------------------------+ CVector2d(CVector2d &other) { m_size = other.size(); m_reserve = other.reserve(); ArrayResize(m_array,m_size,m_reserve); for(int i=0; i<m_size; ++i) m_array[i]= other[i]; } //+------------------------------------------------------------------+ //| destructor | //+------------------------------------------------------------------+ ~CVector2d(void) { } //+------------------------------------------------------------------+ //| Add element to end of array | //+------------------------------------------------------------------+ bool push_back(CVector &value) { ResetLastError(); if(ArrayResize(m_array,int(m_array.Size()+1),m_reserve)<m_size+1) { Print(__FUNCTION__," Critical error: failed to resize underlying array ", GetLastError()); return false; } m_array[m_size++]=value; return true; } //+------------------------------------------------------------------+ //| set value at specified index | //+------------------------------------------------------------------+ bool setAt(int index, CVector &value) { ResetLastError(); if(index < 0 || index >= m_size) { Print(__FUNCTION__," index out of bounds "); return false; } m_array[index]=value; return true; } //+------------------------------------------------------------------+ //|access by index | //+------------------------------------------------------------------+ CVector* operator[](int index) { return GetPointer(m_array[uint(index)]); } //+------------------------------------------------------------------+ //|overload assignment operator | //+------------------------------------------------------------------+ CVector2d operator=(CVector2d &other) { clear(); m_size = other.size(); m_reserve = other.reserve(); ArrayResize(m_array,m_size,m_reserve); for(int i=0; i<m_size; ++i) m_array[i]= other[i]; return this; } //+------------------------------------------------------------------+ //|access last element | //+------------------------------------------------------------------+ CVector* back(void) { return GetPointer(m_array[m_size-1]); } //+-------------------------------------------------------------------+ //|access by first index | //+------------------------------------------------------------------+ CVector* front(void) { return GetPointer(m_array[0]); } //+------------------------------------------------------------------+ //| Get current size of collection ,the number of elements | //+------------------------------------------------------------------+ int size(void) { return ArraySize(m_array); } //+------------------------------------------------------------------+ //|Get the reserved memory size | //+------------------------------------------------------------------+ int reserve(void) { return m_reserve; } //+------------------------------------------------------------------+ //|set the reserved memory size | //+------------------------------------------------------------------+ void reserve(int new_reserve) { if(new_reserve > 0) m_reserve = new_reserve; } //+------------------------------------------------------------------+ //| clear | //+------------------------------------------------------------------+ void clear(void) { for(uint i = 0; i<m_array.Size(); i++) m_array[i].clear(); ArrayFree(m_array); m_size = 0; } };
- Criterion - Esta clase implementa el cálculo de varios criterios externos basados en un tipo de criterio seleccionado.
//+---------------------------------------------------------------------------------+ //|Class that implements calculations of internal and individual external criterions| //+---------------------------------------------------------------------------------+ class Criterion { protected: CriterionType criterionType; // Selected CriterionType object Solver solver; // Selected Solver object public: /** Implements the internal criterion calculation param xTrain Matrix of input variables that should be used to calculate the model coefficients param yTrain Target values vector for the corresponding xTrain parameter return Coefficients vector representing a solution of the linear equations system constructed from the parameters data */ vector findBestCoeffs(matrix& xTrain, vector& yTrain) { vector solution; matrix q,r; xTrain.QR(q,r); matrix qT = q.Transpose(); vector y = qT.MatMul(yTrain); solution = r.LstSq(y); return solution; } /** Calculate the value of the selected external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param _criterionType Selected external criterion type param bufferValues Temporary storage for calculated coefficients and target values return The value of external criterion and calculated model coefficients */ PairDVXd getResult(matrix& xTrain, matrix& xTest, vector& yTrain, vector& yTest, CriterionType _criterionType, BufferValues& bufferValues) { switch(_criterionType) { case reg: return regularity(xTrain, xTest, yTrain, yTest, bufferValues); case symReg: return symRegularity(xTrain, xTest, yTrain, yTest, bufferValues); case stab: return stability(xTrain, xTest, yTrain, yTest, bufferValues); case symStab: return symStability(xTrain, xTest, yTrain, yTest, bufferValues); case unbiasedOut: return unbiasedOutputs(xTrain, xTest, yTrain, yTest, bufferValues); case symUnbiasedOut: return symUnbiasedOutputs(xTrain, xTest, yTrain, yTest, bufferValues); case unbiasedCoef: return unbiasedCoeffs(xTrain, xTest, yTrain, yTest, bufferValues); case absoluteNoiseImmun: return absoluteNoiseImmunity(xTrain, xTest, yTrain, yTest, bufferValues); case symAbsoluteNoiseImmun: return symAbsoluteNoiseImmunity(xTrain, xTest, yTrain, yTest, bufferValues); } PairDVXd pd; return pd; } /** Calculate the regularity external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param bufferValues Temporary storage for calculated coefficients and target values param inverseSplit True, if it is necessary to swap the roles of training and testing data, otherwise false return The value of the regularity external criterion and calculated model coefficients */ PairDVXd regularity(matrix& xTrain, matrix& xTest, vector &yTrain, vector& yTest, BufferValues& bufferValues, bool inverseSplit = false) { PairDVXd pdv; vector f; if(!inverseSplit) { if(bufferValues.coeffsTrain.Size() == 0) bufferValues.coeffsTrain = findBestCoeffs(xTrain, yTrain); if(bufferValues.yPredTestByTrain.Size() == 0) bufferValues.yPredTestByTrain = xTest.MatMul(bufferValues.coeffsTrain); f = MathPow((yTest - bufferValues.yPredTestByTrain),2.0); pdv.first = f.Sum(); pdv.second = bufferValues.coeffsTrain; } else { if(bufferValues.coeffsTest.Size() == 0) bufferValues.coeffsTest = findBestCoeffs(xTest, yTest); if(bufferValues.yPredTrainByTest.Size() == 0) bufferValues.yPredTrainByTest = xTrain.MatMul(bufferValues.coeffsTest); f = MathPow((yTrain - bufferValues.yPredTrainByTest),2.0); pdv.first = f.Sum(); pdv.second = bufferValues.coeffsTest; } return pdv; } /** Calculate the symmetric regularity external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param bufferValues Temporary storage for calculated coefficients and target values return The value of the symmertic regularity external criterion and calculated model coefficients */ PairDVXd symRegularity(matrix& xTrain, matrix& xTest, vector& yTrain, vector& yTest, BufferValues& bufferValues) { PairDVXd pdv1,pdv2,pdsum; pdv1 = regularity(xTrain,xTest,yTrain,yTest,bufferValues); pdv2 = regularity(xTrain,xTest,yTrain,yTest,bufferValues,true); pdsum.first = pdv1.first+pdv2.first; pdsum.second = pdv1.second; return pdsum; } /** Calculate the stability external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param bufferValues Temporary storage for calculated coefficients and target values param inverseSplit True, if it is necessary to swap the roles of training and testing data, otherwise false return The value of the stability external criterion and calculated model coefficients */ PairDVXd stability(matrix& xTrain, matrix& xTest, vector& yTrain, vector& yTest, BufferValues& bufferValues, bool inverseSplit = false) { PairDVXd pdv; vector f1,f2; if(!inverseSplit) { if(bufferValues.coeffsTrain.Size() == 0) bufferValues.coeffsTrain = findBestCoeffs(xTrain, yTrain); if(bufferValues.yPredTrainByTrain.Size() == 0) bufferValues.yPredTrainByTrain = xTrain.MatMul(bufferValues.coeffsTrain); if(bufferValues.yPredTestByTrain.Size() == 0) bufferValues.yPredTestByTrain = xTest.MatMul(bufferValues.coeffsTrain); f1 = MathPow((yTrain - bufferValues.yPredTrainByTrain),2.0); f2 = MathPow((yTest - bufferValues.yPredTestByTrain),2.0); pdv.first = f1.Sum()+f2.Sum(); pdv.second = bufferValues.coeffsTrain; } else { if(bufferValues.coeffsTest.Size() == 0) bufferValues.coeffsTest = findBestCoeffs(xTest, yTest); if(bufferValues.yPredTrainByTest.Size() == 0) bufferValues.yPredTrainByTest = xTrain.MatMul(bufferValues.coeffsTest); if(bufferValues.yPredTestByTest.Size() == 0) bufferValues.yPredTestByTest = xTest.MatMul(bufferValues.coeffsTest); f1 = MathPow((yTrain - bufferValues.yPredTrainByTest),2.0); f2 = MathPow((yTest - bufferValues.yPredTestByTest),2.0); pdv.first = f1.Sum() + f2.Sum(); pdv.second = bufferValues.coeffsTest; } return pdv; } /** Calculate the symmetric stability external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param bufferValues Temporary storage for calculated coefficients and target values return The value of the symmertic stability external criterion and calculated model coefficients */ PairDVXd symStability(matrix& xTrain, matrix& xTest, vector& yTrain, vector& yTest, BufferValues& bufferValues) { PairDVXd pdv1,pdv2,pdsum; pdv1 = stability(xTrain, xTest, yTrain, yTest, bufferValues); pdv2 = stability(xTrain, xTest, yTrain, yTest, bufferValues, true); pdsum.first=pdv1.first+pdv2.first; pdsum.second = pdv1.second; return pdsum; } /** Calculate the unbiased outputs external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param bufferValues Temporary storage for calculated coefficients and target values return The value of the unbiased outputs external criterion and calculated model coefficients */ PairDVXd unbiasedOutputs(matrix& xTrain, matrix& xTest, vector& yTrain, vector& yTest, BufferValues& bufferValues) { PairDVXd pdv; vector f; if(bufferValues.coeffsTrain.Size() == 0) bufferValues.coeffsTrain = findBestCoeffs(xTrain, yTrain); if(bufferValues.coeffsTest.Size() == 0) bufferValues.coeffsTest = findBestCoeffs(xTest, yTest); if(bufferValues.yPredTestByTrain.Size() == 0) bufferValues.yPredTestByTrain = xTest.MatMul(bufferValues.coeffsTrain); if(bufferValues.yPredTestByTest.Size() == 0) bufferValues.yPredTestByTest = xTest.MatMul(bufferValues.coeffsTest); f = MathPow((bufferValues.yPredTestByTrain - bufferValues.yPredTestByTest),2.0); pdv.first = f.Sum(); pdv.second = bufferValues.coeffsTrain; return pdv; } /** Calculate the symmetric unbiased outputs external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param bufferValues Temporary storage for calculated coefficients and target values return The value of the symmetric unbiased outputs external criterion and calculated model coefficients */ PairDVXd symUnbiasedOutputs(matrix &xTrain, matrix &xTest, vector &yTrain, vector& yTest,BufferValues& bufferValues) { PairDVXd pdv; vector f1,f2; if(bufferValues.coeffsTrain.Size() == 0) bufferValues.coeffsTrain = findBestCoeffs(xTrain, yTrain); if(bufferValues.coeffsTest.Size() == 0) bufferValues.coeffsTest = findBestCoeffs(xTest, yTest); if(bufferValues.yPredTrainByTrain.Size() == 0) bufferValues.yPredTrainByTrain = xTrain.MatMul(bufferValues.coeffsTrain); if(bufferValues.yPredTrainByTest.Size() == 0) bufferValues.yPredTrainByTest = xTrain.MatMul(bufferValues.coeffsTest); if(bufferValues.yPredTestByTrain.Size() == 0) bufferValues.yPredTestByTrain = xTest.MatMul(bufferValues.coeffsTrain); if(bufferValues.yPredTestByTest.Size() == 0) bufferValues.yPredTestByTest = xTest.MatMul(bufferValues.coeffsTest); f1 = MathPow((bufferValues.yPredTrainByTrain - bufferValues.yPredTrainByTest),2.0); f2 = MathPow((bufferValues.yPredTestByTrain - bufferValues.yPredTestByTest),2.0); pdv.first = f1.Sum() + f2.Sum(); pdv.second = bufferValues.coeffsTrain; return pdv; } /** Calculate the unbiased coefficients external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param bufferValues Temporary storage for calculated coefficients and target values return The value of the unbiased coefficients external criterion and calculated model coefficients */ PairDVXd unbiasedCoeffs(matrix& xTrain, matrix& xTest, vector& yTrain, vector& yTest,BufferValues& bufferValues) { PairDVXd pdv; vector f1; if(bufferValues.coeffsTrain.Size() == 0) bufferValues.coeffsTrain = findBestCoeffs(xTrain, yTrain); if(bufferValues.coeffsTest.Size() == 0) bufferValues.coeffsTest = findBestCoeffs(xTest, yTest); f1 = MathPow((bufferValues.coeffsTrain - bufferValues.coeffsTest),2.0); pdv.first = f1.Sum(); pdv.second = bufferValues.coeffsTrain; return pdv; } /** Calculate the absolute noise immunity external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param bufferValues Temporary storage for calculated coefficients and target values return The value of the absolute noise immunity external criterion and calculated model coefficients */ PairDVXd absoluteNoiseImmunity(matrix& xTrain, matrix& xTest, vector& yTrain, vector& yTest,BufferValues& bufferValues) { vector yPredTestByAll,f1,f2; PairDVXd pdv; if(bufferValues.coeffsTrain.Size() == 0) bufferValues.coeffsTrain = findBestCoeffs(xTrain, yTrain); if(bufferValues.coeffsTest.Size() == 0) bufferValues.coeffsTest = findBestCoeffs(xTest, yTest); if(bufferValues.coeffsAll.Size() == 0) { matrix dataX(xTrain.Rows() + xTest.Rows(), xTrain.Cols()); for(ulong i = 0; i<xTrain.Rows(); i++) dataX.Row(xTrain.Row(i),i); for(ulong i = 0; i<xTest.Rows(); i++) dataX.Row(xTest.Row(i),i+xTrain.Rows()); vector dataY(yTrain.Size() + yTest.Size()); for(ulong i=0; i<yTrain.Size(); i++) dataY[i] = yTrain[i]; for(ulong i=0; i<yTest.Size(); i++) dataY[i+yTrain.Size()] = yTest[i]; bufferValues.coeffsAll = findBestCoeffs(dataX, dataY); } if(bufferValues.yPredTestByTrain.Size() == 0) bufferValues.yPredTestByTrain = xTest.MatMul(bufferValues.coeffsTrain); if(bufferValues.yPredTestByTest.Size() == 0) bufferValues.yPredTestByTest = xTest.MatMul(bufferValues.coeffsTest); yPredTestByAll = xTest.MatMul(bufferValues.coeffsAll); f1 = yPredTestByAll - bufferValues.yPredTestByTrain; f2 = bufferValues.yPredTestByTest - yPredTestByAll; pdv.first = f1.Dot(f2); pdv.second = bufferValues.coeffsTrain; return pdv; } /** Calculate the symmetric absolute noise immunity external criterion for the given data param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data param bufferValues Temporary storage for calculated coefficients and target values return The value of the symmetric absolute noise immunity external criterion and calculated model coefficients */ PairDVXd symAbsoluteNoiseImmunity(matrix& xTrain, matrix& xTest, vector& yTrain, vector& yTest,BufferValues& bufferValues) { PairDVXd pdv; vector yPredAllByTrain, yPredAllByTest, yPredAllByAll,f1,f2; matrix dataX(xTrain.Rows() + xTest.Rows(), xTrain.Cols()); for(ulong i = 0; i<xTrain.Rows(); i++) dataX.Row(xTrain.Row(i),i); for(ulong i = 0; i<xTest.Rows(); i++) dataX.Row(xTest.Row(i),i+xTrain.Rows()); vector dataY(yTrain.Size() + yTest.Size()); for(ulong i=0; i<yTrain.Size(); i++) dataY[i] = yTrain[i]; for(ulong i=0; i<yTest.Size(); i++) dataY[i+yTrain.Size()] = yTest[i]; if(bufferValues.coeffsTrain.Size() == 0) bufferValues.coeffsTrain = findBestCoeffs(xTrain, yTrain); if(bufferValues.coeffsTest.Size() == 0) bufferValues.coeffsTest = findBestCoeffs(xTest, yTest); if(bufferValues.coeffsAll.Size() == 0) bufferValues.coeffsAll = findBestCoeffs(dataX, dataY); yPredAllByTrain = dataX.MatMul(bufferValues.coeffsTrain); yPredAllByTest = dataX.MatMul(bufferValues.coeffsTest); yPredAllByAll = dataX.MatMul(bufferValues.coeffsAll); f1 = yPredAllByAll - yPredAllByTrain; f2 = yPredAllByTest - yPredAllByAll; pdv.first = f1.Dot(f2); pdv.second = bufferValues.coeffsTrain; return pdv; } /** Get k models from the given ones with the best values of the external criterion param combinations Vector of the trained models param data Object containing parts of a split dataset used in model training. Parameter is used in sequential criterion param func Function returning the new X train and X test data constructed from the original data using given combination of input variables column indexes. Parameter is used in sequential criterion param k Number of best models return Vector containing k best models */ virtual void getBestCombinations(CVector &combinations, CVector &bestCombo,SplittedData& data, MatFunc func, int k) { double proxys[]; int best[]; ArrayResize(best,combinations.size()); ArrayResize(proxys,combinations.size()); for(int i = 0 ; i<combinations.size(); i++) { proxys[i] = combinations[i].evaluation(); best[i] = i; } MathQuickSortAscending(proxys,best,0,combinations.size()-1); for(int i = 0; i<int(MathMin(MathAbs(k),combinations.size())); i++) bestCombo.push_back(combinations[best[i]]); } /** Calculate the value of the selected external criterion for the given data. For the individual criterion this method only calls the getResult() method param xTrain Input variables matrix of the training data param xTest Input variables matrix of the testing data param yTrain Target values vector of the training data param yTest Target values vector of the testing data return The value of the external criterion and calculated model coefficients */ virtual PairDVXd calculate(matrix& xTrain, matrix& xTest, vector& yTrain, vector& yTest) { BufferValues tempValues; return getResult(xTrain, xTest, yTrain, yTest, criterionType, tempValues); } public: /// Construct a new Criterion object Criterion() {}; /** Construct a new Criterion object param _criterionType Selected external criterion type param _solver Selected method for linear equations solving */ Criterion(CriterionType _criterionType) { criterionType = _criterionType; solver = balanced; } };
Por último, tenemos dos funciones que marcan el final de gmdh_internal.mqh:
validateInputData() - se utiliza para garantizar que los valores pasados a los métodos de clase u otras funciones independientes se especifican correctamente.
** * Validate input parameters values * * param testSize Fraction of the input data that should be placed into the second part * param pAverage The number of best models based of which the external criterion for each level will be calculated * param threads The number of threads used for calculations. Set -1 to use max possible threads * param verbose 1 if the printing detailed infomation about training process is needed, otherwise 0 * param limit The minimum value by which the external criterion should be improved in order to continue training * param kBest The number of best models based of which new models of the next level will be constructed * return Method exit status */ int validateInputData(double testSize=0.0, int pAverage=0, double limit=0.0, int kBest=0) { int errorCode = 0; // if(testSize <= 0 || testSize >= 1) { Print("testsize value must be in the (0, 1) range"); errorCode |= 1; } if(pAverage && pAverage < 1) { Print("p_average value must be a positive integer"); errorCode |= 4; } if(limit && limit < 0) { Print("limit value must be non-negative"); errorCode |= 8; } if(kBest && kBest < 1) { Print("k_best value must be a positive integer"); errorCode |= 16; } return errorCode; }
timeSeriesTransformation() - Es una función de utilidad que toma como entrada una serie en un vector y la transforma en una estructura de datos de entradas y objetivos según el número de retardos elegido.
/** * Convert the time series vector to the 2D matrix format required to work with GMDH algorithms * * param timeSeries Vector of time series data * param lags The lags (length) of subsets of time series into which the original time series should be divided * return Transformed time series data */ PairMVXd timeSeriesTransformation(vector& timeSeries, int lags) { PairMVXd p; string errorMsg = ""; if(timeSeries.Size() == 0) errorMsg = "time_series value is empty"; else if(lags <= 0) errorMsg = "lags value must be a positive integer"; else if(lags >= int(timeSeries.Size())) errorMsg = "lags value can't be greater than time_series size"; if(errorMsg != "") return p; ulong last = timeSeries.Size() - ulong(lags); vector yTimeSeries(last,slice,timeSeries,ulong(lags)); matrix xTimeSeries(last, ulong(lags)); vector vect; for(ulong i = 0; i < last; ++i) { vect.Init(ulong(lags),slice,timeSeries,i,i+ulong(lags-1)); xTimeSeries.Row(vect,i); } p.first = xTimeSeries; p.second = yTimeSeries; return p; }
Aquí, "lags" se refiere al número de valores anteriores de la serie utilizados como predictores para calcular un término subsecuente.
Esto completa la descripción de gmdh_internal.mqh. Pasamos al segundo archivo de cabecera, gmdh.mqh.
Comienza con la definición de la función splitData().
/** * Divide the input data into 2 parts * * param x Matrix of input data containing predictive variables * param y Vector of the taget values for the corresponding x data * param testSize Fraction of the input data that should be placed into the second part * param shuffle True if data should be shuffled before splitting into 2 parts, otherwise false * param randomSeed Seed number for the random generator to get the same division every time * return SplittedData object containing 4 elements of data: train x, train y, test x, test y */ SplittedData splitData(matrix& x, vector& y, double testSize = 0.2, bool shuffle = false, int randomSeed = 0) { SplittedData data; if(validateInputData(testSize)) return data; string errorMsg = ""; if(x.Rows() != y.Size()) errorMsg = " x rows number and y size must be equal"; else if(round(x.Rows() * testSize) == 0 || round(x.Rows() * testSize) == x.Rows()) errorMsg = "Result contains an empty array. Change the arrays size or the value for correct splitting"; if(errorMsg != "") { Print(__FUNCTION__," ",errorMsg); return data; } if(!shuffle) data = GmdhModel::internalSplitData(x, y, testSize); else { if(randomSeed == 0) randomSeed = int(GetTickCount64()); MathSrand(uint(randomSeed)); int shuffled_rows_indexes[],shuffled[]; MathSequence(0,int(x.Rows()-1),1,shuffled_rows_indexes); MathSample(shuffled_rows_indexes,int(shuffled_rows_indexes.Size()),shuffled); int testItemsNumber = (int)round(x.Rows() * testSize); matrix Train,Test; vector train,test; Train.Resize(x.Rows()-ulong(testItemsNumber),x.Cols()); Test.Resize(ulong(testItemsNumber),x.Cols()); train.Resize(x.Rows()-ulong(testItemsNumber)); test.Resize(ulong(testItemsNumber)); for(ulong i = 0; i<Train.Rows(); i++) { Train.Row(x.Row(shuffled[i]),i); train[i] = y[shuffled[i]]; } for(ulong i = 0; i<Test.Rows(); i++) { Test.Row(x.Row(shuffled[Train.Rows()+i]),i); test[i] = y[shuffled[Train.Rows()+i]]; } data.xTrain = Train; data.xTest = Test; data.yTrain = train; data.yTest = test; } return data; }
Toma como entrada una matriz y un vector que representan variables y objetivos respectivamente. El parámetro "testSize" define la fracción del conjunto de datos que se utilizará como conjunto de prueba. "shuffle" permite la mezcla aleatoria del conjunto de datos y "randomSeed" especifica la semilla para un generador de números aleatorios utilizado en el proceso de mezcla.
A continuación tenemos la clase "GmdhModel", que define la lógica general de los algoritmos GMDH.
//+------------------------------------------------------------------+ //| Class implementing the general logic of GMDH algorithms | //+------------------------------------------------------------------+ class GmdhModel { protected: string modelName; // model name int level; // Current number of the algorithm training level int inputColsNumber; // The number of predictive variables in the original data double lastLevelEvaluation; // The external criterion value of the previous training level double currentLevelEvaluation; // The external criterion value of the current training level bool training_complete; // flag indicator successful completion of model training CVector2d bestCombinations; // Storage for the best models of previous levels /** *struct for generating vector sequence */ struct unique { private: int current; int run(void) { return ++current; } public: unique(void) { current = -1; } vector generate(ulong t) { ulong s=0; vector ret(t); while(s<t) ret[s++] = run(); return ret; } }; /** * Find all combinations of k elements from n * * param n Number of all elements * param k Number of required elements * return Vector of all combinations of k elements from n */ void nChooseK(int n, int k, vector &combos[]) { if(n<=0 || k<=0 || n<k) { Print(__FUNCTION__," invalid parameters for n and or k", "n ",n , " k ", k); return; } unique q; vector comb = q.generate(ulong(k)); ArrayResize(combos,combos.Size()+1,100); long first, last; first = 0; last = long(k); combos[combos.Size()-1]=comb; while(comb[first]!= double(n - k)) { long mt = last; while(comb[--mt] == double(n - (last - mt))); comb[mt]++; while(++mt != last) comb[mt] = comb[mt-1]+double(1); ArrayResize(combos,combos.Size()+1,100); combos[combos.Size()-1]=comb; } for(uint i = 0; i<combos.Size(); i++) { combos[i].Resize(combos[i].Size()+1); combos[i][combos[i].Size()-1] = n; } return; } /** * Get the mean value of extrnal criterion of the k best models * * param sortedCombinations Sorted vector of current level models * param k The numebr of the best models * return Calculated mean value of extrnal criterion of the k best models */ double getMeanCriterionValue(CVector &sortedCombinations, int k) { k = MathMin(k, sortedCombinations.size()); double crreval=0; for(int i = 0; i<k; i++) crreval +=sortedCombinations[i].evaluation(); if(k) return crreval/double(k); else { Print(__FUNCTION__, " Zero divide error "); return 0.0; } } /** * Get the sign of the polynomial variable coefficient * * param coeff Selected coefficient * param isFirstCoeff True if the selected coefficient will be the first in the polynomial representation, otherwise false * return String containing the sign of the coefficient */ string getPolynomialCoeffSign(double coeff, bool isFirstCoeff) { return ((coeff >= 0) ? ((isFirstCoeff) ? " " : " + ") : " - "); } /** * Get the rounded value of the polynomial variable coefficient without sign * * param coeff Selected coefficient * param isLastCoeff True if the selected coefficient will be the last one in the polynomial representation, otherwise false * return String containing the rounded value of the coefficient without sign */ string getPolynomialCoeffValue(double coeff, bool isLastCoeff) { string stringCoeff = StringFormat("%e",MathAbs(coeff)); return ((stringCoeff != "1" || isLastCoeff) ? stringCoeff : ""); } /** * Train given subset of models and calculate external criterion for them * * param data Data used for training and evaulating models * param criterion Selected external criterion * param beginCoeffsVec Iterator indicating the beginning of a subset of models * param endCoeffsVec Iterator indicating the end of a subset of models * param leftTasks The number of remaining untrained models at the entire level * param verbose 1 if the printing detailed infomation about training process is needed, otherwise 0 */ bool polynomialsEvaluation(SplittedData& data, Criterion& criterion, CVector &combos, uint beginCoeffsVec, uint endCoeffsVec) { vector cmb,ytrain,ytest; matrix x1,x2; for(uint i = beginCoeffsVec; i<endCoeffsVec; i++) { cmb = combos[i].combination(); x1 = xDataForCombination(data.xTrain,cmb); x2 = xDataForCombination(data.xTest,cmb); ytrain = data.yTrain; ytest = data.yTest; PairDVXd pd = criterion.calculate(x1,x2,ytrain,ytest); if(pd.second.HasNan()>0) { Print(__FUNCTION__," No solution found for coefficient at ", i, "\n xTrain \n", x1, "\n xTest \n", x2, "\n yTrain \n", ytrain, "\n yTest \n", ytest); combos[i].setEvaluation(DBL_MAX); combos[i].setBestCoeffs(vector::Ones(3)); } else { combos[i].setEvaluation(pd.first); combos[i].setBestCoeffs(pd.second); } } return true; } /** * Determine the need to continue training and prepare the algorithm for the next level * * param kBest The number of best models based of which new models of the next level will be constructed * param pAverage The number of best models based of which the external criterion for each level will be calculated * param combinations Trained models of the current level * param criterion Selected external criterion * param data Data used for training and evaulating models * param limit The minimum value by which the external criterion should be improved in order to continue training * return True if the algorithm needs to continue training, otherwise fasle */ bool nextLevelCondition(int kBest, int pAverage, CVector &combinations, Criterion& criterion, SplittedData& data, double limit) { MatFunc fun = NULL; CVector bestcombinations; criterion.getBestCombinations(combinations,bestcombinations,data, fun, kBest); currentLevelEvaluation = getMeanCriterionValue(bestcombinations, pAverage); if(lastLevelEvaluation - currentLevelEvaluation > limit) { lastLevelEvaluation = currentLevelEvaluation; if(preparations(data,bestcombinations)) { ++level; return true; } } removeExtraCombinations(); return false; } /** * Fit the algorithm to find the best solution * * param x Matrix of input data containing predictive variables * param y Vector of the taget values for the corresponding x data * param criterion Selected external criterion * param kBest The number of best models based of which new models of the next level will be constructed * param testSize Fraction of the input data that should be used to evaluate models at each level * param pAverage The number of best models based of which the external criterion for each level will be calculated * param limit The minimum value by which the external criterion should be improved in order to continue training * return A pointer to the algorithm object for which the training was performed */ bool gmdhFit(matrix& x, vector& y, Criterion& criterion, int kBest, double testSize, int pAverage, double limit) { if(x.Rows() != y.Size()) { Print("X rows number and y size must be equal"); return false; } level = 1; // reset last training inputColsNumber = int(x.Cols()); lastLevelEvaluation = DBL_MAX; SplittedData data = internalSplitData(x, y, testSize, true) ; training_complete = false; bool goToTheNextLevel; CVector evaluationCoeffsVec; do { vector combinations[]; generateCombinations(int(data.xTrain.Cols() - 1),combinations); if(combinations.Size()<1) { Print(__FUNCTION__," Training aborted"); return training_complete; } evaluationCoeffsVec.clear(); int currLevelEvaluation = 0; for(int it = 0; it < int(combinations.Size()); ++it, ++currLevelEvaluation) { Combination ncomb(combinations[it]); evaluationCoeffsVec.push_back(ncomb); } if(!polynomialsEvaluation(data,criterion,evaluationCoeffsVec,0,uint(currLevelEvaluation))) { Print(__FUNCTION__," Training aborted"); return training_complete; } goToTheNextLevel = nextLevelCondition(kBest, pAverage, evaluationCoeffsVec, criterion, data, limit); // checking the results of the current level for improvement } while(goToTheNextLevel); training_complete = true; return true; } /** * Get new model structures for the new level of training * * param n_cols The number of existing predictive variables at the current training level * return Vector of new model structures */ virtual void generateCombinations(int n_cols,vector &out[]) { return; } /// Removed the saved models that are no longer needed virtual void removeExtraCombinations(void) { return; } /** * Prepare data for the next training level * * param data Data used for training and evaulating models at the current level * param _bestCombinations Vector of the k best models of the current level * return True if the training process can be continued, otherwise false */ virtual bool preparations(SplittedData& data, CVector &_bestCombinations) { return false; } /** * Get the data constructed according to the model structure from the original data * * param x Training data at the current level * param comb Vector containing the indexes of the x matrix columns that should be used in the model * return Constructed data */ virtual matrix xDataForCombination(matrix& x, vector& comb) { return matrix::Zeros(10,10); } /** * Get the designation of polynomial equation * * param levelIndex The number of the level counting from 0 * param combIndex The number of polynomial in the level counting from 0 * return The designation of polynomial equation */ virtual string getPolynomialPrefix(int levelIndex, int combIndex) { return NULL; } /** * Get the string representation of the polynomial variable * * param levelIndex The number of the level counting from 0 * param coeffIndex The number of the coefficient related to the selected variable in the polynomial counting from 0 * param coeffsNumber The number of coefficients in the polynomial * param bestColsIndexes Indexes of the data columns used to construct polynomial of the model * return The string representation of the polynomial variable */ virtual string getPolynomialVariable(int levelIndex, int coeffIndex, int coeffsNumber, vector& bestColsIndexes) { return NULL; } /* * Transform model data to JSON format for further saving * * return JSON value of model data */ virtual CJAVal toJSON(void) { CJAVal json_obj_model; json_obj_model["modelName"] = getModelName(); json_obj_model["inputColsNumber"] = inputColsNumber; json_obj_model["bestCombinations"] = CJAVal(jtARRAY,""); for(int i = 0; i<bestCombinations.size(); i++) { CJAVal Array(jtARRAY,""); for(int k = 0; k<bestCombinations[i].size(); k++) { CJAVal collection; collection["combination"] = CJAVal(jtARRAY,""); collection["bestCoeffs"] = CJAVal(jtARRAY,""); vector combination = bestCombinations[i][k].combination(); vector bestcoeff = bestCombinations[i][k].bestCoeffs(); for(ulong j=0; j<combination.Size(); j++) collection["combination"].Add(int(combination[j])); for(ulong j=0; j<bestcoeff.Size(); j++) collection["bestCoeffs"].Add(bestcoeff[j],-15); Array.Add(collection); } json_obj_model["bestCombinations"].Add(Array); } return json_obj_model; } /** * Set up model from JSON format model data * * param jsonModel Model data in JSON format * return Method exit status */ virtual bool fromJSON(CJAVal &jsonModel) { modelName = jsonModel["modelName"].ToStr(); bestCombinations.clear(); inputColsNumber = int(jsonModel["inputColsNumber"].ToInt()); for(int i = 0; i<jsonModel["bestCombinations"].Size(); i++) { CVector member; for(int j = 0; j<jsonModel["bestCombinations"][i].Size(); j++) { Combination cb; vector c(ulong(jsonModel["bestCombinations"][i][j]["combination"].Size())); vector cf(ulong(jsonModel["bestCombinations"][i][j]["bestCoeffs"].Size())); for(int k = 0; k<jsonModel["bestCombinations"][i][j]["combination"].Size(); k++) c[k] = jsonModel["bestCombinations"][i][j]["combination"][k].ToDbl(); for(int k = 0; k<jsonModel["bestCombinations"][i][j]["bestCoeffs"].Size(); k++) cf[k] = jsonModel["bestCombinations"][i][j]["bestCoeffs"][k].ToDbl(); cb.setBestCoeffs(cf); cb.setCombination(c); member.push_back(cb); } bestCombinations.push_back(member); } return true; } /** * Compare the number of required and actual columns of the input matrix * * param x Given matrix of input data */ bool checkMatrixColsNumber(matrix& x) { if(ulong(inputColsNumber) != x.Cols()) { Print("Matrix must have " + string(inputColsNumber) + " columns because there were " + string(inputColsNumber) + " columns in the training matrix"); return false; } return true; } public: /// Construct a new Gmdh Model object GmdhModel() : level(1), lastLevelEvaluation(0) {} /** * Get full class name * * return String containing the name of the model class */ string getModelName(void) { return modelName; } /** *Get number of inputs required for model */ int getNumInputs(void) { return inputColsNumber; } /** * Save model data into regular file * * param path Path to regular file */ bool save(string file_name) { CFileTxt modelFile; if(modelFile.Open(file_name,FILE_WRITE|FILE_COMMON,0)==INVALID_HANDLE) { Print("failed to open file ",file_name," .Error - ",::GetLastError()); return false; } else { CJAVal js=toJSON(); if(modelFile.WriteString(js.Serialize())==0) { Print("failed write to ",file_name,". Error -",::GetLastError()); return false; } } return true; } /** * Load model data from regular file * * param path Path to regular file */ bool load(string file_name) { training_complete = false; CFileTxt modelFile; CJAVal js; if(modelFile.Open(file_name,FILE_READ|FILE_COMMON,0)==INVALID_HANDLE) { Print("failed to open file ",file_name," .Error - ",::GetLastError()); return false; } else { if(!js.Deserialize(modelFile.ReadString())) { Print("failed to read from ",file_name,".Error -",::GetLastError()); return false; } training_complete = fromJSON(js); } return training_complete; } /** * Divide the input data into 2 parts without shuffling * * param x Matrix of input data containing predictive variables * param y Vector of the taget values for the corresponding x data * param testSize Fraction of the input data that should be placed into the second part * param addOnesCol True if it is needed to add a column of ones to the x data, otherwise false * return SplittedData object containing 4 elements of data: train x, train y, test x, test y */ static SplittedData internalSplitData(matrix& x, vector& y, double testSize, bool addOnesCol = false) { SplittedData data; ulong testItemsNumber = ulong(round(double(x.Rows()) * testSize)); matrix Train,Test; vector train,test; if(addOnesCol) { Train.Resize(x.Rows() - testItemsNumber, x.Cols() + 1); Test.Resize(testItemsNumber, x.Cols() + 1); for(ulong i = 0; i<Train.Rows(); i++) Train.Row(x.Row(i),i); Train.Col(vector::Ones(Train.Rows()),x.Cols()); for(ulong i = 0; i<Test.Rows(); i++) Test.Row(x.Row(Train.Rows()+i),i); Test.Col(vector::Ones(Test.Rows()),x.Cols()); } else { Train.Resize(x.Rows() - testItemsNumber, x.Cols()); Test.Resize(testItemsNumber, x.Cols()); for(ulong i = 0; i<Train.Rows(); i++) Train.Row(x.Row(i),i); for(ulong i = 0; i<Test.Rows(); i++) Test.Row(x.Row(Train.Rows()+i),i); } train.Init(y.Size() - testItemsNumber,slice,y,0,y.Size() - testItemsNumber - 1); test.Init(testItemsNumber,slice,y,y.Size() - testItemsNumber); data.yTrain = train; data.yTest = test; data.xTrain = Train; data.xTest = Test; return data; } /** * Get long-term forecast for the time series * * param x One row of the test time series data * param lags The number of lags (steps) to make a forecast for * return Vector containing long-term forecast */ virtual vector predict(vector& x, int lags) { return vector::Zeros(1); } /** * Get the String representation of the best polynomial * * return String representation of the best polynomial */ string getBestPolynomial(void) { string polynomialStr = ""; int ind = 0; for(int i = 0; i < bestCombinations.size(); ++i) { for(int j = 0; j < bestCombinations[i].size(); ++j) { vector bestColsIndexes = bestCombinations[i][j].combination(); vector bestCoeffs = bestCombinations[i][j].bestCoeffs(); polynomialStr += getPolynomialPrefix(i, j); bool isFirstCoeff = true; for(int k = 0; k < int(bestCoeffs.Size()); ++k) { if(bestCoeffs[k]) { polynomialStr += getPolynomialCoeffSign(bestCoeffs[k], isFirstCoeff); string coeffValuelStr = getPolynomialCoeffValue(bestCoeffs[k], (k == (bestCoeffs.Size() - 1))); polynomialStr += coeffValuelStr; if(coeffValuelStr != "" && k != bestCoeffs.Size() - 1) polynomialStr += "*"; polynomialStr += getPolynomialVariable(i, k, int(bestCoeffs.Size()), bestColsIndexes); isFirstCoeff = false; } } if(i < bestCombinations.size() - 1 || j < (bestCombinations[i].size() - 1)) polynomialStr += "\n"; }//j if(i < bestCombinations.size() - 1 && bestCombinations[i].size() > 1) polynomialStr += "\n"; }//i return polynomialStr; } ~GmdhModel() { for(int i = 0; i<bestCombinations.size(); i++) bestCombinations[i].clear(); bestCombinations.clear(); } }; //+------------------------------------------------------------------+
Es la clase base de la que derivarán otros tipos GMDH. Proporciona métodos para entrenar o construir un modelo y posteriormente hacer predicciones con él. Los métodos "save" y "load" permiten guardar un modelo y cargarlo desde un archivo para su uso posterior. Los modelos se guardan en formato JSON en un archivo de texto en el directorio común a todos los terminales MetaTrader.
El último archivo de cabecera, mia.mqh contiene la definición de la clase "MIA".
//+------------------------------------------------------------------+ //| Class implementing multilayered iterative algorithm MIA | //+------------------------------------------------------------------+ class MIA : public GmdhModel { protected: PolynomialType polynomialType; // Selected polynomial type void generateCombinations(int n_cols,vector &out[]) override { GmdhModel::nChooseK(n_cols,2,out); return; } /** * Get predictions for the input data * * param x Test data of the regression task or one-step time series forecast * return Vector containing prediction values */ virtual vector calculatePrediction(vector& x) { if(x.Size()<ulong(inputColsNumber)) return vector::Zeros(ulong(inputColsNumber)); matrix modifiedX(1,x.Size()+ 1); modifiedX.Row(x,0); modifiedX[0][x.Size()] = 1.0; for(int i = 0; i < bestCombinations.size(); ++i) { matrix xNew(1, ulong(bestCombinations[i].size()) + 1); for(int j = 0; j < bestCombinations[i].size(); ++j) { vector comb = bestCombinations[i][j].combination(); matrix xx(1,comb.Size()); for(ulong i = 0; i<xx.Cols(); ++i) xx[0][i] = modifiedX[0][ulong(comb[i])]; matrix ply = getPolynomialX(xx); vector c,b; c = bestCombinations[i][j].bestCoeffs(); b = ply.MatMul(c); xNew.Col(b,ulong(j)); } vector n = vector::Ones(xNew.Rows()); xNew.Col(n,xNew.Cols() - 1); modifiedX = xNew; } return modifiedX.Col(0); } /** * Construct vector of the new variable values according to the selected polynomial type * * param x Matrix of input variables values for the selected polynomial type * return Construct vector of the new variable values */ matrix getPolynomialX(matrix& x) { matrix polyX = x; if((polynomialType == linear_cov)) { polyX.Resize(x.Rows(), 4); polyX.Col(x.Col(0)*x.Col(1),2); polyX.Col(x.Col(2),3); } else if((polynomialType == quadratic)) { polyX.Resize(x.Rows(), 6); polyX.Col(x.Col(0)*x.Col(1),2) ; polyX.Col(x.Col(0)*x.Col(0),3); polyX.Col(x.Col(1)*x.Col(1),4); polyX.Col(x.Col(2),5) ; } return polyX; } /** * Transform data in the current training level by constructing new variables using selected polynomial type * * param data Data used to train models at the current level * param bestCombinations Vector of the k best models of the current level */ virtual void transformDataForNextLevel(SplittedData& data, CVector &bestCombs) { matrix xTrainNew(data.xTrain.Rows(), ulong(bestCombs.size()) + 1); matrix xTestNew(data.xTest.Rows(), ulong(bestCombs.size()) + 1); for(int i = 0; i < bestCombs.size(); ++i) { vector comb = bestCombs[i].combination(); matrix train(xTrainNew.Rows(),comb.Size()),test(xTrainNew.Rows(),comb.Size()); for(ulong k = 0; k<comb.Size(); k++) { train.Col(data.xTrain.Col(ulong(comb[k])),k); test.Col(data.xTest.Col(ulong(comb[k])),k); } matrix polyTest,polyTrain; vector bcoeff = bestCombs[i].bestCoeffs(); polyTest = getPolynomialX(test); polyTrain = getPolynomialX(train); xTrainNew.Col(polyTrain.MatMul(bcoeff),i); xTestNew.Col(polyTest.MatMul(bcoeff),i); } xTrainNew.Col(vector::Ones(xTrainNew.Rows()),xTrainNew.Cols() - 1); xTestNew.Col(vector::Ones(xTestNew.Rows()),xTestNew.Cols() - 1); data.xTrain = xTrainNew; data.xTest = xTestNew; } virtual void removeExtraCombinations(void) override { CVector2d realBestCombinations(bestCombinations.size()); CVector n; n.push_back(bestCombinations[level-2][0]); realBestCombinations.setAt(realBestCombinations.size() - 1,n); vector comb(1); for(int i = realBestCombinations.size() - 1; i > 0; --i) { double usedCombinationsIndexes[],unique[]; int indexs[]; int prevsize = 0; for(int j = 0; j < realBestCombinations[i].size(); ++j) { comb = realBestCombinations[i][j].combination(); ArrayResize(usedCombinationsIndexes,prevsize+int(comb.Size()-1),100); for(ulong k = 0; k < comb.Size() - 1; ++k) usedCombinationsIndexes[ulong(prevsize)+k] = comb[k]; prevsize = int(usedCombinationsIndexes.Size()); } MathUnique(usedCombinationsIndexes,unique); ArraySort(unique); for(uint it = 0; it<unique.Size(); ++it) realBestCombinations[i - 1].push_back(bestCombinations[i - 1][int(unique[it])]); for(int j = 0; j < realBestCombinations[i].size(); ++j) { comb = realBestCombinations[i][j].combination(); for(ulong k = 0; k < comb.Size() - 1; ++k) comb[k] = ArrayBsearch(unique,comb[k]); comb[comb.Size() - 1] = double(unique.Size()); realBestCombinations[i][j].setCombination(comb); } ZeroMemory(usedCombinationsIndexes); ZeroMemory(unique); ZeroMemory(indexs); } bestCombinations = realBestCombinations; } virtual bool preparations(SplittedData& data, CVector &_bestCombinations) override { bestCombinations.push_back(_bestCombinations); transformDataForNextLevel(data, bestCombinations[level - 1]); return true; } virtual matrix xDataForCombination(matrix& x, vector& comb) override { matrix xx(x.Rows(),comb.Size()); for(ulong i = 0; i<xx.Cols(); ++i) xx.Col(x.Col(ulong(comb[i])),i); return getPolynomialX(xx); } string getPolynomialPrefix(int levelIndex, int combIndex) override { return ((levelIndex < bestCombinations.size() - 1) ? "f" + string(levelIndex + 1) + "_" + string(combIndex + 1) : "y") + " ="; } string getPolynomialVariable(int levelIndex, int coeffIndex, int coeffsNumber, vector &bestColsIndexes) override { if(levelIndex == 0) { if(coeffIndex < 2) return "x" + string(int(bestColsIndexes[coeffIndex]) + 1); else if(coeffIndex == 2 && coeffsNumber > 3) return "x" + string(int(bestColsIndexes[0]) + 1) + "*x" + string(int(bestColsIndexes[1]) + 1); else if(coeffIndex < 5 && coeffsNumber > 4) return "x" + string(int(bestColsIndexes[coeffIndex - 3]) + 1) + "^2"; } else { if(coeffIndex < 2) return "f" + string(levelIndex) + "_" + string(int(bestColsIndexes[coeffIndex]) + 1); else if(coeffIndex == 2 && coeffsNumber > 3) return "f" + string(levelIndex) + "_" + string(int(bestColsIndexes[0]) + 1) + "*f" + string(levelIndex) + "_" + string(int(bestColsIndexes[1]) + 1); else if(coeffIndex < 5 && coeffsNumber > 4) return "f" + string(levelIndex) + "_" + string(int(bestColsIndexes[coeffIndex - 3]) + 1) + "^2"; } return ""; } CJAVal toJSON(void) override { CJAVal json_obj_model = GmdhModel::toJSON(); json_obj_model["polynomialType"] = int(polynomialType); return json_obj_model; } bool fromJSON(CJAVal &jsonModel) override { bool parsed = GmdhModel::fromJSON(jsonModel); if(!parsed) return false; polynomialType = PolynomialType(jsonModel["polynomialType"].ToInt()); return true; } public: //+------------------------------------------------------------------+ //| Constructor | //+------------------------------------------------------------------+ MIA(void) { modelName = "MIA"; } //+------------------------------------------------------------------+ //| model a time series | //+------------------------------------------------------------------+ virtual bool fit(vector &time_series,int lags,double testsize=0.5,PolynomialType _polynomialType=linear_cov,CriterionType criterion=stab,int kBest = 10,int pAverage = 1,double limit = 0.0) { if(lags < 3) { Print(__FUNCTION__," lags must be >= 3"); return false; } PairMVXd transformed = timeSeriesTransformation(time_series,lags); SplittedData splited = splitData(transformed.first,transformed.second,testsize); Criterion criter(criterion); if(kBest < 3) { Print(__FUNCTION__," kBest value must be an integer >= 3"); return false; } if(validateInputData(testsize, pAverage, limit, kBest)) return false; polynomialType = _polynomialType; return GmdhModel::gmdhFit(splited.xTrain, splited.yTrain, criter, kBest, testsize, pAverage, limit); } //+------------------------------------------------------------------+ //| model a multivariable data set of inputs and targets | //+------------------------------------------------------------------+ virtual bool fit(matrix &vars,vector &targets,double testsize=0.5,PolynomialType _polynomialType=linear_cov,CriterionType criterion=stab,int kBest = 10,int pAverage = 1,double limit = 0.0) { if(vars.Cols() < 3) { Print(__FUNCTION__," columns in vars must be >= 3"); return false; } if(vars.Rows() != targets.Size()) { Print(__FUNCTION__, " vars dimensions donot correspond with targets"); return false; } SplittedData splited = splitData(vars,targets,testsize); Criterion criter(criterion); if(kBest < 3) { Print(__FUNCTION__," kBest value must be an integer >= 3"); return false; } if(validateInputData(testsize, pAverage, limit, kBest)) return false; polynomialType = _polynomialType; return GmdhModel::gmdhFit(splited.xTrain, splited.yTrain, criter, kBest, testsize, pAverage, limit); } virtual vector predict(vector& x, int lags) override { if(lags <= 0) { Print(__FUNCTION__," lags value must be a positive integer"); return vector::Zeros(1); } if(!training_complete) { Print(__FUNCTION__," model was not successfully trained"); return vector::Zeros(1); } vector expandedX = vector::Zeros(x.Size() + ulong(lags)); for(ulong i = 0; i<x.Size(); i++) expandedX[i]=x[i]; for(int i = 0; i < lags; ++i) { vector vect(x.Size(),slice,expandedX,ulong(i),x.Size()+ulong(i)-1); vector res = calculatePrediction(vect); expandedX[x.Size() + i] = res[0]; } vector vect(ulong(lags),slice,expandedX,x.Size()); return vect; } }; //+------------------------------------------------------------------+
Hereda de "GmdhModel" para implementar el algoritmo iterativo multicapa. "MIA" tiene dos sobrecargas de "fit()" que se pueden llamar para modelar un conjunto de datos dado. Estos métodos se distinguen por su primer y segundo parámetro. Cuando se desea modelar una serie temporal utilizando únicamente valores históricos, se utiliza la función "fit()" que se indica a continuación.
fit(vector &time_series,int lags,double testsize=0.5,PolynomialType _polynomialType=linear_cov,CriterionType criterion=stab,int kBest = 10,int pAverage = 1,double limit = 0.0)
Mientras que el otro es útil cuando se modela un conjunto de datos de variables dependientes e independientes. Los parámetros de ambos métodos se documentan en la siguiente tabla:
| Tipo de datos | Nombre del parámetro | Descripción |
|---|---|---|
| vector | time_series | Representa una serie temporal contenida en un vector. |
| integer | lags | Define el número de valores retardados que se utilizarán como predictores en el modelo. |
| matrix | vars | Matriz de datos de entrada que contiene variables predictivas. |
| vector | targets | Vector de los valores objetivo para los miembros de fila correspondientes de vars. |
| CriterionType | criterion | Variable de enumeración que especifica los criterios externos para el proceso de construcción del modelo. |
| integer | kBest | Define el número de los mejores modelos parciales a partir de los cuales se construirán las nuevas entradas de la capa siguiente. |
| PolynomialType | _polynomialType | Tipo de polinomio seleccionado que se utilizará para construir nuevas variables a partir de las existentes durante el entrenamiento. |
| double | testSize | Fracción de los datos de entrada que deben utilizarse para evaluar los modelos. |
| int | pAverage | El número de los mejores modelos parciales que se deben considerar en el cálculo de los criterios de detención. |
| double | limit | El valor mínimo por el cual el criterio externo debe mejorar para continuar con el entrenamiento. |
Una vez que se ha entrenado un modelo, puede utilizarse para hacer predicciones, llamando a "predict()". El método requiere un vector de entradas y un valor entero que especifique el número deseado de predicciones. Si se ejecuta correctamente, el método devuelve un vector que contiene las predicciones calculadas. En caso contrario, se devuelve un vector de ceros. En la sección siguiente veremos algunos ejemplos sencillos, para hacernos una mejor idea de cómo utilizar el código que acabamos de describir.
Ejemplos
Repasaremos tres ejemplos implementados como scripts. Cubriendo cómo se puede aplicar MIA en diferentes escenarios. El primero se ocupa de construir un modelo de una serie temporal. Donde un cierto número de valores anteriores de la serie pueden utilizarse para determinar los términos subsecuentes. Este ejemplo está contenido en el script MIA_Test.mq5, cuyo código se muestra a continuación.
//+------------------------------------------------------------------+ //| MIA_Test.mq5 | //| Copyright 2024, MetaQuotes Ltd. | //| https://www.mql5.com | //+------------------------------------------------------------------+ #property copyright "Copyright 2024, MetaQuotes Ltd." #property link "https://www.mql5.com" #property version "1.00" #property script_show_inputs #include <GMDH\mia.mqh> input int NumLags = 3; input int NumPredictions = 6; input CriterionType critType = stab; input PolynomialType polyType = linear_cov; input double DataSplitSize = 0.33; input int NumBest = 10; input int pAverge = 1; input double critLimit = 0; //+------------------------------------------------------------------+ //| Script program start function | //+------------------------------------------------------------------+ void OnStart() { //---time series we want to model vector tms = {1,2,3,4,5,6,7,8,9,10,11,12}; //--- if(NumPredictions<1) { Alert("Invalid setting for NumPredictions, has to be larger than 0"); return; } //---instantiate MIA object MIA mia; //---fit the series according to user defined hyper parameters if(!mia.fit(tms,NumLags,DataSplitSize,polyType,critType,NumBest,pAverge,critLimit)) return; //---generate filename based on user defined parameter settings string modelname = mia.getModelName()+"_"+EnumToString(critType)+"_"+string(DataSplitSize)+"_"+string(pAverge)+"_"+string(critLimit); //---save the trained model mia.save(modelname+".json"); //---inputs from original series to be used for making predictions vector in(ulong(NumLags),slice,tms,tms.Size()-ulong(NumLags)); //---predictions made from the model vector out = mia.predict(in,NumPredictions); //---output result of prediction Print(modelname, " predictions ", out); //---output the polynomial that defines the model Print(mia.getBestPolynomial()); } //+------------------------------------------------------------------+
Al ejecutar el script, el usuario puede cambiar varios aspectos del modelo. "NumLags" especifica el número de valores de la serie anterior para calcular el siguiente término. "NumPredictions" indica el número de predicciones que deben realizarse más allá de la serie especificada. El resto de parámetros ajustables por el usuario corresponden a los argumentos pasados al método "fit()". Cuando un modelo se ha construido correctamente, se guarda en un archivo. Y las predicciones se realizan y se envían a la pestaña Expertos del terminal, junto con el polinimio final que representa el modelo. A continuación se muestran los resultados de ejecutar el script con la configuración predeterminada. El polinomio mostrado representa el modelo matemático que mejor describe la serie temporal en cuestión. Es evidente que se complica innecesariamente si se tiene en cuenta la sencillez de la serie. Aunque, teniendo en cuenta los resultados de la predicción, el modelo sigue captando la tendencia general de la serie.
PS 0 22:37:31.246 MIA_Test (USDCHF,D1) MIA_stab_0.33_1_0.0 predictions [13.00000000000001,14.00000000000002,15.00000000000004,16.00000000000005,17.0000000000001,18.0000000000001] OG 0 22:37:31.246 MIA_Test (USDCHF,D1) y = - 9.340179e-01*x1 + 1.934018e+00*x2 + 3.865363e-16*x1*x2 + 1.065982e+00
En una segunda ejecución del script. NumLags se incrementa a 4. Veamos qué efecto tiene esto en el modelo.
Fíjese en la mayor complejidad que se introduce en el modelo al añadir un predictor adicional. Así como el impacto que esto tiene en las predicciones. El polinomio abarca ahora varias líneas, a pesar de que no hay ninguna mejora perceptible en las predicciones del modelo.
22:37:42.921 MIA_Test (USDCHF,D1) MIA_stab_0.33_1_0.0 predictions [13.00000000000001,14.00000000000002,15.00000000000005,16.00000000000007,17.00000000000011,18.00000000000015] ML 0 22:37:42.921 MIA_Test (USDCHF,D1) f1_1 = - 1.666667e-01*x2 + 1.166667e+00*x4 + 8.797938e-16*x2*x4 + 6.666667e-01 CO 0 22:37:42.921 MIA_Test (USDCHF,D1) f1_2 = - 6.916614e-15*x3 + 1.000000e+00*x4 + 1.006270e-15*x3*x4 + 1.000000e+00 NN 0 22:37:42.921 MIA_Test (USDCHF,D1) f1_3 = - 5.000000e-01*x1 + 1.500000e+00*x3 + 1.001110e-15*x1*x3 + 1.000000e+00 QR 0 22:37:42.921 MIA_Test (USDCHF,D1) f2_1 = 5.000000e-01*f1_1 + 5.000000e-01*f1_3 - 5.518760e-16*f1_1*f1_3 - 1.729874e-14 HR 0 22:37:42.921 MIA_Test (USDCHF,D1) f2_2 = 5.000000e-01*f1_1 + 5.000000e-01*f1_2 - 1.838023e-16*f1_1*f1_2 - 8.624525e-15 JK 0 22:37:42.921 MIA_Test (USDCHF,D1) y = 5.000000e-01*f2_1 + 5.000000e-01*f2_2 - 2.963544e-16*f2_1*f2_2 - 1.003117e-14
En nuestro último ejemplo, examinamos un escenario diferente, donde queremos modelar salidas definidas por variables independientes. En este ejemplo estamos intentando enseñar al modelo a sumar 3 entradas. El código de este ejemplo se encuentra en MIA_Multivariable_test.mq5.
//+------------------------------------------------------------------+ //| MIA_miavariable_test.mq5 | //| Copyright 2024, MetaQuotes Ltd. | //| https://www.mql5.com | //+------------------------------------------------------------------+ #property copyright "Copyright 2024, MetaQuotes Ltd." #property link "https://www.mql5.com" #property version "1.00" #property script_show_inputs #include <GMDH\mia.mqh> input CriterionType critType = stab; input PolynomialType polyType = linear_cov; input double DataSplitSize = 0.33; input int NumBest = 10; input int pAverge = 1; input double critLimit = 0; //+------------------------------------------------------------------+ //| Script program start function | //+------------------------------------------------------------------+ void OnStart() { //---simple independent and dependent data sets we want to model matrix independent = {{1,2,3},{3,2,1},{1,4,2},{1,1,3},{5,3,1},{3,1,9}}; vector dependent = {6,6,7,5,9,13}; //---declare MIA object MIA mia; //---train the model based on chosen hyper parameters if(!mia.fit(independent,dependent,DataSplitSize,polyType,critType,NumBest,pAverge,critLimit)) return; //---construct filename for generated model string modelname = mia.getModelName()+"_"+EnumToString(critType)+"_"+string(DataSplitSize)+"_"+string(pAverge)+"_"+string(critLimit)+"_multivars"; //---save the model mia.save(modelname+".json"); //---input data to be used as input for making predictions matrix unseen = {{1,2,4},{1,5,3},{9,1,3}}; //---make predictions and output to the terminal for(ulong row = 0; row<unseen.Rows(); row++) { vector in = unseen.Row(row); Print("inputs ", in , " prediction ", mia.predict(in,1)); } //---output the polynomial that defines the model Print(mia.getBestPolynomial()); } //+------------------------------------------------------------------+
Los predictores están en la matriz "vars". Cada fila corresponde a un objetivo del vector "objetivos". Como en el ejemplo anterior, tenemos la opción de configurar diversos aspectos de los hiperparámetros de entrenamiento del modelo. Los resultados del entrenamiento con la configuración por defecto son muy pobres, como se muestra a continuación.
RE 0 22:38:57.445 MIA_Multivariable_test (USDCHF,D1) inputs [1,2,4] prediction [5.999999999999997] JQ 0 22:38:57.445 MIA_Multivariable_test (USDCHF,D1) inputs [1,5,3] prediction [7.5] QI 0 22:38:57.445 MIA_Multivariable_test (USDCHF,D1) inputs [9,1,3] prediction [13.1] QK 0 22:38:57.445 MIA_Multivariable_test (USDCHF,D1) y = 1.900000e+00*x1 + 1.450000e+00*x2 - 9.500000e-01*x1*x2 + 3.100000e+00
El modelo puede mejorarse ajustando los parámetros de entrenamiento. Los mejores resultados se obtuvieron utilizando los ajustes que se muestran a continuación.
Con esta configuración, el modelo es capaz de realizar predicciones precisas sobre un conjunto de variables de entrada "invisibles". Aunque, al igual que en el primer ejemplo, el polinomio generado es excesivamente complejo.
DM 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) inputs [1,2,4] prediction [6.999999999999998] JI 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) inputs [1,5,3] prediction [8.999999999999998] CD 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) inputs [9,1,3] prediction [13.00000000000001] OO 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) f1_1 = 1.071429e-01*x1 + 6.428571e-01*x2 + 4.392857e+00 IQ 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) f1_2 = 6.086957e-01*x2 - 8.695652e-02*x3 + 4.826087e+00 PS 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) f1_3 = - 1.250000e+00*x1 - 1.500000e+00*x3 + 1.125000e+01 LO 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) f2_1 = 1.555556e+00*f1_1 - 6.666667e-01*f1_3 + 6.666667e-01 HN 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) f2_2 = 1.620805e+00*f1_2 - 7.382550e-01*f1_3 + 7.046980e-01 PP 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) f2_3 = 3.019608e+00*f1_1 - 2.029412e+00*f1_2 + 5.882353e-02 JM 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) f3_1 = 1.000000e+00*f2_1 - 3.731079e-15*f2_3 + 1.155175e-14 NO 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) f3_2 = 8.342665e-01*f2_2 + 1.713326e-01*f2_3 - 3.359462e-02 FD 0 22:44:25.269 MIA_Multivariable_test (USDCHF,D1) y = 1.000000e+00*f3_1 + 3.122149e-16*f3_2 - 1.899249e-15
De los ejemplos sencillos que hemos observado se desprende claramente que el algoritmo iterativo multicapa puede resultar excesivo para conjuntos de datos elementales. Los polinomios generados pueden complicarse mucho. Estos modelos corren el riesgo de sobreajustarse a los datos de entrenamiento. El algoritmo puede acabar captando ruido o valores atípicos en los datos, lo que da lugar a un mal rendimiento de la generalización en muestras no vistas. En general, el rendimiento de los algoritmos MIA y GMDH depende en gran medida de la calidad y las características de los datos de entrada. Los datos ruidosos o incompletos pueden afectar negativamente a la precisión y estabilidad del modelo, lo que puede dar lugar a predicciones poco fiables. Por último, aunque el proceso de entrenamiento es bastante sencillo, sigue siendo necesario un cierto nivel de ajuste de los hiperparámetros para obtener los mejores resultados. No está completamente automatizado.
Para nuestra última demostración, tenemos un script que carga un modelo desde un archivo y lo utiliza para hacer predicciones. Este ejemplo se da en LoadModelFromFile.mq5.
//+------------------------------------------------------------------+ //| LoadModelFromFile.mq5 | //| Copyright 2024, MetaQuotes Ltd. | //| https://www.mql5.com | //+------------------------------------------------------------------+ #property copyright "Copyright 2024, MetaQuotes Ltd." #property link "https://www.mql5.com" #property version "1.00" #property script_show_inputs #include <GMDH\mia.mqh> //--- input parameters input string JsonFileName=""; //+------------------------------------------------------------------+ //| Script program start function | //+------------------------------------------------------------------+ void OnStart() { //---declaration of MIA instance MIA mia; //---load the model from file if(!mia.load(JsonFileName)) return; //---get the number of required inputs for the loaded model int numlags = mia.getNumInputs(); //---generate arbitrary inputs to make a prediction with vector inputs(ulong(numlags),arange,21.0,1.0); //---make prediction and output results to terminal Print(JsonFileName," input ", inputs," prediction ", mia.predict(inputs,1)); //---output the model's polynomial Print(mia.getBestPolynomial()); } //+------------------------------------------------------------------+
El siguiente gráfico ilustra el funcionamiento del script y el resultado de una ejecución correcta.
Conclusión
La implementación del algoritmo iterativo multicapa GMDH en MQL5 ofrece a los operadores la oportunidad de aplicar el concepto en sus estrategias. Al ofrecer un marco dinámico, este algoritmo permite a los usuarios adaptar y perfeccionar continuamente sus análisis de mercado. Sin embargo, a pesar de sus promesas, es esencial que los profesionales sorteen sus limitaciones con criterio. Los usuarios deben ser conscientes de las exigencias computacionales inherentes a los algoritmos GMDH, sobre todo cuando se trata de conjuntos de datos extensos o de gran dimensionalidad. La naturaleza iterativa del algoritmo requiere múltiples cálculos para determinar la estructura óptima del modelo, lo que consume mucho tiempo y recursos en el proceso.
A la luz de estas consideraciones, se insta a los profesionales a abordar el uso del algoritmo iterativo multicapa GMDH con una comprensión matizada de sus puntos fuertes y sus limitaciones. Aunque ofrece una poderosa herramienta para el análisis dinámico de los mercados, sus complejidades exigen una navegación cuidadosa para aprovechar todo su potencial con eficacia. Mediante una aplicación cuidadosa y la consideración de sus complejidades, los operadores pueden aprovechar el algoritmo GMDH para enriquecer sus estrategias de negociación y obtener información valiosa de los datos del mercado.
Todo el código MQL5 se adjunta al final del artículo.
| Archivo | Descripción |
|---|---|
| Mql5\include\VectorMatrixTools.mqh | Archivo de cabecera de definiciones de funciones utilizadas para manipular vectores y matrices. |
| Mql5\include\JAson.mqh | Contiene la definición de los tipos personalizados utilizados para analizar y generar objetos JSON. |
| Mql5\include\GMDH\gmdh_internal.mqh | Archivo de cabecera que contiene las definiciones de tipos personalizados utilizados en la biblioteca GMDH. |
| Mql5\include\GMDH\gmdh.mqh | Archivo de inclusión con la definición de la clase base 'GmdhModel'. |
| Mql5\include\GMDH\mia.mqh | Contiene la clase 'MIA', que implementa el algoritmo iterativo multicapa. |
| Mql5\script\MIA_Test.mq5 | Un script que demuestra el uso de la clase 'MIA' mediante la construcción de un modelo de una serie temporal simple. |
| Mql5\script\MIA_Multivarible_test.mq5 | Otro script que muestra la aplicación de la clase 'MIA' para construir un modelo a partir de un conjunto de datos multivariable. |
| Mql5\script\LoadModelFromFile.mq5 | Script que demuestra cómo cargar un modelo desde un archivo JSON. |
Traducción del inglés realizada por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/en/articles/14454
 Introducción a MQL5 (Parte 6): Guía para principiantes sobre las funciones de matriz en MQL5 (II)
Introducción a MQL5 (Parte 6): Guía para principiantes sobre las funciones de matriz en MQL5 (II)
 Obtenga una ventaja sobre cualquier mercado
Obtenga una ventaja sobre cualquier mercado
 Aprendizaje automático y Data Science (Parte 20): Elección entre LDA y PCA en tareas de trading algorítmico en MQL5
Aprendizaje automático y Data Science (Parte 20): Elección entre LDA y PCA en tareas de trading algorítmico en MQL5
 Características del Wizard MQL5 que debe conocer (Parte 12): Polinomio de Newton
Características del Wizard MQL5 que debe conocer (Parte 12): Polinomio de Newton
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso