Algoritmos de optimización de la población: microsistema inmune artificial (Micro Artificial immune system, Micro-AIS)
Contenido:
1. Introducción
2. Descripción del algoritmo
3. Resultados de las pruebas
1. Introducción
El sistema inmunitario es un mecanismo asombroso que desempeña un papel esencial en la protección de nuestro organismo frente a amenazas externas. Como un escudo invisible, combate bacterias, virus y hongos, manteniendo nuestro organismo sano. Pero, ¿y si pudiéramos usar este potente mecanismo para resolver problemas complejos de optimización y aprendizaje? Este es el enfoque utilizado en el método de optimización del Sistema Inmune Artificial (Artificial Immune System, AIS).
El sistema inmunitario es un complejo sistema de células, tejidos y órganos que protege al organismo de infecciones, enfermedades y otras influencias externas. Actúa reconociendo y destruyendo sustancias extrañas y microorganismos como bacterias, virus, hongos, etcétera.
El algoritmo AIS modela estos procesos usando los conceptos de antígenos (entradas), anticuerpos (soluciones) y células asesinas (procesos de optimización) para resolver el problema de forma óptima. Los antígenos representan los datos de entrada que deben optimizarse. Los anticuerpos representan soluciones potenciales al problema. Las células asesinas son procesos de optimización que buscan las mejores soluciones a un problema de optimización.
En los años 90 se propuso un método de optimización conocido como Artificial Immune System (AIS). Las primeras investigaciones sobre este método se iniciaron a mediados de los años ochenta, cuando Farmer, Packard y Perelson (1986) y Bersini y Varela (1990) contribuyeron de forma significativa al desarrollo y la aplicación de los AIS en sus trabajos sobre redes inmunitarias.
Desde entonces, el método AIS ha seguido evolucionando y siendo objeto de investigación permanente en la comunidad científica: se han propuesto numerosas variaciones y modificaciones de este método, así como su aplicación a diversos problemas de optimización y aprendizaje. El sistema inmunitario del organismo también desempeña un papel vital en la protección contra influencias externas, como infecciones y tumores. Tiene capacidad para reconocer y detectar anomalías y atacar a agentes hostiles, al tiempo que conserva la capacidad de distinguirlos y guardar información sobre ellos para su uso futuro.
El Micro-AIS (Micro-Immune Algorithm) es una modificación del algoritmo del sistema inmune (AIS) que se ha desarrollado para resolver problemas de optimización. Se diferencia del AIS convencional en que usa un modelo más simple del sistema inmunitario y operaciones de procesamiento de la información inmunitaria más sencillas.
La idea básica del Micro-AIS es crear y hacer evolucionar una población de agentes que imiten a las células inmunitarias. Los agentes se mueven por el espacio de soluciones sin interactuar entre sí y sin intercambiar información sobre las soluciones. De este modo, los agentes pueden aprender y adaptarse a las condiciones cambiantes de la tarea.
El AIS convencional usa un modelo complejo del sistema inmunitario que incluye muchos tipos de células y moléculas como células B, células T, anticuerpos, citocinas, etc. El Micro-AIS utiliza un modelo más simple que solo incluye anticuerpos. Además, Micro-AIS usa operaciones de procesamiento de la información inmunitaria más sencillas, como la mutación y la selección.
Una de las ventajas de Micro-AIS, en comparación con AIS, es su sencillez y facilidad de aplicación, sin embargo, en algunos casos, un modelo más sofisticado del sistema inmunitario puede resultar más eficaz para resolver problemas complejos.
En general, la elección entre Micro-AIS y AIS convencional depende de la tarea específica y del contexto de la aplicación. Si el problema de optimización es relativamente simple y se requiere una solución rápida, Micro-AIS puede ser una buena elección. Si la tarea es más compleja y se requiere una solución más precisa, el AIS convencional puede ser una mejor opción.
2. Descripción del algoritmo
El Micro-AIS utiliza el concepto de afinidad para determinar la idoneidad, es una medida de similitud entre un anticuerpo y un antígeno. La afinidad mide el grado de similitud entre un anticuerpo y un antígeno. Cuanto mayor sea la afinidad, más similares serán el anticuerpo y el antígeno. En el Micro-AIS, la afinidad se utiliza para seleccionar los mejores anticuerpos que se usarán para crear nuevos anticuerpos mediante mutación y cruce. Los anticuerpos con mayor afinidad tendrán más probabilidades de ser seleccionados para crear nuevos anticuerpos.
La afinidad puede definirse como la distancia entre el vector de características de un objeto y el vector de pesos del clasificador. Cuanto menor sea la distancia, más similares serán los vectores y mayor será la afinidad. En general, la afinidad puede definirse como una función de la distancia entre dos números de coma flotante. La función de distancia puede seleccionarse según la tarea específica y los requisitos del algoritmo Micro-AIS. Por ejemplo, para los problemas de optimización, la distancia suele definirse como distancia euclidiana, distancia Manhattan o de otro tipo.
No obstante, los experimentos con Micro-AIS han demostrado que el uso de la afinidad no es el enfoque más eficaz en esta estrategia de búsqueda, y en su lugar se puede usar directamente el valor de la función de aptitud.
El Micro-AIS original usa una aplicación probabilística de las mutaciones para los genes, en la que cuanto mayor sea la adaptación, menos probable será la mutación. Este enfoque también tuvo que desecharse debido a su ineficacia, que por cierto, se puede comprobar fácilmente: solo hay que añadir un par de líneas de código.
Pseudocódigo de Micro-AIS:
- Creamos una población de clones de anticuerpos y los distribuimos aleatoriamente por el espacio de búsqueda. Los anticuerpos y sus clones representan soluciones potenciales al problema de la optimización. Los clones se crean generando aleatoriamente un genotipo que determine los valores de los parámetros del problema de optimización.
- Definimos la aptitud, que supone una medida de la calidad de la solución. Podemos calcular el valor de aptitud evaluando la función objetivo para cada anticuerpo.
- Para que cada anticuerpo cree clones en la cantidad correspondiente a la regla de progresión decreciente, el primer anticuerpo creará más clones que el segundo, el segundo más que el tercero y así sucesivamente. Así pues, el número de clones no se corresponderá con el grado de aptitud, sino con una regla de progresión rígidamente definida. Los anticuerpos más adaptados crearán más clones que los menos adaptados siempre en la misma proporción.
- Aplicamos la mutación para clonar genes. En cada clon se produce una mutación del genotipo, lo cual permitirá crear nuevas soluciones y explorar el espacio de parámetros del problema de optimización.
- Determinamos de la aptitud de los clones.
- Tras el cálculo de la mutación y la adaptación, la población de clones se añadirá a la población padre de anticuerpos.
- Clasificamos la población (anticuerpos+clones) en orden descendente de aptitud, de modo que las mejores soluciones puedan ser seleccionadas para crear una nueva población de clones en la siguiente iteración, realizando así la competencia entre los descendientes de los clones y los anticuerpos padre.
- Repetimos desde el paso 2 hasta que se cumpla el criterio de parada. El criterio de parada puede definirse de antemano, por ejemplo, al alcanzar un determinado valor de aptitud o un número máximo de iteraciones.
La mutación de genes en clones consiste en la generación de números aleatorios con distribución uniforme según la fórmula:
X' = X + dist * rnd * k * mutation
donde:
- X' - nuevo valor del gen (coordenadas) del clon
- X - valor del gen del anticuerpo padre
- dist - incremento del gen padre
- rnd - número aleatorio con distribución uniforme en el intervalo [-1,0;1,0].
- k - coeficiente uniformemente decreciente según la época actual
- mutation - coeficiente de mutación, en la práctica, el factor de escala de incremento (parámetro externo)
Calcularemos el coeficiente "k" usando la siguiente fórmula:
k = (epochs - epochsCNT) / epochs
donde:
- epochs - valor límite de épocas
- epochsCNT - contador de épocas (iteraciones)
El tamaño de incremento "dist" será la distancia desde el valor máximo del parámetro optimizado hasta "X" si "rnd" es mayor que 0 y desde "X" hasta el valor mínimo del parámetro optimizado en caso contrario.
Así, la mutación permitirá una variación aleatoria de los valores de los parámetros de la solución, lo cual posibilitará una exploración del espacio del problema. El coeficiente decreciente "k" permitirá reducir la probabilidad de cambios demasiado bruscos de los parámetros en iteraciones posteriores, mejorando así la convergencia del algoritmo hacia la solución óptima y afinando las coordenadas encontradas.
Vamos a escribir una estructura que actúe como anticuerpo, S_Agent. La estructura solo contiene dos campos:
- c: array de coordenadas del agente
- f: indicador de aptitud
El método "Init" inicializa los campos de la estructura, redimensiona el array "c" y asigna el valor inicial "-DBL_MAX" al campo "f".
//—————————————————————————————————————————————————————————————————————————————— struct S_Agent { void Init (int coords) { ArrayResize (c, coords); f = -DBL_MAX; } double c []; //coordinates double f; //fitness }; //——————————————————————————————————————————————————————————————————————————————
Ahora declaramos una clase de algoritmo de microsistema inmune C_AO_Micro_AIS en la que se definen varios campos y métodos.
Campos de clase:
- cB: array con las mejores coordenadas.
- fB: índice de aptitud de ajuste de las mejores coordenadas.
- a: array de agentes de tipo "S_Agent".
- rangeMax: array de valores máximos del rango de búsqueda.
- rangeMin: array de valores mínimos del rango de búsqueda.
- rangeStep: array de pasos de búsqueda.
Las variables "coords", "popSize", "minClonesNumber", "cloneStep", "mutation" y "epochs" tomarán los parámetros externos del algoritmo.
Métodos de clase:
- Init: inicializa los campos de la clase con los valores establecidos.
- Moving: realiza el desplazamiento de los agentes.
- Revision: realiza la revisión.
También se definen en la clase los métodos privados "SeInDiSp", "RNDfromCI", "Sorting" para la normalización, la generación de números aleatorios y la clasificación, respectivamente.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_Micro_AIS { //---------------------------------------------------------------------------- public: double cB []; //best coordinates public: double fB; //FF of the best coordinates public: S_Agent a []; //agent public: double rangeMax []; //maximum search range public: double rangeMin []; //manimum search range public: double rangeStep []; //step search public: void Init (const int coordsP, //coordinates number const int popSizeP, //population size const int minClonesNumberP, //minimum number of clones const int cloneStepP, //clone step const double mutationP, //mutation const int epochP); //total epochs public: void Moving (); public: void Revision (); //---------------------------------------------------------------------------- private: int coords; //coordinates number private: int popSize; //population size private: int minClonesNumber; //minimum number of clones private: int cloneStep; //clone step private: double mutation; //mutation private: int epochs; //total epochs private: int epochsCNT; //epoch counter private: int parentsNumb; //number of parents private: bool revision; private: S_Agent parents []; //parents private: int ind []; private: double val []; private: S_Agent pTemp []; private: int cCnt []; //clone counters for each antibody private: double SeInDiSp (double In, double InMin, double InMax, double Step); private: double RNDfromCI (double min, double max); private: void Sorting (S_Agent &p [], int size); }; //——————————————————————————————————————————————————————————————————————————————
Para inicializar el objeto de clase, implementaremos el método "Init" para inicializar los campos de clase con los valores especificados.
Al principio del método, el generador de números aleatorios se inicializa usando la función "MathSrand" y su estado se restablece mediante la función "GetMicrosecondCount".
A continuación, asignamos a las variables "fB" y "revision" los valores "-DBL_MAX" y "false", respectivamente, e inicializamos los campos privados con los parámetros de entrada del método.
Luego tiene lugar el cálculo de los valores del array "cCnt" utilizado para el almacenamiento el número de clones de cada anticuerpo con ayuda de un ciclo utilizando una fórmula de progresión en la que el primer término de la progresión es "a1", la diferencia de la progresión es "d" y la suma de todos los términos de la progresión es "Sn". Los valores de la progresión se almacenarán en el array "cCnt".
A continuación, el método define el valor de la variable "parentsNumb" como el tamaño del array "cCnt".
Luego se muestra el redimensionamiento de los arrays: "ind", "val", "pTemp", "a", "parents", "rangeMax", "rangeMin", "rangeStep" y "cB". Los tamaños de los arrays se establecen en función de los valores "popSize" y "parentsNumb".
A continuación, los elementos del array "a" se inicializan en un ciclo usando el método "Init" de la clase "S_Agent", y los elementos del array "parents" también se inicializan mediante el método "Init".
Al final del método, los arrays "rangeMax", "rangeMin", "rangeStep" y "cB" se redimensionan según el valor "coords".
Así, el método "Init" inicializa los campos de la clase "C_AO_Micro_AIS" y calcula los valores de progresión para el array "cCnt".
//—————————————————————————————————————————————————————————————————————————————— void C_AO_Micro_AIS::Init (const int coordsP, //coordinates number const int popSizeP, //population size const int minClonesNumberP, //minimum number of clones const int cloneStepP, //clone step const double mutationP, //mutation const int epochP) //total epochs { MathSrand ((int)GetMicrosecondCount ()); // reset of the generator fB = -DBL_MAX; revision = false; coords = coordsP; popSize = popSizeP; minClonesNumber = minClonesNumberP; cloneStep = cloneStepP; mutation = mutationP; epochs = epochP; epochsCNT = 1; //---------------------------------------------------------------------------- int Sn = popSize; //suma int a1 = minClonesNumber; //primer miembro de la progresión int d = cloneStep; //paso de la progresión int an = 0; //n-ésimo miembro de la progresión, int Ssum = 0; ArrayResize (cCnt, 1); for (int n = 1;; n++) { an = a1 + (n - 1) * d; Ssum = n * (a1 + an) / 2; if (Ssum == Sn) { ArrayResize (cCnt, n); cCnt [n - 1] = an; break; } else { if (Ssum < Sn) { ArrayResize (cCnt, n); cCnt [n - 1] = an; } else { if (n == 1) { ArrayResize (cCnt, n); cCnt [n - 1] = Sn; break; } else { n--; an = a1 + (n - 1) * d; int diff = Sn - ((n) * (a1 + an) / 2); int index = ArraySize (cCnt) - 1; while (true) { if (index < 0) index = ArraySize (cCnt) - 1; cCnt [index]++; index--; diff--; if (diff <= 0) break; } break; } } } } parentsNumb = ArraySize (cCnt); ArrayReverse (cCnt, 0, WHOLE_ARRAY); ArrayResize (ind, popSize + parentsNumb); ArrayResize (val, popSize + parentsNumb); ArrayResize (pTemp, popSize + parentsNumb); ArrayResize (a, popSize); for (int i = 0; i < popSize; i++) a [i].Init (coords); ArrayResize (parents, popSize + parentsNumb); for (int i = 0; i < popSize + parentsNumb; i++) parents [i].Init (coords); ArrayResize (rangeMax, coords); ArrayResize (rangeMin, coords); ArrayResize (rangeStep, coords); ArrayResize (cB, coords); } //——————————————————————————————————————————————————————————————————————————————
Usanod el método de la clase "Moving", implementamos el desplazamiento de los anticuerpos a través del espacio de búsqueda.
Al principio del método, comprobamos el valor de la variable "revision". Si es "false", la ubicación de los clones de los anticuerpos en el espacio de búsqueda se realizará generando coordenadas con distribución uniforme.
Una vez se hayan generado los clones de los anticuerpos en la población, asignaremos a la variable "revision" el valor "true" y el método finalizará.
Si el valor de la variable "revision" no es "false", se ejecutará el siguiente bloque de código.
A esto le seguirá un ciclo "for" anidado que iterará sobre el número de agentes padre "parentsNumb". Dentro de este ciclo, ocurre lo siguiente:
- el ciclo "for" anidado se itera sobre el número de clones para el anticuerpo padre dado "cCnt[i]".
- dentro de este ciclo, se itera un ciclo "for" anidado sobre todas las coordenadas c del agente.
- el valor de la coordenada "a[indx].c[c]" se establece igual al valor de la coordenada "parents[i].c[c]".
A continuación, ejecutamos el siguiente bloque de código:
- calculamos el valor de la variable "k" como la diferencia entre "epochs" y "epochsCNT" dividida por "epochs".
- generamos un número aleatorio "rnd" en el intervalo [-1,0;1,0].
- "rnd" es mayor que 0,0, entonces el valor de la variable "dist" se calculará como la diferencia entre "rangeMax[c]" y "a[indx].c[c]". En caso contrario, "dist" será igual a la diferencia entre "a[indx].c[c]" y "rangeMin[c]".
- el valor de "a[indx].c[c]" se recalcula usando la fórmula "a[indx].c[c] + dist * rnd * k * mutation". Aquí, "mutation" es la tasa de mutación.
- El valor "a[indx].c[c]" pasa por la función "SeInDiSp", que lo normaliza en el rango de "rangeMin[c]" a "rangeMax[c]" con el paso "rangeStep[c]".
//—————————————————————————————————————————————————————————————————————————————— void C_AO_Micro_AIS::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } revision = true; return; } //---------------------------------------------------------------------------- int indx = 0; double min = DBL_MAX; double max = -DBL_MAX; double dist = 0.0; int cnt = 0; double rnd = 0.0; for (int i = 0; i < parentsNumb; i++) { for (int cl = 0; cl < cCnt [i]; cl++) { for (int c = 0; c < coords; c++) { a [indx].c [c] = parents [i].c [c]; //---------------------------------------------------------------------- double k = ((double)epochs - (double)epochsCNT) / (double)epochs; rnd = RNDfromCI (-1.0, 1.0); if (rnd > 0.0) dist = (rangeMax [c] - a [indx].c [c]); else dist = (a [indx].c [c] - rangeMin [c]); a [indx].c [c] = a [indx].c [c] + dist * rnd * k * mutation; a [indx].c [c] = SeInDiSp (a [indx].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } indx++; } } } //——————————————————————————————————————————————————————————————————————————————
Y por último, implementamos el método "Revision". Este método realiza una revisión del estado actual de la población de agentes en el algoritmo Micro-AIS.
El primer bloque de código, separado por un comentario, realizamos una actualización de la mejor solución global comprobando los valores de aptitud de la población de clones.
A continuación, copiamos los clones de la población de antígenos padre hasta el final del array en un ciclo.
Luego llamamos a la función "Sorting" con los argumentos "parents" y "parentsNumb + popSize". Esta función clasifica el array padre según el valor del índice de aptitud en orden descendente.
Al final del método, incrementamos la variable "epochsCNT", que se encarga de contar el número de épocas del algoritmo.
Así, el método Revision realiza una revisión del estado actual de la población de anticuerpos (agentes), encuentra el agente con el mejor valor de índice de aptitud y actualiza el array de agentes padre.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_Micro_AIS::Revision () { //---------------------------------------------------------------------------- int indx = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) indx = i; } if (indx != -1) { fB = a [indx].f; ArrayCopy (cB, a [indx].c, 0, 0, WHOLE_ARRAY); } //---------------------------------------------------------------------------- for (int i = parentsNumb; i < parentsNumb + popSize; i++) { parents [i] = a [i - parentsNumb]; } Sorting (parents, parentsNumb + popSize); epochsCNT++; } //——————————————————————————————————————————————————————————————————————————————
3. Resultados de las pruebas
Impresión del rendimiento del algoritmo de microsistema inmune artificial en un banco de pruebas:
C_AO_Micro_AIS:50:1:2:0.3
=============================
5 Hilly's; Func runs: 10000; result: 0.7954680903046107
25 Hilly's; Func runs: 10000; result: 0.5192246492565626
500 Hilly's; Func runs: 10000; result: 0.30860655744850657
=============================
5 Forest's; Func runs: 10000; result: 0.7295587642801589
25 Forest's; Func runs: 10000; result: 0.36878621216829993
500 Forest's; Func runs: 10000; result: 0.09398090798741626
=============================
5 Megacity's; Func runs: 10000; result: 0.37666666666666665
25 Megacity's; Func runs: 10000; result: 0.15866666666666668
500 Megacity's; Func runs: 10000; result: 0.028016666666666672
=============================
All score: 3.37898 (37.54%)
Desde el artículo anterior, hemos comenzado a estudiar los valores absolutos de los resultados de las pruebas, y ahora resulta muy sencillo navegar y comparar los resultados de distintos algoritmos en la tabla. Un "37,54%" no es un resultado sobresaliente, pero no deja de suponer una posición en la mitad superior de la tabla.
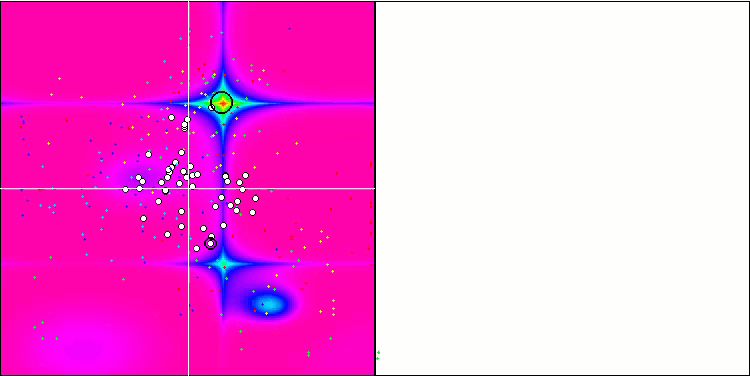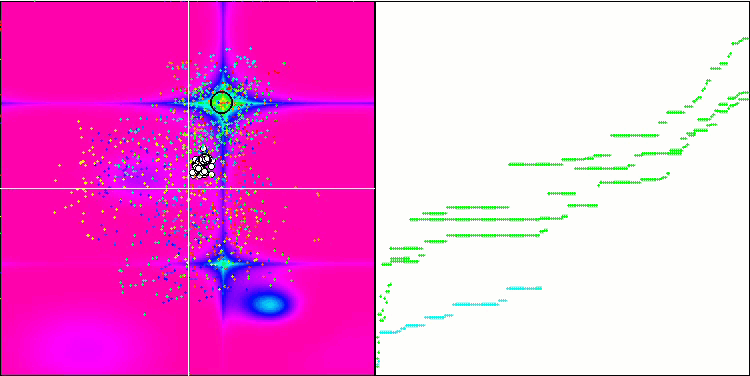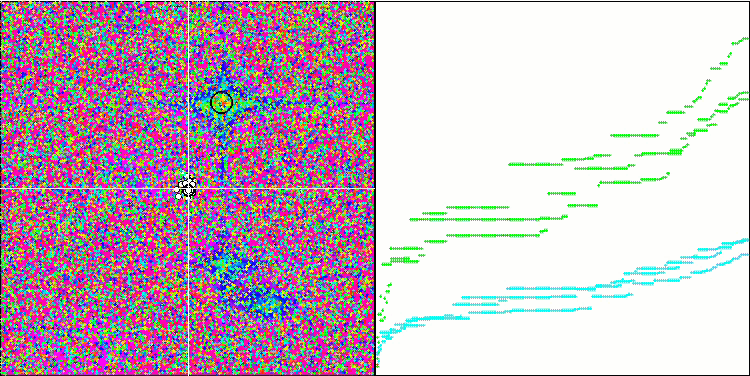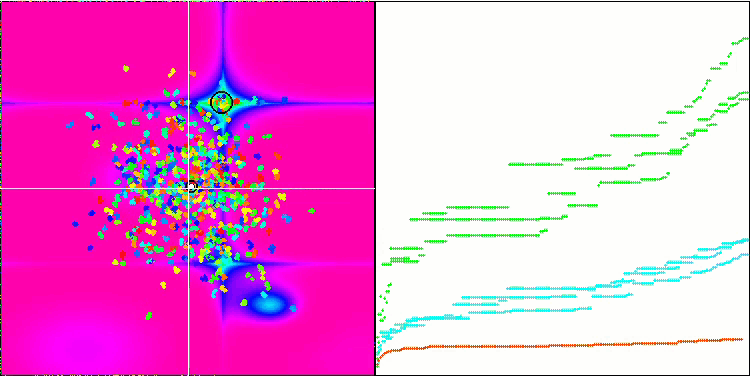
La visualización del proceso de optimización ha mostrado que el algoritmo demuestra persistencia en la exploración de extremos locales significativos para alcanzar una solución mejor. Esto provoca una concentración de agentes en diferentes regiones del espacio. El gráfico de convergencia ha mostrado un comportamiento inusual. Por lo general, en los algoritmos analizados se produce un fuerte aumento de la convergencia en la primera mitad de las iteraciones y, a continuación, la tasa de convergencia disminuye suavemente. Sin embargo, en este algoritmo, el gráfico de convergencia tiene forma de S. Solo se observa un fuerte aumento de la convergencia en el primer 10-20% de las iteraciones, después la tasa de convergencia disminuye, pero más cerca del final de la optimización se vuelve a observar una aceleración significativa de la convergencia.
Este comportamiento del gráfico de convergencia puede explicarse por la estrategia de reducción del rango de incrementos en las mutaciones según la ley lineal. Sin embargo, la convergencia no sigue una ley lineal debido a que el algoritmo "acumula" de forma desigual la información sobre la superficie de la función de prueba. El estrechamiento del rango de mutación empieza a desempeñar un papel más destacado solo cerca del final de la optimización. La sustitución de la ley lineal de estrechamiento del intervalo de mutaciones por otras variantes de leyes no lineales no ha producido una mejora de la convergencia, pero tal vez haya lugar para la imaginación constructiva de los investigadores en la elección de otras variantes de leyes de estrechamiento del intervalo.
El gráfico de convergencia deja mucho que desear, sin embargo, su aparición da esperanzas de seguir mejorando la estrategia de búsqueda.
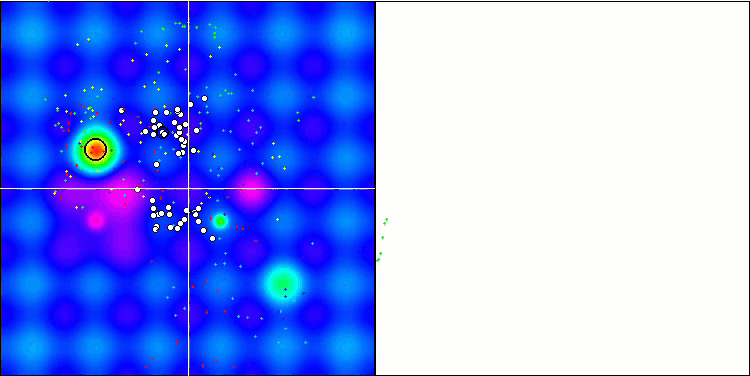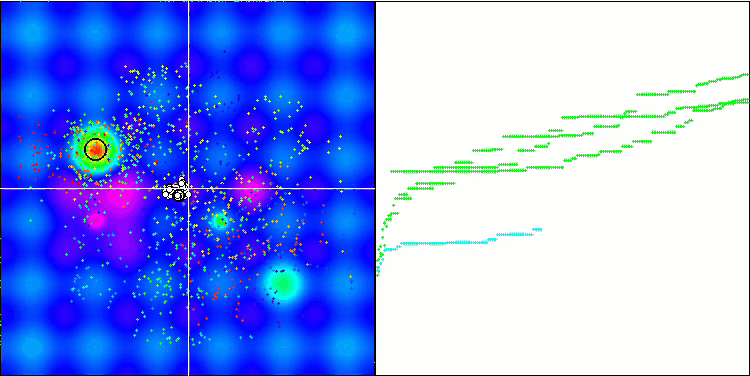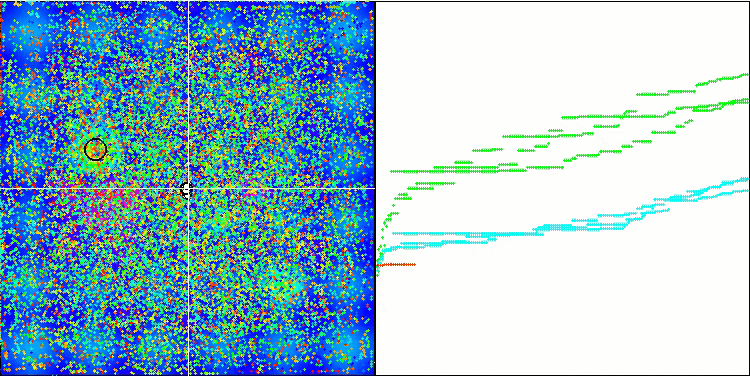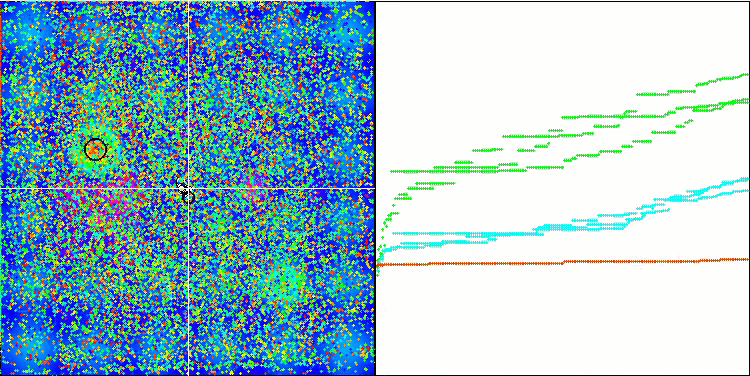
Micro-AIS en la función de prueba Hilly.
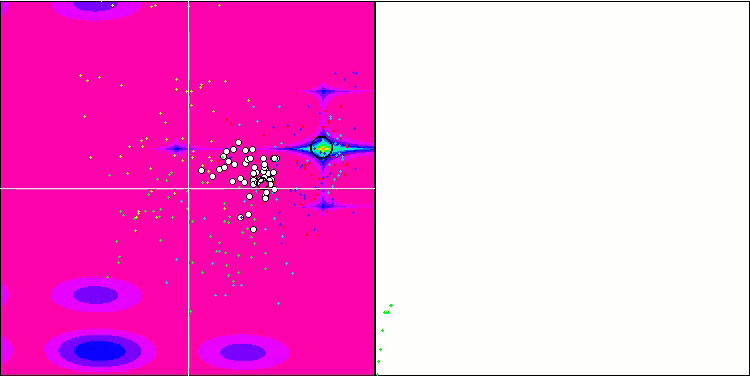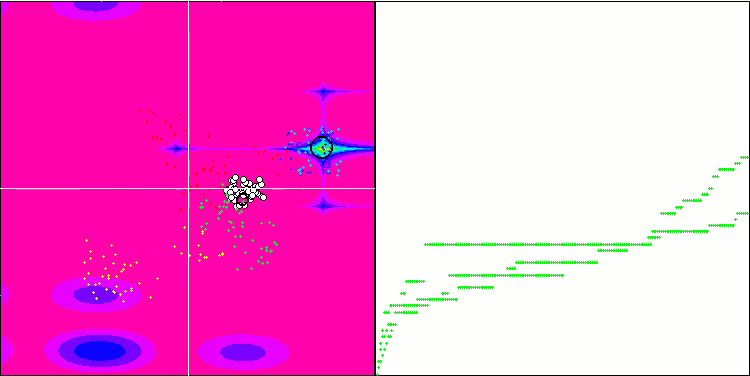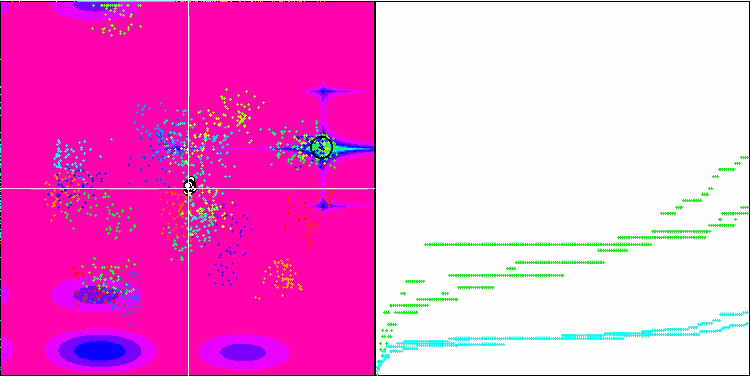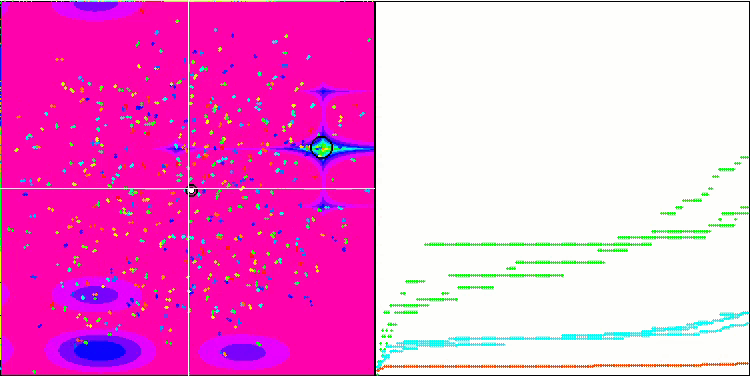
Micro-AIS en la función de prueba Forest.
Micro-AIS en la función de prueba Megacity.
El algoritmo Micro-AIS ha ocupado la undécima línea de la tabla de clasificación, por delante de algoritmos tan conocidos y populares como el algoritmo de optimización de cuco, la colonia artificial de abejas y el recocido simulado. Esto indica la eficacia y el prometedor rendimiento de este algoritmo para resolver problemas de optimización complejos.
No obstante, en la tabla de colores podemos observar una disminución del rendimiento en funciones con un gran número de variables, lo cual indica que la capacidad de escalado del algoritmo es débil. Esto puede deberse al hecho de que Micro-AIS usa un modelo de sistema inmune más simple y operaciones de procesamiento de la información inmune más sencillas, lo que puede limitar su capacidad para encontrar la solución óptima en espacios de alta dimensionalidad.
Sin embargo, esto no significa que Micro-AIS no pueda usarse para resolver problemas con un gran número de variables. Quizá podamos mejorar su rendimiento modificando el algoritmo o combinándolo con otras técnicas de optimización.
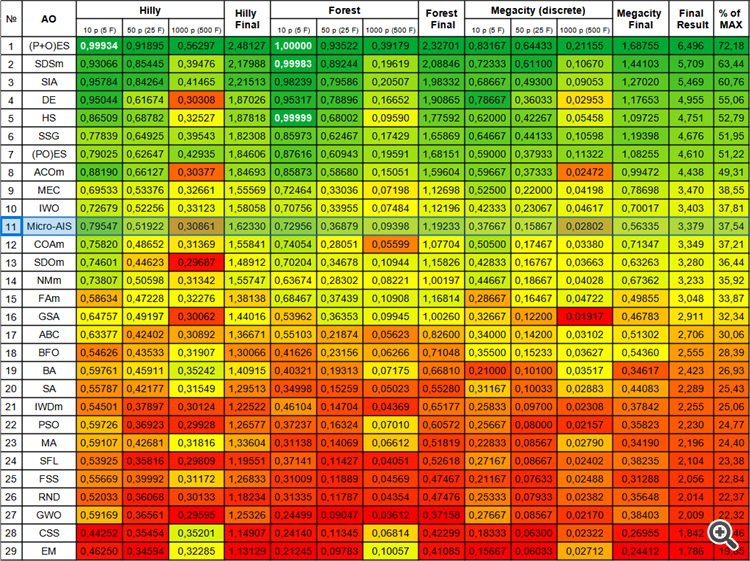
| № | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % de MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | (P+O)ES | (P+O) evolution strategies | 0,99934 | 0,91895 | 0,56297 | 2,48127 | 1,00000 | 0,93522 | 0,39179 | 2,32701 | 0,83167 | 0,64433 | 0,21155 | 1,68755 | 6,496 | 72,18 |
| 2 | SDSm | stochastic diffusion search M | 0,93066 | 0,85445 | 0,39476 | 2,17988 | 0,99983 | 0,89244 | 0,19619 | 2,08846 | 0,72333 | 0,61100 | 0,10670 | 1,44103 | 5,709 | 63,44 |
| 3 | SIA | simulated isotropic annealing | 0,95784 | 0,84264 | 0,41465 | 2,21513 | 0,98239 | 0,79586 | 0,20507 | 1,98332 | 0,68667 | 0,49300 | 0,09053 | 1,27020 | 5,469 | 60,76 |
| 4 | DE | differential evolution | 0,95044 | 0,61674 | 0,30308 | 1,87026 | 0,95317 | 0,78896 | 0,16652 | 1,90865 | 0,78667 | 0,36033 | 0,02953 | 1,17653 | 4,955 | 55,06 |
| 5 | HS | harmony search | 0,86509 | 0,68782 | 0,32527 | 1,87818 | 0,99999 | 0,68002 | 0,09590 | 1,77592 | 0,62000 | 0,42267 | 0,05458 | 1,09725 | 4,751 | 52,79 |
| 6 | SSG | saplings sowing and growing | 0,77839 | 0,64925 | 0,39543 | 1,82308 | 0,85973 | 0,62467 | 0,17429 | 1,65869 | 0,64667 | 0,44133 | 0,10598 | 1,19398 | 4,676 | 51,95 |
| 7 | (PO)ES | (PO) evolution strategies | 0,79025 | 0,62647 | 0,42935 | 1,84606 | 0,87616 | 0,60943 | 0,19591 | 1,68151 | 0,59000 | 0,37933 | 0,11322 | 1,08255 | 4,610 | 51,22 |
| 8 | ACOm | ant colony optimization M | 0,88190 | 0,66127 | 0,30377 | 1,84693 | 0,85873 | 0,58680 | 0,15051 | 1,59604 | 0,59667 | 0,37333 | 0,02472 | 0,99472 | 4,438 | 49,31 |
| 9 | MEC | mind evolutionary computation | 0,69533 | 0,53376 | 0,32661 | 1,55569 | 0,72464 | 0,33036 | 0,07198 | 1,12698 | 0,52500 | 0,22000 | 0,04198 | 0,78698 | 3,470 | 38,55 |
| 10 | IWO | invasive weed optimization | 0,72679 | 0,52256 | 0,33123 | 1,58058 | 0,70756 | 0,33955 | 0,07484 | 1,12196 | 0,42333 | 0,23067 | 0,04617 | 0,70017 | 3,403 | 37,81 |
| 11 | Micro-AIS | micro artificial immune system | 0,79547 | 0,51922 | 0,30861 | 1,62330 | 0,72956 | 0,36879 | 0,09398 | 1,19233 | 0,37667 | 0,15867 | 0,02802 | 0,56335 | 3,379 | 37,54 |
| 12 | COAm | cuckoo optimization algorithm M | 0,75820 | 0,48652 | 0,31369 | 1,55841 | 0,74054 | 0,28051 | 0,05599 | 1,07704 | 0,50500 | 0,17467 | 0,03380 | 0,71347 | 3,349 | 37,21 |
| 13 | SDOm | spiral dynamics optimization M | 0,74601 | 0,44623 | 0,29687 | 1,48912 | 0,70204 | 0,34678 | 0,10944 | 1,15826 | 0,42833 | 0,16767 | 0,03663 | 0,63263 | 3,280 | 36,44 |
| 14 | NMm | Nelder-Mead method M | 0,73807 | 0,50598 | 0,31342 | 1,55747 | 0,63674 | 0,28302 | 0,08221 | 1,00197 | 0,44667 | 0,18667 | 0,04028 | 0,67362 | 3,233 | 35,92 |
| 15 | FAm | firefly algorithm M | 0,58634 | 0,47228 | 0,32276 | 1,38138 | 0,68467 | 0,37439 | 0,10908 | 1,16814 | 0,28667 | 0,16467 | 0,04722 | 0,49855 | 3,048 | 33,87 |
| 16 | GSA | gravitational search algorithm | 0,64757 | 0,49197 | 0,30062 | 1,44016 | 0,53962 | 0,36353 | 0,09945 | 1,00260 | 0,32667 | 0,12200 | 0,01917 | 0,46783 | 2,911 | 32,34 |
| 17 | ABC | artificial bee colony | 0,63377 | 0,42402 | 0,30892 | 1,36671 | 0,55103 | 0,21874 | 0,05623 | 0,82600 | 0,34000 | 0,14200 | 0,03102 | 0,51302 | 2,706 | 30,06 |
| 18 | BFO | bacterial foraging optimization | 0,54626 | 0,43533 | 0,31907 | 1,30066 | 0,41626 | 0,23156 | 0,06266 | 0,71048 | 0,35500 | 0,15233 | 0,03627 | 0,54360 | 2,555 | 28,39 |
| 19 | BA | bat algorithm | 0,59761 | 0,45911 | 0,35242 | 1,40915 | 0,40321 | 0,19313 | 0,07175 | 0,66810 | 0,21000 | 0,10100 | 0,03517 | 0,34617 | 2,423 | 26,93 |
| 20 | SA | simulated annealing | 0,55787 | 0,42177 | 0,31549 | 1,29513 | 0,34998 | 0,15259 | 0,05023 | 0,55280 | 0,31167 | 0,10033 | 0,02883 | 0,44083 | 2,289 | 25,43 |
| 21 | IWDm | intelligent water drops M | 0,54501 | 0,37897 | 0,30124 | 1,22522 | 0,46104 | 0,14704 | 0,04369 | 0,65177 | 0,25833 | 0,09700 | 0,02308 | 0,37842 | 2,255 | 25,06 |
| 22 | PSO | particle swarm optimisation | 0,59726 | 0,36923 | 0,29928 | 1,26577 | 0,37237 | 0,16324 | 0,07010 | 0,60572 | 0,25667 | 0,08000 | 0,02157 | 0,35823 | 2,230 | 24,77 |
| 23 | MA | monkey algorithm | 0,59107 | 0,42681 | 0,31816 | 1,33604 | 0,31138 | 0,14069 | 0,06612 | 0,51819 | 0,22833 | 0,08567 | 0,02790 | 0,34190 | 2,196 | 24,40 |
| 24 | SFL | shuffled frog-leaping | 0,53925 | 0,35816 | 0,29809 | 1,19551 | 0,37141 | 0,11427 | 0,04051 | 0,52618 | 0,27167 | 0,08667 | 0,02402 | 0,38235 | 2,104 | 23,38 |
| 25 | FSS | fish school search | 0,55669 | 0,39992 | 0,31172 | 1,26833 | 0,31009 | 0,11889 | 0,04569 | 0,47467 | 0,21167 | 0,07633 | 0,02488 | 0,31288 | 2,056 | 22,84 |
| 26 | RND | random | 0,52033 | 0,36068 | 0,30133 | 1,18234 | 0,31335 | 0,11787 | 0,04354 | 0,47476 | 0,25333 | 0,07933 | 0,02382 | 0,35648 | 2,014 | 22,37 |
| 27 | GWO | grey wolf optimizer | 0,59169 | 0,36561 | 0,29595 | 1,25326 | 0,24499 | 0,09047 | 0,03612 | 0,37158 | 0,27667 | 0,08567 | 0,02170 | 0,38403 | 2,009 | 22,32 |
| 28 | CSS | charged system search | 0,44252 | 0,35454 | 0,35201 | 1,14907 | 0,24140 | 0,11345 | 0,06814 | 0,42299 | 0,18333 | 0,06300 | 0,02322 | 0,26955 | 1,842 | 20,46 |
| 29 | EM | electroMagnetism-like algorithm | 0,46250 | 0,34594 | 0,32285 | 1,13129 | 0,21245 | 0,09783 | 0,10057 | 0,41085 | 0,15667 | 0,06033 | 0,02712 | 0,24412 | 1,786 | 19,85 |
Conclusiones
El método de optimización mediante el microsistema inmune artificial (Micro-AIS) es un planteamiento interesante y prometedor para resolver problemas de optimización basados en los principios de funcionamiento del sistema inmune. Aprovecha el potente mecanismo del sistema inmune para resolver complejos problemas de optimización y aprendizaje.
Las ventajas de Micro-AIS incluyen pocos parámetros externos y una implementación sencilla del algoritmo. Esto lo hace muy atractivo para su uso en aplicaciones prácticas.
Sin embargo, el algoritmo Micro-AIS también tiene desventajas. Tiende a atascarse en extremos locales y posee una baja convergencia. Además, el rendimiento del algoritmo disminuye en funciones con un gran número de variables, lo cual indica su escasa escalabilidad.
No obstante, el Micro-AIS es un método de optimización prometedor que puede mejorarse modificando el algoritmo o combinándolo con otros métodos de optimización. En conjunto, el método de optimización mediante Micro-AIS constituye una importante contribución al campo de la optimización y puede ser una herramienta útil para resolver problemas complejos en diversos campos, como el aprendizaje automático, la inteligencia artificial, la bioinformática, etc.
El algoritmo deja la impresión de ser una especie de "plantilla" sobre la que se puede construir usando diferentes métodos y ampliando las capacidades de búsqueda, lo cual se ve favorecido por la sencilla arquitectura del algoritmo, que deja un margen realmente enorme para experimentar con este algoritmo tan interesante.
Figura 1. Gradación por colores de los algoritmos según sus respectivas pruebas.
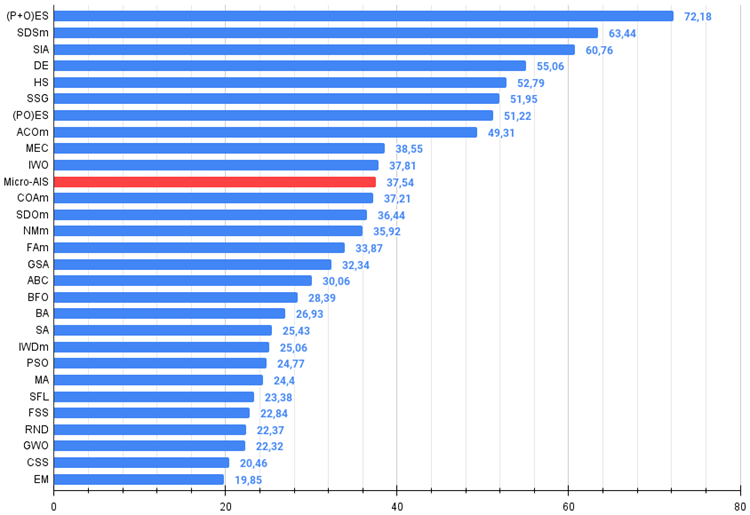
Figura 2. Histograma con los resultados de las pruebas de los algoritmos (en una escala de 0 a 100, cuanto mayor, mejor),
donde 100 es el máximo resultado teórico posible; hay un script para calcular la tabla de puntuación en el archivo).
Ventajas y desventajas del algoritmo del microsistema inmune artificial (Micro-AIS):
- Número reducido de parámetros externos.
- Aplicación sencilla del algoritmo.
- Tendencia a atascarse.
- Baja convergencia.
Adjuntamos al artículo un archivo con las versiones reales actualizadas de los códigos de los algoritmos descritos en los artículos anteriores. El autor de este artículo no se responsabiliza de la exactitud absoluta de la descripción de los algoritmos canónicos, muchos de ellos han sido modificados para mejorar las capacidades de búsqueda. Las conclusiones y juicios presentados en los artículos se basan en los resultados de los experimentos realizados.
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/13951
 Utilizando redes neuronales en MetaTrader
Utilizando redes neuronales en MetaTrader
 Aprendiendo MQL5 de principiante a profesional (Parte II): Tipos de datos básicos y uso de variables
Aprendiendo MQL5 de principiante a profesional (Parte II): Tipos de datos básicos y uso de variables
 Particularidades del trabajo con números del tipo double en MQL4
Particularidades del trabajo con números del tipo double en MQL4
 Algoritmos de optimización de la población: Algoritmo híbrido de optimización de forrajeo bacteriano con algoritmo genético (Bacterial Foraging Optimization - Genetic Algorithm, BFO-GA)
Algoritmos de optimización de la población: Algoritmo híbrido de optimización de forrajeo bacteriano con algoritmo genético (Bacterial Foraging Optimization - Genetic Algorithm, BFO-GA)
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso