
Алгорим оптимизации химическими реакциями — Chemical reaction optimisation, CRO (Часть II): Сборка и результаты
Содержание:
1. Введение
Во второй части нашей статьи мы продолжаем погружение в увлекательный мир оптимизации химическими реакциями (CRO). Основываясь на концепции "молекул" и "элементарных реакций", мы познакомились с принципами, лежащими в основе работы алгоритма, и рассмотрели, как эти концепции применяются для решения сложных задач оптимизации. Также мы узнали о ключевых моментах сохранения энергии в рамках CRO и функциях алгоритма, таких как: декомпозиция, синтез, внутримолекулярное и межмолекулярное неэффективные столкновения, которые выполняют главную роль в исследовании пространства поиска и нахождении оптимальных решений.
После того, как мы рассмотрели основные концепции и принципы работы химических операторов алгоритма оптимизации химическими реакциями, теперь пришло время перейти к общей сборке алгоритма и к практическому применению. В этой части статьи мы сфокусируемся на результатах работы алгоритма на различных тестовых функциях, чтобы проанализировать его эффективность и потенциал в решении реальных задач. Мы рассмотрим его производительность, сходимость и способность находить глобальные оптимумы, что позволит нам оценить его применимость, а также сравним результаты работы алгоритма CRO с другими методами оптимизации, выявив его преимущества и недостатки.
2.Реализация алгоритма
Продолжим написание кода алгоритма по уже знакомому вам шаблону для всех алгоритмов, который включает стандартные функции: "Init", "Moving", "Revision", и так как структура у нас уже объявлена, написаны основные операторы химических реакций, перейдем к написанию класса алгоритма, чтобы связать все компоненты вместе в одно целое.
Нам понадобится написать псевдокод, чтобы осуществить сборку алгоритма:
Если флаг ревизии сброшен - инициализация:
Для каждой молекулы i от 0 до popSize:
Для каждой координаты c от 0 до coords
Генерировать случайное значение для структуры молекулы в диапазоне от rangeMin до rangeMax
Ограничить значение структуры в заданном диапазоне
Сохранить структуру в массиве a
Выход
Расчет кинетической энергии:
minKE = максимальное значение типа double
Для каждой молекулы i от 0 до popSize:
Если fitness молекулы меньше minKE:
minKE = fitness молекулы
Для каждой молекулы i от 0 до popSize:
Рассчитать кинетическую энергию молекулы KE как масштабирование fitness из диапазона от minKE до fB (лучшее глобальное решение) в диапазон от 0.0 до 1.0
molCNT = 0
Пока не остановлено:
Если случайное число меньше moleColl:
Выбрать две случайные молекулы M1 и M2
Если KE обеих молекул больше или равно β:
Выполнить синтез молекул M1 и M2
Иначе:
Выполнить межмолекулярное неэффективное столкновение между M1 и M2
Иначе:
Выбрать случайную молекулу M
Если NumHit молекулы больше α:
Выполнить разложение молекулы M
Иначе:
Выполнить столкновение молекулы M
Копировать структуры молекул из Mfilial в a
Выполнить расчет fitness для особей популяции a
Копировать структуры молекул из a в Mfilial
Инициализация: ind = -1
Для каждой молекулы i от 0 до popSize:
Если fitness молекулы больше fB:
fB = fitness молекулы
ind = i
Если ind не равно -1:
Копировать структуру молекулы в cB
Обновление молекул:
Если нет ревизии:
Для каждой молекулы i от 0 до popSize:
Обновить fitness молекулы в Mparent
Установить ревизию в true
Выход
Для каждой молекулы i от 0 до popSize:
Обновить fitness молекулы в Mfilial
В зависимости от типа молекулы (синтез, межмолекулярное неэффективное столкновение, разложение, столкновение):
Выполнить соответствующую постоперацию
Теперь, когда у нас есть псевдокод и химические операторы, описанные в первой части статьи, мы можем приступить к сборке реализации алгоритма CRO в коде.
Объявим класс "C_AO_CRO", который является наследником базового класса "C_AO" и представляет собой реализацию алгоритма Chemical Reaction Optimisation (CRO).
1. Публичные поля:
- "popSize" - размер популяции.
- "moleColl", "alpha", "beta", "molecPerturb" - параметры алгоритма.
- "params" - массив для хранения параметров алгоритма.
- "Mparent[]", "Mfilial[]" - объекты структуры "S_CRO_Agent", представляющие молекулы.
2. Методы:
- "C_AO_CRO()" - конструктор класса, инициализирующий поля класса.
- "SetParams()" - метод для установки параметров алгоритма.
- "Init()" - метод для инициализации алгоритма. Принимает минимальный и максимальный диапазоны поиска, шаг поиска и количество эпох.
- "Moving()", "Revision()" - методы, реализующие основные операции алгоритма.
3. Приватные поля и методы:
- "Synthesis()", "InterMolInefColl()", "Decomposition()", "InefCollision()" - методы, реализующие различные типы реакций.
- "PostSynthesis()", "PostInterMolInefColl()", "PostDecomposition()", "PostInefCollision()" - методы, выполняющие действия после соответствующих реакций.
- "N()" - метод для модификации компонента (координаты) структуры молекулы.
Этот класс представляет собой полную реализацию алгоритма Chemical Reaction Optimisation (CRO) и содержит все необходимые для этого данные и методы.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_CRO : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_CRO () { } C_AO_CRO () { ao_name = "CRO"; ao_desc = "Chemical Reaction Optimisation"; ao_link = "https://www.mql5.com/ru/articles/15041"; popSize = 50; //population size moleColl = 0.9; alpha = 200; beta = 0.01; molecPerturb = 0.5; ArrayResize (params, 5); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "moleColl"; params [1].val = moleColl; params [2].name = "alpha"; params [2].val = alpha; params [3].name = "beta"; params [3].val = beta; params [4].name = "molecPerturb"; params [4].val = molecPerturb; } void SetParams () { popSize = (int)params [0].val; moleColl = params [1].val; alpha = (int)params [2].val; beta = params [3].val; molecPerturb = params [4].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); S_CRO_Agent Mparent []; S_CRO_Agent Mfilial []; //---------------------------------------------------------------------------- double moleColl; int alpha; double beta; double molecPerturb; private: //------------------------------------------------------------------- bool Synthesis (int index1, int index2, int &molCNT); bool InterMolInefColl (int index1, int index2, int &molCNT); bool Decomposition (int index, int &molCNT); bool InefCollision (int index, int &molCNT); void PostSynthesis (S_CRO_Agent &mol); void PostInterMolInefColl (S_CRO_Agent &mol); void PostDecomposition (S_CRO_Agent &mol); void PostInefCollision (S_CRO_Agent &mol); void N (double &coord, int coordPos); }; //——————————————————————————————————————————————————————————————————————————————
Метод "Init" класса "C_AO_CRO" используется для инициализации переменных класса на основе переданных параметров. Вот что происходит в этом методе:
1. Метод вызывает функцию "StandardInit", которая принимает минимальный и максимальный диапазоны поиска, а также шаг поиска. Если "StandardInit" возвращает "false", то метод "Init" также возвращает "false" и завершает свою работу.
2. Затем метод изменяет размеры массивов "Mparent" и "Mfilial" до размера "popSize", который представляет собой размер популяции.
3. Далее, для каждого элемента в массивах "Mparent" и "Mfilial" вызывается метод "Init" с параметром "coords". Этот метод инициализирует поля каждого агента в популяции.
4. В конце метод возвращает "true", указывая на успешное завершение инициализации.
Этот метод выполняет начальную настройку алгоритма Chemical Reaction Optimisation (CRO) с заданными параметрами и готовит его к выполнению оптимизации.
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_CRO::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (Mparent, popSize); ArrayResize (Mfilial, popSize); for (int i = 0; i < popSize; i++) { Mparent [i].Init (coords); Mfilial [i].Init (coords); } return true; } //——————————————————————————————————————————————————————————————————————————————
Метод "Moving" класса "C_AO_CRO" используется для вызова химических операторов, которые осуществляют изменение структур молекул, тем самым перемещают молекулы в процессе оптимизации. Метод выполняет следующие действия:
1. Если "revision" равно "false", то для каждой молекулы в популяции "Mparent" инициализируются структуры случайными значениями в заданном диапазоне "rangeMin" до "rangeMax". Затем эти значения копируются в массив "a".
2. Если "revision" не равно "false", то вычисляется минимальное значение функции "f" среди всех молекул в популяции "Mparent". Затем для каждой молекулы вычисляется значение "KE" на основе масштабирования её значения функции "f", минимального значения "f" по популяции родительских молекул и лучшего глобального решения fB в диапазон от 0.0 до 1.0.
3. Далее, пока один из химических операторов не вернёт false (это означает, что в дочерней популяции закончились места для дочерних молекул), происходит следующее:
- Если случайное число меньше "moleColl", то выбираются две случайные молекулы "M1" и "M2". Если "KE" обеих молекул больше или равно "beta", то выполняется синтез (т.е., синтез выполняется для молекул не ниже заданного в параметрах относительного значения приспособленности, именно для этих целей было предварительно проведено масштабирование значений приспособленности молекул в диапазон от 0.0 до 1.0). В противном случае выполняется межмолекулярное неэффективное столкновение.
- Если случайное число больше или равно "moleColl", то выбирается одна случайная молекула "M". Если "NumHit" этой молекулы больше "alpha" (если молекула претерпела столкновений больше, чем задано в параметрах алгоритма, то молекула "распадается"), то выполняется разложение. В противном случае выполняется столкновение.
4. В конце метода структуры всех молекул в "Mfilial" копируются в массив популяции "a".
Этот метод отвечает за обновление структур молекул в алгоритме Chemical Reaction Optimisation (CRO) в соответствии с текущим состоянием системы и заданными параметрами. Метод реализует основные операции алгоритма CRO, такие как синтез, межмолекулярное неэффективное столкновение, разложение и столкновение.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mparent [i].structure [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); // Случайная структура в диапазоне от rangeMin до rangeMax Mparent [i].structure [c] = u.SeInDiSp (Mparent [i].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].c [c] = Mparent [i].structure [c]; } } return; } //---------------------------------------------------------------------------- double minKE = DBL_MAX; for (int i = 0; i < popSize; i++) { if (Mparent [i].f < minKE) minKE = Mparent [i].f; } for (int i = 0; i < popSize; i++) { Mparent [i].KE = u.Scale (Mparent [i].f, minKE, fB, 0.0, 1.0); } //---------------------------------------------------------------------------- int molCNT = 0; while (!IsStopped ()) { if (u.RNDprobab () < moleColl) { // Выбор двух случайных молекул M1 и M2 int index1 = u.RNDminusOne (popSize); int index2 = u.RNDminusOne (popSize); // Если KE ≤ β: if (Mparent [index1].KE >= beta && Mparent [index2].KE >= beta) { // Выполнить Синтез if (!Synthesis (index1, index2, molCNT)) break; } else { // Выполнить Межмолекулярное Неэффективное Столкновение if (!InterMolInefColl (index1, index2, molCNT)) break; } } else { // Выбор случайной молекулы M int index = u.RNDminusOne (popSize); // Если NumHit > α: if (Mparent [index].NumHit > alpha) { // Выполнить Разложение if (!Decomposition (index, molCNT)) break; } else { // Выполнить Столкновение if (!InefCollision (index, molCNT)) break; } } } for (int i = 0; i < popSize; i++) { ArrayCopy (a [i].c, Mfilial [i].structure); } } //——————————————————————————————————————————————————————————————————————————————
Метод "Revision" класса "C_AO_CRO" используется для обновления лучшего глобального решения и обновления состояний молекул в родительской популяции посредством выполнения химических постоператоров. Действия, выполняемые этим методом:
1. Обновление лучшего глобального решения. В цикле "for" метод проходит по всем молекулам. Если значение функции "f" текущей молекулы превышает текущее лучшее значение "fB", то обновляется "fB" и массив координат текущей молекулы копируется в массив "cB".
2. Если "revision" равно "false", то для каждой молекулы в популяции "Mparent" значение "f" устанавливается равным значению "f" из массива "a". Затем "revision" устанавливается в "true" и метод завершает работу. На этом этапе важно получить значения приспособленности родительских молекул, чтобы в последующих эпохах можно было использовать химические операторы, которые зависят от кинетической энергии (значение фитнес-функции, нормализованное до диапазона от 0.0 до 1.0).
3. Если "revision" не равно "false", то для каждой молекулы в популяции "Mfilial" значение "f" устанавливается равным значению "f" из массива "a". Затем, в зависимости от типа реакции "rType" молекулы (в которой участвовала молекула), вызывается соответствующий метод "PostSynthesis", "PostInterMolInefColl", "PostDecomposition", "PostInefCollision".
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::Revision () { //---------------------------------------------------------------------------- int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mparent [i].f = a [i].f; } } revision = true; return; } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { Mfilial [i].f = a [i].f; } switch (Mfilial [i].rType) { case synthesis: PostSynthesis (Mfilial [i]); break; case interMolecularInefColl: PostInterMolInefColl (Mfilial [i]); break; case decomposition: PostDecomposition (Mfilial [i]); break; case inefCollision: PostInefCollision (Mfilial [i]); break; } } } //——————————————————————————————————————————————————————————————————————————————
3. Результаты тестов
Алгоритм CRO (Chemical Reaction Optimization) был протестирован на тестовых функциях Hilly, Forest и Megacity. В каждом случае было выполнено десять запусков функций для каждого типа ландшафта (5, 25 и 500 функций), и были получены результаты оптимизации.
CRO|Chemical Reaction Optimisation|50.0|0.9|200.0|0.01|0.5|
=============================
5 Hilly's; Func runs: 10000; result: 0.9462894520167225
25 Hilly's; Func runs: 10000; result: 0.6611186250435438
500 Hilly's; Func runs: 10000; result: 0.2985263035668822
=============================
5 Forest's; Func runs: 10000; result: 0.8790568514481787
25 Forest's; Func runs: 10000; result: 0.584216839762206
500 Forest's; Func runs: 10000; result: 0.2114595696419046
=============================
5 Megacity's; Func runs: 10000; result: 0.7584615384615384
25 Megacity's; Func runs: 10000; result: 0.4264615384615384
500 Megacity's; Func runs: 10000; result: 0.12686153846153955
=============================
All score: 4.89245 (54.36%)
Визуализация работы тестового стенда с алгоритмом CRO демонстрирует интересные особенности этого алгоритма. Несмотря на то, что CRO иногда может застревать, что видно по продолжительным пологим участкам графика ходимости, он все же показывает достойные общие результаты.
Одним из заметных аспектов работы CRO является движение "молекул" в области поиска. На первый взгляд, это движение кажется хаотичным и напоминает броуновское движение. Однако, несмотря на внешнюю случайность, "молекулы" умудряются отыскать зону глобального оптимума. Это свидетельствует о сложной и изощренной природе алгоритма CRO, который использует принципы химических реакций для решения задач оптимизации.
В целом, алгоритм CRO представляет собой мощный инструмент оптимизации, способный справиться с различными задачами, несмотря на некоторые трудности. Его уникальные свойства и способность находить глобальные оптимумы делают его ценным инструментом в области оптимизации.
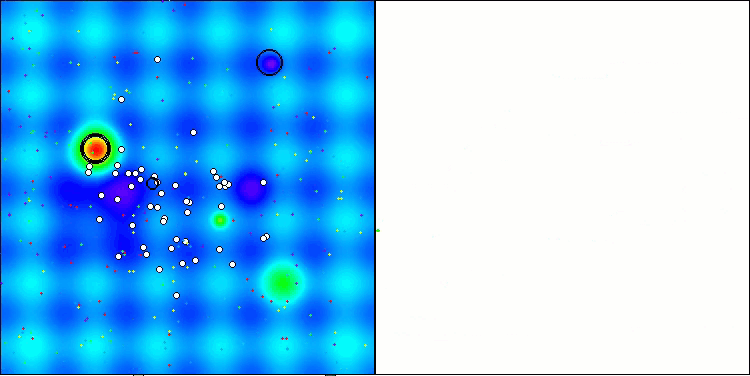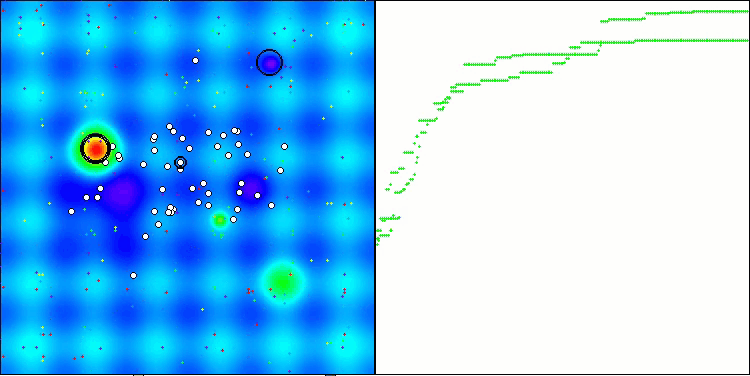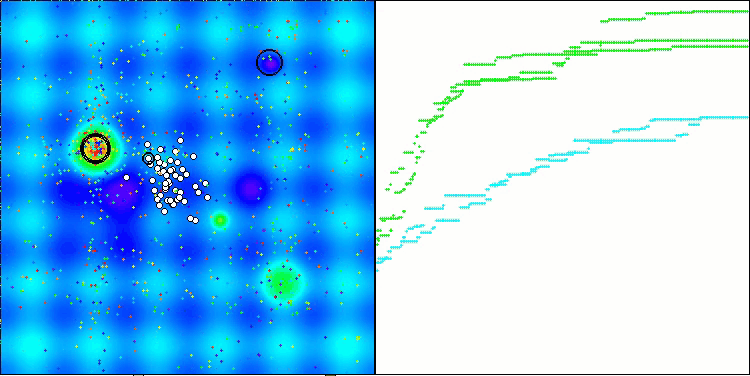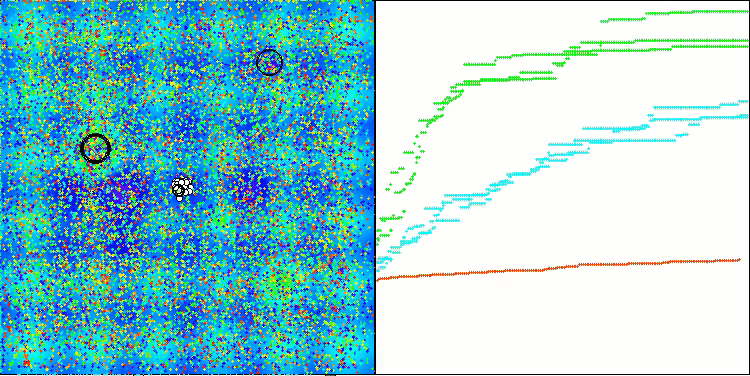
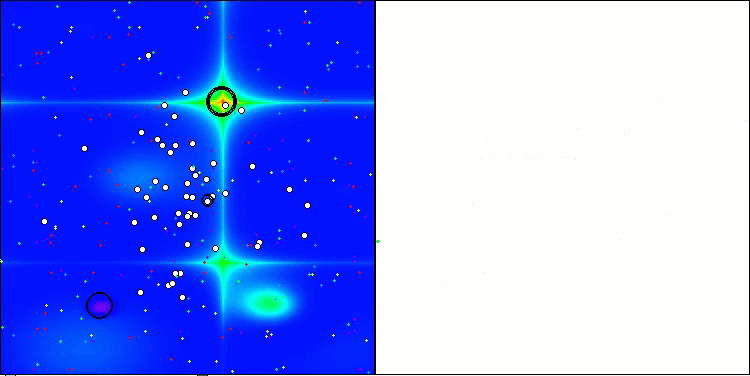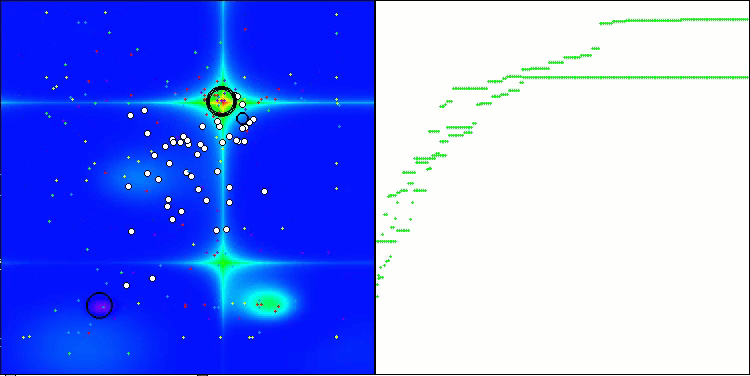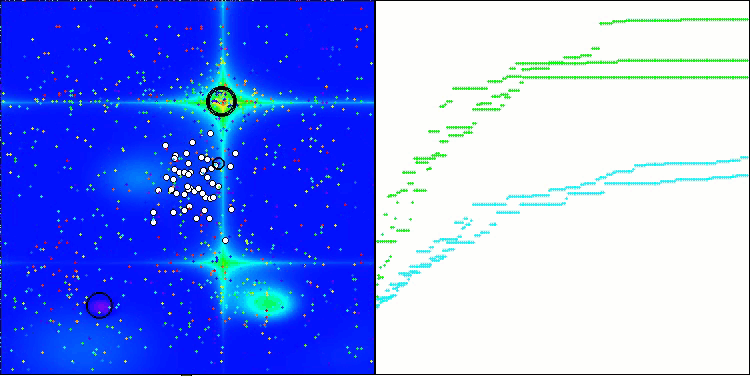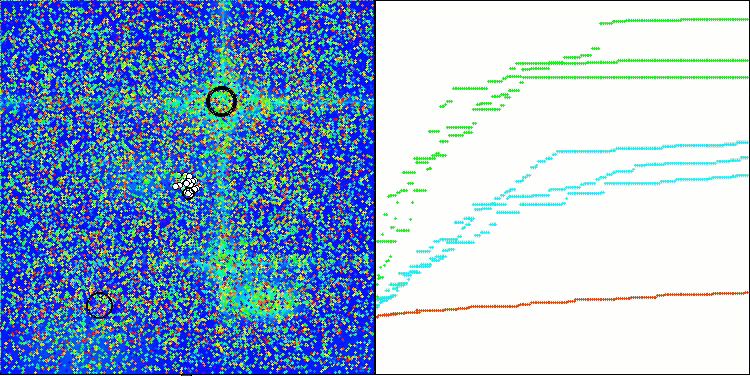
CRO на тестовой функции Hilly.
CRO на тестовой функции Forest.
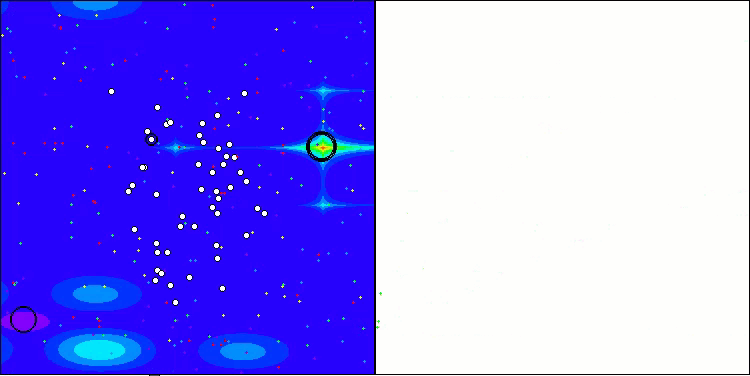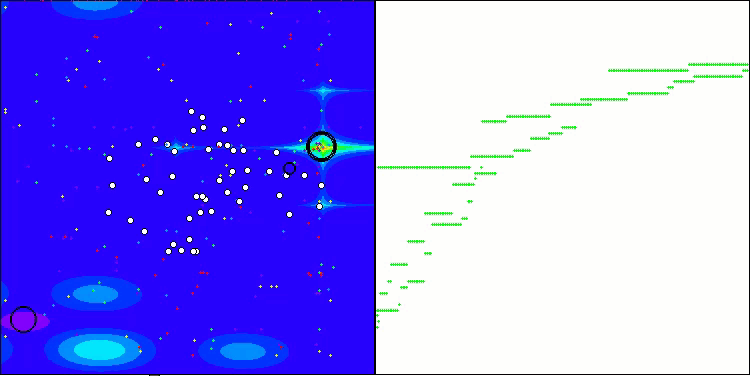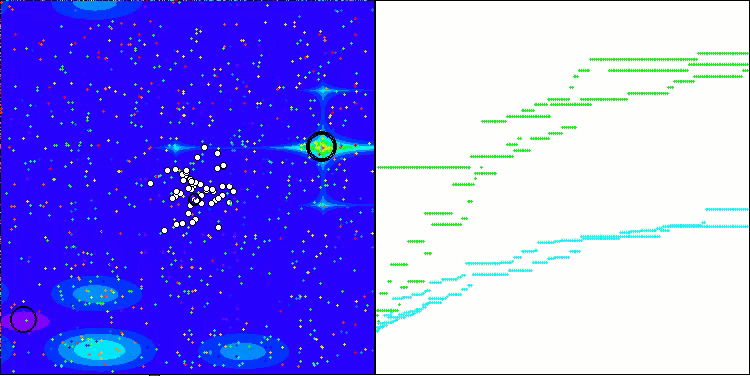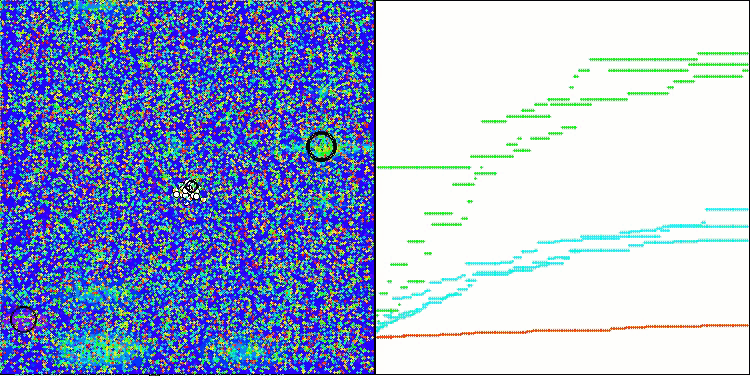
CRO на тестовой функции Megacity.
| № | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | BGA | binary genetic algorithm | 0,99989 | 0,99518 | 0,42835 | 2,42341 | 0,96153 | 0,96181 | 0,32027 | 2,24360 | 0,91385 | 0,95908 | 0,24220 | 2,11512 | 6,782 | 75,36 |
| 2 | CLA | code lock algorithm | 0,95345 | 0,87107 | 0,37590 | 2,20042 | 0,98942 | 0,91709 | 0,31642 | 2,22294 | 0,79692 | 0,69385 | 0,19303 | 1,68380 | 6,107 | 67,86 |
| 3 | (P+O)ES | (P+O) evolution strategies | 0,92256 | 0,88101 | 0,40021 | 2,20379 | 0,97750 | 0,87490 | 0,31945 | 2,17185 | 0,67385 | 0,62985 | 0,18634 | 1,49003 | 5,866 | 65,17 |
| 4 | CTA | comet tail algorithm | 0,95346 | 0,86319 | 0,27770 | 2,09435 | 0,99794 | 0,85740 | 0,33949 | 2,19484 | 0,88769 | 0,56431 | 0,10512 | 1,55712 | 5,846 | 64,96 |
| 5 | SDSm | stochastic diffusion search M | 0,93066 | 0,85445 | 0,39476 | 2,17988 | 0,99983 | 0,89244 | 0,19619 | 2,08846 | 0,72333 | 0,61100 | 0,10670 | 1,44103 | 5,709 | 63,44 |
| 6 | ESG | evolution of social groups | 0,99906 | 0,79654 | 0,35056 | 2,14616 | 1,00000 | 0,82863 | 0,13102 | 1,95965 | 0,82333 | 0,55300 | 0,04725 | 1,42358 | 5,529 | 61,44 |
| 7 | SIA | simulated isotropic annealing | 0,95784 | 0,84264 | 0,41465 | 2,21513 | 0,98239 | 0,79586 | 0,20507 | 1,98332 | 0,68667 | 0,49300 | 0,09053 | 1,27020 | 5,469 | 60,76 |
| 8 | ACS | artificial cooperative search | 0,75547 | 0,74744 | 0,30407 | 1,80698 | 1,00000 | 0,88861 | 0,22413 | 2,11274 | 0,69077 | 0,48185 | 0,13322 | 1,30583 | 5,226 | 58,06 |
| 9 | TSEA | turtle shell evolution algorithm | 0,96798 | 0,64480 | 0,29672 | 1,90949 | 0,99449 | 0,61981 | 0,22708 | 1,84139 | 0,69077 | 0,42646 | 0,13598 | 1,25322 | 5,004 | 55,60 |
| 10 | DE | differential evolution | 0,95044 | 0,61674 | 0,30308 | 1,87026 | 0,95317 | 0,78896 | 0,16652 | 1,90865 | 0,78667 | 0,36033 | 0,02953 | 1,17653 | 4,955 | 55,06 |
| 11 | CRO | chemical reaction optimisation | 0,94629 | 0,66112 | 0,29853 | 1,90593 | 0,87906 | 0,58422 | 0,21146 | 1,67473 | 0,75846 | 0,42646 | 0,12686 | 1,31178 | 4,892 | 54,36 |
| 12 | BSA | bird swarm algorithm | 0,89306 | 0,64900 | 0,26250 | 1,80455 | 0,92420 | 0,71121 | 0,24939 | 1,88479 | 0,69385 | 0,32615 | 0,10012 | 1,12012 | 4,809 | 53,44 |
| 13 | HS | harmony search | 0,86509 | 0,68782 | 0,32527 | 1,87818 | 0,99999 | 0,68002 | 0,09590 | 1,77592 | 0,62000 | 0,42267 | 0,05458 | 1,09725 | 4,751 | 52,79 |
| 14 | SSG | saplings sowing and growing | 0,77839 | 0,64925 | 0,39543 | 1,82308 | 0,85973 | 0,62467 | 0,17429 | 1,65869 | 0,64667 | 0,44133 | 0,10598 | 1,19398 | 4,676 | 51,95 |
| 15 | (PO)ES | (PO) evolution strategies | 0,79025 | 0,62647 | 0,42935 | 1,84606 | 0,87616 | 0,60943 | 0,19591 | 1,68151 | 0,59000 | 0,37933 | 0,11322 | 1,08255 | 4,610 | 51,22 |
| 16 | BSO | brain storm optimization | 0,93736 | 0,57616 | 0,29688 | 1,81041 | 0,93131 | 0,55866 | 0,23537 | 1,72534 | 0,55231 | 0,29077 | 0,11914 | 0,96222 | 4,498 | 49,98 |
| 17 | WOAm | wale optimization algorithm M | 0,84521 | 0,56298 | 0,26263 | 1,67081 | 0,93100 | 0,52278 | 0,16365 | 1,61743 | 0,66308 | 0,41138 | 0,11357 | 1,18803 | 4,476 | 49,74 |
| 18 | ACOm | ant colony optimization M | 0,88190 | 0,66127 | 0,30377 | 1,84693 | 0,85873 | 0,58680 | 0,15051 | 1,59604 | 0,59667 | 0,37333 | 0,02472 | 0,99472 | 4,438 | 49,31 |
| 19 | BFO-GA | bacterial foraging optimization - ga | 0,89150 | 0,55111 | 0,31529 | 1,75790 | 0,96982 | 0,39612 | 0,06305 | 1,42899 | 0,72667 | 0,27500 | 0,03525 | 1,03692 | 4,224 | 46,93 |
| 20 | MEC | mind evolutionary computation | 0,69533 | 0,53376 | 0,32661 | 1,55569 | 0,72464 | 0,33036 | 0,07198 | 1,12698 | 0,52500 | 0,22000 | 0,04198 | 0,78698 | 3,470 | 38,55 |
| 21 | IWO | invasive weed optimization | 0,72679 | 0,52256 | 0,33123 | 1,58058 | 0,70756 | 0,33955 | 0,07484 | 1,12196 | 0,42333 | 0,23067 | 0,04617 | 0,70017 | 3,403 | 37,81 |
| 22 | Micro-AIS | micro artificial immune system | 0,79547 | 0,51922 | 0,30861 | 1,62330 | 0,72956 | 0,36879 | 0,09398 | 1,19233 | 0,37667 | 0,15867 | 0,02802 | 0,56335 | 3,379 | 37,54 |
| 23 | COAm | cuckoo optimization algorithm M | 0,75820 | 0,48652 | 0,31369 | 1,55841 | 0,74054 | 0,28051 | 0,05599 | 1,07704 | 0,50500 | 0,17467 | 0,03380 | 0,71347 | 3,349 | 37,21 |
| 24 | SDOm | spiral dynamics optimization M | 0,74601 | 0,44623 | 0,29687 | 1,48912 | 0,70204 | 0,34678 | 0,10944 | 1,15826 | 0,42833 | 0,16767 | 0,03663 | 0,63263 | 3,280 | 36,44 |
| 25 | NMm | Nelder-Mead method M | 0,73807 | 0,50598 | 0,31342 | 1,55747 | 0,63674 | 0,28302 | 0,08221 | 1,00197 | 0,44667 | 0,18667 | 0,04028 | 0,67362 | 3,233 | 35,92 |
| 26 | FAm | firefly algorithm M | 0,58634 | 0,47228 | 0,32276 | 1,38138 | 0,68467 | 0,37439 | 0,10908 | 1,16814 | 0,28667 | 0,16467 | 0,04722 | 0,49855 | 3,048 | 33,87 |
| 27 | GSA | gravitational search algorithm | 0,64757 | 0,49197 | 0,30062 | 1,44016 | 0,53962 | 0,36353 | 0,09945 | 1,00260 | 0,32667 | 0,12200 | 0,01917 | 0,46783 | 2,911 | 32,34 |
| 28 | BFO | bacterial foraging optimization | 0,61171 | 0,43270 | 0,31318 | 1,35759 | 0,54410 | 0,21511 | 0,05676 | 0,81597 | 0,42167 | 0,13800 | 0,03195 | 0,59162 | 2,765 | 30,72 |
| 29 | ABC | artificial bee colony | 0,63377 | 0,42402 | 0,30892 | 1,36671 | 0,55103 | 0,21874 | 0,05623 | 0,82600 | 0,34000 | 0,14200 | 0,03102 | 0,51302 | 2,706 | 30,06 |
| 30 | BA | bat algorithm | 0,59761 | 0,45911 | 0,35242 | 1,40915 | 0,40321 | 0,19313 | 0,07175 | 0,66810 | 0,21000 | 0,10100 | 0,03517 | 0,34617 | 2,423 | 26,93 |
| 31 | SA | simulated annealing | 0,55787 | 0,42177 | 0,31549 | 1,29513 | 0,34998 | 0,15259 | 0,05023 | 0,55280 | 0,31167 | 0,10033 | 0,02883 | 0,44083 | 2,289 | 25,43 |
| 32 | IWDm | intelligent water drops M | 0,54501 | 0,37897 | 0,30124 | 1,22522 | 0,46104 | 0,14704 | 0,04369 | 0,65177 | 0,25833 | 0,09700 | 0,02308 | 0,37842 | 2,255 | 25,06 |
| 33 | PSO | particle swarm optimisation | 0,59726 | 0,36923 | 0,29928 | 1,26577 | 0,37237 | 0,16324 | 0,07010 | 0,60572 | 0,25667 | 0,08000 | 0,02157 | 0,35823 | 2,230 | 24,77 |
| 34 | Boids | boids algorithm | 0,43340 | 0,30581 | 0,25425 | 0,99346 | 0,35718 | 0,20160 | 0,15708 | 0,71586 | 0,27846 | 0,14277 | 0,09834 | 0,51957 | 2,229 | 24,77 |
| 35 | MA | monkey algorithm | 0,59107 | 0,42681 | 0,31816 | 1,33604 | 0,31138 | 0,14069 | 0,06612 | 0,51819 | 0,22833 | 0,08567 | 0,02790 | 0,34190 | 2,196 | 24,40 |
| 36 | SFL | shuffled frog-leaping | 0,53925 | 0,35816 | 0,29809 | 1,19551 | 0,37141 | 0,11427 | 0,04051 | 0,52618 | 0,27167 | 0,08667 | 0,02402 | 0,38235 | 2,104 | 23,38 |
| 37 | FSS | fish school search | 0,55669 | 0,39992 | 0,31172 | 1,26833 | 0,31009 | 0,11889 | 0,04569 | 0,47467 | 0,21167 | 0,07633 | 0,02488 | 0,31288 | 2,056 | 22,84 |
| 38 | RND | random | 0,52033 | 0,36068 | 0,30133 | 1,18234 | 0,31335 | 0,11787 | 0,04354 | 0,47476 | 0,25333 | 0,07933 | 0,02382 | 0,35648 | 2,014 | 22,37 |
| 39 | GWO | grey wolf optimizer | 0,59169 | 0,36561 | 0,29595 | 1,25326 | 0,24499 | 0,09047 | 0,03612 | 0,37158 | 0,27667 | 0,08567 | 0,02170 | 0,38403 | 2,009 | 22,32 |
| 40 | CSS | charged system search | 0,44252 | 0,35454 | 0,35201 | 1,14907 | 0,24140 | 0,11345 | 0,06814 | 0,42299 | 0,18333 | 0,06300 | 0,02322 | 0,26955 | 1,842 | 20,46 |
| 41 | EM | electroMagnetism-like algorithm | 0,46250 | 0,34594 | 0,32285 | 1,13129 | 0,21245 | 0,09783 | 0,10057 | 0,41085 | 0,15667 | 0,06033 | 0,02712 | 0,24412 | 1,786 | 19,85 |
Выводы
На основании предоставленной таблицы и результатов, можно сделать следующие выводы о производительности алгоритма Chemical Reaction Optimization (CRO):
1. CRO показывает отличные результаты на тестовой функции Hilly. С 5 параметрами результат составил около 0.95, с 25 параметрами - около 0.66, и с 500 параметрами - около 0.30. Это указывает на то, что CRO эффективен на гладких функциях, особенно при меньшем количестве параметров.
2. На тестовой функции Forest CRO также показывает хорошие результаты. С 5 параметрами результат составил около 0.88, с 25 параметрами - около 0.58, и с 500 параметрами - около 0.21. Это говорит о том, что CRO также эффективен на функциях с "острыми" экстремумами, но испытывает некоторые сложности с поиском точечных оптимумов.
3. На тестовой функции Megacity CRO продолжает демонстрировать хорошую производительность. С 5 параметрами результат составил около 0.76, с 25 параметрами - около 0.43, и с 500 параметрами - около 0.13. Это указывает на то, что CRO эффективен на этой дискретной функции, его результаты равномерно "зелёные" по сравнению с другими алгоритмами даже тех, которые располагаются выше в таблице.
На основании предоставленной таблицы, алгоритм Chemical Reaction Optimization (CRO) показывает сильные результаты по сравнению с другими алгоритмами. В частности, на функциях Hilly, Forest и Megacity CRO демонстрирует конкурентоспособность, особенно при меньшем количестве параметров.
Алгоритм CRO занял 11 место в рейтинговой таблице. На основании цветовой градации в таблице ниже (где темно-зеленый указывает на лучшие результаты), можно сказать, что CRO в целом показывает хорошую и стабильную производительность (стабильная и равномерная окраска). Только на функции "Hilly" c 1000 параметрами результаты выглядят несколько слабее.
Алгоритм Chemical Reaction Optimization (CRO) показал себя как многообещающий подход к оптимизации. Он использует две популяции агентов (в моей реализации), которые взаимодействуют друг с другом, чтобы обеспечить разнообразие и избежать застревания в локальных оптимумах. Одной из отличительных особенностей алгоритма является использование специальных операторов, подобных химическим реакциям, декомпозиции, синтезу и другим.
В целом, алгоритм CRO представляет собой перспективный метод оптимизации, который отличается своей оригинальностью и способностью достигать высоких результатов в различных задачах оптимизации.
Важно отметить, что выбор алгоритма оптимизации должен основываться на конкретной задаче и требованиях к производительности, и наша рейтинговая таблица поможет в этом. Благодаря переработке авторского варианта реализации алгоритма CRO к наследованию от класса C_AO, принятому нами для популяционных алгоритмов, этот интересный алгоритм можно применять для задач оптимизации в общем виде.
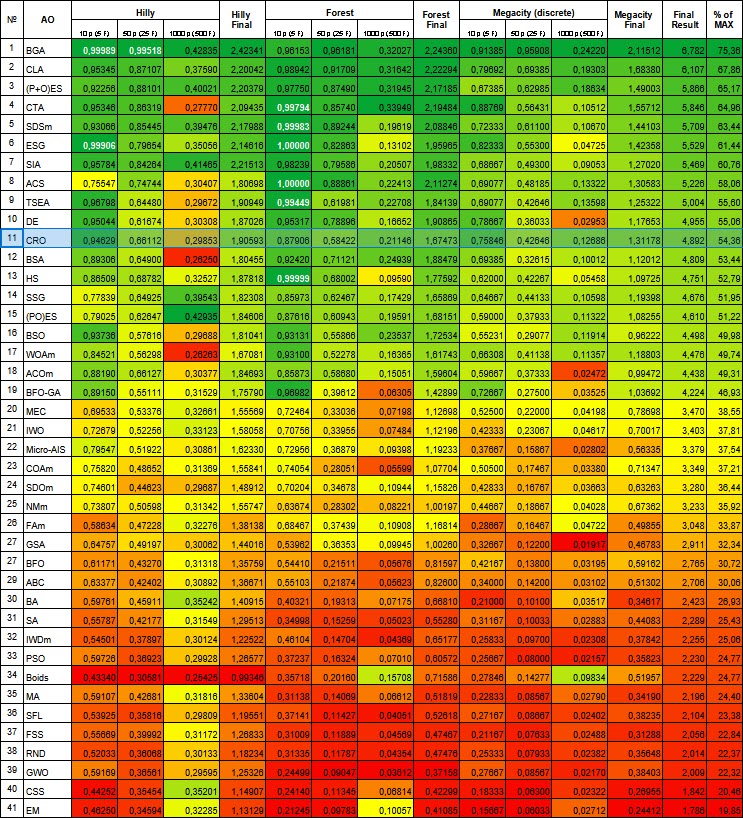
Рисунок 1. Цветовая градация алгоритмов по соответствующим тестам. Белым цветом подсвечены результаты больше или равные 0.99
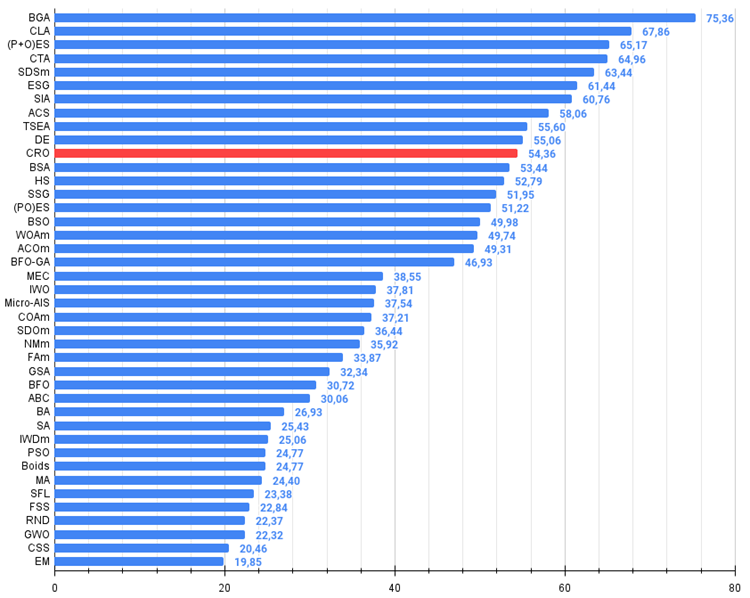
Рисунок 2. Гистограмма результатов тестирования алгоритмов (по шкале от 0 до 100, чем больше, тем лучше,
где 100 - максимально возможный теоретический результат, в архиве скрипт для расчета рейтинговой таблицы)
Общие плюсы и минусы алгоритма оптимизации химическими реакциями (CRO):
Плюсы:
- Хорошая сходимость на различных типах функций.
- Очень быстрый, несмотря на сложную архитектуру.
- Хорошая масштабируемость.
Минусы:
- Иногда застревает в локальных экстремумах.
К статье прикреплён архив с актуальными версиями кодов алгоритмов. Автор статьи не несёт ответственности за абсолютную точность в описании канонических алгоритмов, во многие из них были внесены изменения для улучшения поисковых возможностей. Выводы и суждения, представленные в статьях, основываются на результатах проведённых экспериментов.
 Разработка системы репликации (Часть 40): Начало второй фазы (I)
Разработка системы репликации (Часть 40): Начало второй фазы (I)
 Разработка системы репликации (Часть 39): Прокладываем путь (III)
Разработка системы репликации (Часть 39): Прокладываем путь (III)
 Нейросети — это просто (Часть 95): Снижение потребления памяти в моделях Transformer
Нейросети — это просто (Часть 95): Снижение потребления памяти в моделях Transformer
 Алгорим оптимизации химическими реакциями — Chemical reaction optimisation, CRO (Часть I): Химия процессов в оптимизации
Алгорим оптимизации химическими реакциями — Chemical reaction optimisation, CRO (Часть I): Химия процессов в оптимизации
- Бесплатные приложения для трейдинга
- 8 000+ сигналов для копирования
- Экономические новости для анализа финансовых рынков
Вы принимаете политику сайта и условия использования