Алгорим оптимизации химическими реакциями — Chemical reaction optimisation, CRO (Часть I): Химия процессов в оптимизации
Содержание:
1. Введение
Оптимизация химическими реакциями (CRO) - это захватывающий и инновационный метод, вдохновленный самой сутью химических превращений. Представьте себе, что вы наблюдаете за танцем молекул, где каждое движение и столкновение играет ключевую роль в решении сложных задач. Этот метод искусно сплетает воедино принципы сохранения энергии, разложения и синтеза молекул, создавая гибкий и адаптивный подход к оптимизации.
CRO — это техника, свободно связывающая химические реакции с оптимизацией, и использующая общие принципы молекулярного взаимодействия, которые определяются первыми двумя законами термодинамики. Алгоритм Chemical Reaction Optimization (CRO) был предложен и опубликован Lam и Li в 2012 году.
Первый закон термодинамики (закон сохранения энергии) гласит, что энергия не может быть создана или уничтожена, энергия может превращаться из одной формы в другую и передаваться от одной сущности к другой. В контексте CRO, химическая реагирующая система состоит из веществ и окружающей среды, и каждая частица обладает потенциальной и кинетической энергией.
Второй закон термодинамики гласит, что энтропия системы имеет тенденцию увеличиваться, где энтропия - мера беспорядка, система жаждущая большей свободы. Потенциальная энергия - это энергия, сохраненная в молекуле относительно ее молекулярной конфигурации. Когда потенциальная энергия молекул высвобождается, превращаясь в кинетическую, система становится все более хаотичной. Но именно в этом хаосе CRO находит свою силу, улавливая и направляя энергетические потоки к оптимальному решению.
Например, когда молекулы с более высокой кинетической энергией (преобразованной из потенциальной энергии) движутся быстрее, система становится более неупорядоченной, и ее энтропия увеличивается. Таким образом, все реагирующие системы стремятся достичь состояния равновесия, при котором потенциальная энергия снижается до минимума. В CRO мы улавливаем этот феномен, преобразуя потенциальную энергию в кинетическую и постепенно теряя энергию химических молекул в окружающей среде.
Химическая система, подверженная химической реакции, нестабильна и, обладая избыточной энергией, старается избавиться от нее и стабилизироваться. Система состоит из молекул, которые являются наименьшими частицами соединения и классифицируются по различным видам на основе основных химических свойств.
Химические реакции образуют сложный танец, где молекулы сталкиваются, создают и разрывают связи, меняя свои структуры. Каждый шаг этого танца - это последовательность подреакций, ведущих к более стабильным продуктам с минимальной энергией. Молекулы хранят энергию в своих химических связях, и даже незначительные изменения в их структуре могут привести к удивительным трансформациям, которые приводят к образованию более стабильных продуктов. Молекулярная структура влияет на химическое поведение соединения, и даже незначительные изменения в структуре могут привести к значительным различиям в химических свойствах.
Химические реакции инициируются столкновениями молекул, которые могут быть унимолекулярными (с внешними веществами) или бимолекулярными (с другими молекулами). Эти столкновения могут быть как эффективными, так и неэффективными, в зависимости от таких факторов, как энергия активации и стерические эффекты. Таким образом молекулы танцуют, переплетаясь и разделяясь, создавая новые формы и структуры.
Важно отметить, что химические реакции часто, но не всегда, приводят к образованию более устойчивых продуктов с более низкой энергией Гиббса. Этот процесс осуществляется через последовательные и/или параллельные стадии, включающие образование промежуточных соединений и прохождение через переходные состояния. Понимание этих процессов важно для поиска оптимальных условий проведения химических реакций и выяснения механизмов превращения химических соединений.
Также химический синтез может происходить как в результате эффективных, так и неэффективных столкновений реагентов. Эффективность столкновений зависит от факторов, таких как энергия активации, стерические эффекты и т.д. Межмолекулярный синтез, в отличие от внутримолекулярного, приводит к более значительным изменениям в молекулярных структурах.
Алгоритм CRO как виртуозный хореограф, который использует законы химии, словно ноты, для создания невероятно сложных и изящных оптимизационных решений. Теперь разберем все по полочкам, от сложного к простому.
2.Реализация химических операторов
Основной единицей в CRO является "молекула", где в популяции каждая из них имеет определенные характеристики, такие как "потенциальная и кинетическая энергии", "молекулярные структуры" и т.д. Элементарные реакции определяют взаимодействия между молекулами. Мы можем определить класс, в котором поля данных представляют характеристики молекулы, а методы описывают элементарные реакции.
Одна из основных концепций в CRO - это сохранение энергии. Этот принцип гарантирует, что общая энергия в системе остается постоянной на протяжении всего процесса оптимизации. Сохранение энергии определяет переходы между различными состояниями молекул во время реакций, поддерживая баланс энергий, влияющий на поиск оптимальных решений.
Используя манипулируемые агенты (молекулы), элементарные реакции и концепцию сохранения энергии, CRO предлагает гибкий и адаптивный метод для решения сложных задач оптимизации. Возможность настройки операторов, динамическое изменение размера популяции и интеграция различных атрибутов делают CRO многообещающим подходом в области метаэвристик. Его уникальные особенности и гибкость позволяют исследователям искать новые возможности в оптимизации.
В алгоритме CRO используются следующие операторы:
1. Межмолекулярное неэффективное столкновение (InterMolecularIneffectiveCollision). Этот оператор моделирует процесс, при котором две молекулы сталкиваются, но остаются целыми, и их структуры немного изменяются. Это позволяет алгоритму проводить локальный поиск в окрестности текущих решений.
2. Декомпозиция (Decomposition). Этот оператор моделирует процесс, при котором молекула сталкивается со стенкой и распадается на две новые молекулы. Это позволяет алгоритму исследовать новые области пространства решений.
3. Внутримолекулярное неэффективное столкновение (IntraMolecularReaction). Этот оператор моделирует процесс, при котором молекула сталкивается со стенкой и остается целой, но ее структура немного изменяется. Это позволяет алгоритму проводить локальный поиск в окрестности текущего решения.
4. Синтез (Synthesis). Этот оператор моделирует процесс, при котором две молекулы сталкиваются и объединяются в одну новую молекулу. Это позволяет алгоритму комбинировать хорошие решения для создания потенциально лучших решений.
Каждый из этих операторов играет важную роль в алгоритме CRO, позволяя ему исследовать пространство поиска и находить оптимальные решения. Их совместная работа обеспечивает баланс между исследованием (поиском в новых областях пространства поиска) и эксплуатацией (усовершенствованием текущих лучших решений).
Перейдем непосредственно к рассмотрению каждого отдельного химического оператора в алгоритме (поиск глобального минимума):
Алгоритм #1: On-wall Ineffective Collision (Неэффективное столкновение со стенкой)
1. Входные данные: молекула Mω
2. Генерация нового положения молекулы: ω = N(ω)
3. Расчет потенциальной энергии нового положения: PEω = f(ω)
4. Увеличение счетчика столкновений: NumHitω = NumHitω + 1
5. Если потенциальная энергия нового положения + кинетическая энергия ≥ потенциальная энергия текущего положения, то:
6. Генерация случайного числа a в диапазоне [KELossRate, 1]
7. Обновление кинетической энергии: KEω = (PEω − PEω + KEω) × a
8. Обновление буфера: buffer = buffer + (PEω − PEω + KEω) × (1 − a)
9. Сохранение текущего положения и энергий
10. Если потенциальная энергия нового положения < минимальной потенциальной энергии, то обновление минимальных значений
11. Конец условия
Алгоритм #2: Decomposition (Декомпозиция)
1. Входные данные: молекула Mω
2. Создание двух новых молекул Mω1 и Mω2
3. Получение положений для новых молекул: ω1 и ω2 из ω
4. Расчет потенциальной энергии для новых молекул: PEω1 = f(ω1) и PEω2 = f(ω2)
5. Если потенциальная энергия текущего положения + кинетическая энергия ≥ суммарной потенциальной энергии новых положений, то:
6. Расчет энергии для декомпозиции: Edec = PEω + KEω − (PEω1 + PEω2)
7. Переход к шагу 13
8. Иначе:
9. Генерация случайных чисел δ1, δ2 в диапазоне [0, 1]
10. Расчет энергии для декомпозиции: Edec = PEω + KEω + δ1δ2 × buffer − (PEω1 + PEω2)
11. Если Edec ≥ 0, то:
12. Обновление буфера
13. Генерация случайного числа δ3 в диапазоне [0, 1]
14. Распределение кинетической энергии между новыми молекулами
15. Сохранение минимальных значений для каждой новой молекулы
16. Уничтожение текущей молекулы
17. Иначе:
18. Увеличение счетчика столкновений
19. Уничтожение новых молекул
20. Конец условия
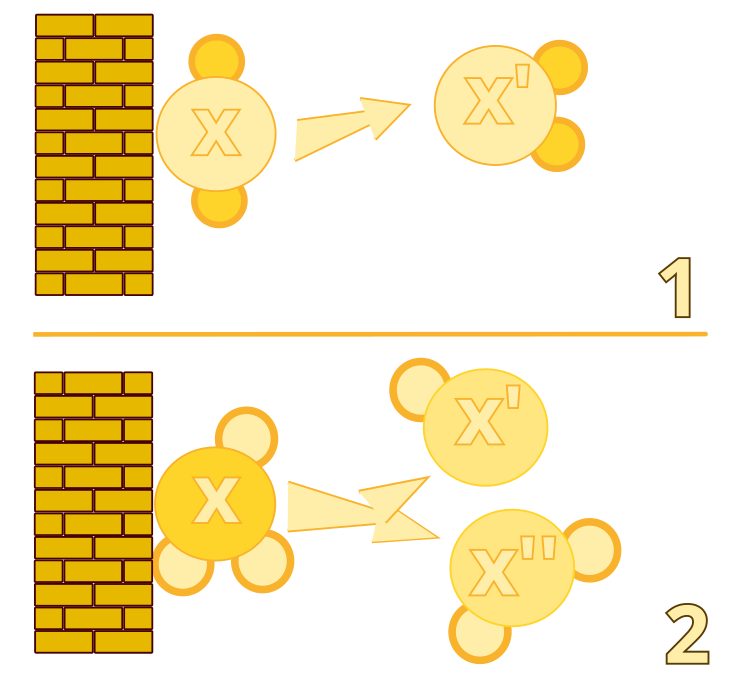
Рисунок 1. Неэффективное столкновение со стенкой: алгоритм #1. Декомпозиция: алгоритм #2.
Алгоритм #3: Intermolecular Ineffective Collision (Межмолекулярное неэффективное столкновение)
1. Входные данные: молекулы Mω1 и Mω2
2. Генерация новых положений для молекул: ω1 = N(ω1) и ω2 = N(ω2)
3. Расчет потенциальной энергии для новых положений: PEω1 = f(ω1) и PEω2 = f(ω2)
4. Увеличение счетчиков столкновений: NumHitω1 = NumHitω1 + 1 и NumHitω2 = NumHitω2 + 1
5. Расчет энергии межмолекулярного взаимодействия: Einter = (PEω1 + PEω2 + KEω1 + KEω2) − (PEω1 + PEω2)
6. Если Einter ≥ 0, то:
7. Генерация случайного числа δ4 в диапазоне [0, 1]
8. Распределение кинетической энергии между молекулами: KEω1 = Einter × δ4 и KEω2 = Einter × (1 − δ4)
9. Обновление положений и энергий молекул
10. Если потенциальная энергия нового положения меньше минимальной потенциальной энергии, то обновление минимальных значений для каждой молекулы
11. Конец условия
Алгоритм #4: Synthesis (Синтез)
1. Входные данные: молекулы Mω1 и Mω2
2. Создание новой молекулы Mω
3. Получение положения для новой молекулы: ω из ω1 и ω2
4. Расчет потенциальной энергии для новой молекулы: PEω = f(ω)
5. Если суммарная потенциальная энергия и кинетическая энергия для новой молекулы больше или равна суммарной потенциальной энергии и кинетической энергии для исходных молекул, то:
6. Распределение лишней кинетической энергии на новую молекулу: KEω = (PEω1 + PEω2 + KEω1 + KEω2) − PEω
7. Обновление минимальных значений для новой молекулы
8. Уничтожение исходных молекул
9. Иначе:
10. Увеличение счетчиков столкновений для исходных молекул
11. Уничтожение новой молекулы
12. Конец условия
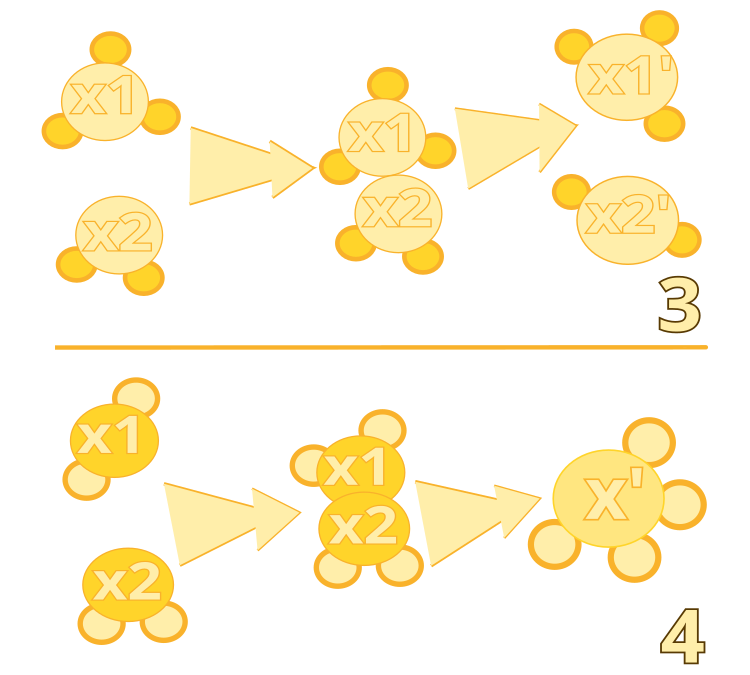
Рисунок 2. Неэффективное межмолекулярное столкновение: алгоритм #3. Синтез: алгоритм #4.
Предложенное описание алгоритма оптимизации химическими реакциями (CRO) отражает авторское видение данного подхода. Однако, в нем не учтены некоторые важные моменты, которые могут значительно повлиять на производительность и поисковые возможности алгоритма.
Алгоритм предполагает сохранение энергии в замкнутом пространстве и переход от одного вида энергии к другому. Однако, авторы не раскрывают соответствие численных показателей энергии и значение целевой функции (fitness function). Очевидно, что роль показателя приспособленности авторы определяют как потенциальную энергию, а кинетическая энергия выполняет роль механизма для компенсации снижения потенциальной энергии (сумма всех энергий должна быть константой в идеале).
Например, если критерием задачи оптимизации является профит фактор, то значения целевой функции будут колебаться в небольшом диапазоне по сравнению с задачей, где в качестве критерия используется баланс. В этом случае мы видим, что значения целевой функции будут варьироваться в зависимости от конкретного критерия оптимизации, но авторы используют при этом в качестве внешнего параметра алгоритма константы, которые никак не могут быть сопоставлены с энергиями, вычисляемыми в алгоритме.
Таким образом, алгоритм CRO в авторском варианте, без доработок, имеет существенно ограниченный список задач, в которых он может быть применен, то есть не является универсальным. В рамках данной серии статей мы рассматриваем исключительно универсальные алгоритмы, применимые к задачам оптимизации в общем виде, и если какой-либо алгоритм не позволяет этого сделать, мы обычно его модифицируем и приводим к единому общему виду.
Второй момент - авторский алгоритм предусматривает расчет фитнес-функции неупорядоченно, в разных участках логики, что неизбежно приведёт к проблемам при использовании в практических задачах и интеграции в проекты. Это мы также исправим, для этого понадобится разбить химические операторы на две части: собственно сами операторы и так называемые "постоператоры", первые из которых производят модификацию положения молекул, а вторые – необходимые действия с молекулами после вычисления их приспособленности.
Третий момент, алгоритм авторов предусматривает динамический размер популяции молекул, появляются новые молекулы и часть старых уничтожается. При этом не предусмотрены никакие механизмы регулирования размера популяции, и он может варьироваться в очень широких пределах. Так, эксперименты показали, что при определённых настройках внешних параметров, популяция может разрастаться из начальных 50 молекул до более тысячи. Эту проблему мы также решили путём последовательного заполнения популяции молекулами в результате выполнения операторов, используя простой счетчик и сохранение индекса родительских молекул в родительской популяции. Это позволяет сохранять константой размер популяции и при этом избавиться от необходимости выполнять операцию удаления молекул - дочерние молекулы при выполнении условий просто заменяют собой соответствующие родительские молекулы.
Эти изменения позволили привести алгоритм CRO к виду, пригодному для решения задач оптимизации в общем виде, сделать удобной возможность интеграции алгоритма в проекты и при этом сохранить общую оригинальную концепцию химических реакций. Алгоритм CRO подробно описан, и энтузиасты могут при желании попытаться реализовать учёт потенциальной и кинетической энергий в алгоритме, а я отказался от этих процедур.
Далее переходим к написанию кода. Опишем в перечислении типы реакций, чтобы мы могли выбрать соответствующий постоператор после реакций, и структуру молекул.
Перечисление "E_ReactionType":
1. "synthesis" - это синтез, процесс, при котором две молекулы объединяются для образования новой молекулы.
2. "interMolecularInefColl" - это межмолекулярное неэффективное столкновение, процесс, при котором молекулы сталкиваются, но не реагируют друг с другом.
3. "decomposition" - это разложение, процесс, при котором сложная молекула разбивается на более простые составляющие.
4. "inefCollision" - это неэффективное столкновение со стенкой, при котором изменяется структура молекулы.
Определяет структуру "S_CRO_Agent", которая представляет собой модель молекулы в контексте алгоритма. Давайте разберем, какие поля содержит следующая структура:
- structure[] - массив, представляющий структуру молекулы.
- NumHit - счетчик количества "ударов" или взаимодействий молекулы.
- indMolecule_1" и "indMolecule_2 - индексы взаимодействующих молекул.
- KE - переменная, представляющая кинетическую энергию молекулы.
- f - фитнес-функция (приспособленность молекулы).
- rType - переменная типа "E_ReactionType", тип реакции, в которой участвует молекула.
"Init" - метод структуры, инициализирует поля. Он принимает целочисленный аргумент "coords", который используется для изменения размера массива "structure" с помощью функции "ArrayResize". Значения "NumHit", "indMolecule_1", "indMolecule_2", "f", и "KE" инициализируются нулями или "-DBL_MAX".
Этот код представляет собой базовую структуру данных для молекул в алгоритме CRO и инициализирует их поля при создании новой молекулы.
enum E_ReactionType
{
synthesis,
interMolecularInefColl,
decomposition,
inefCollision
}; // Структура молекулы struct S_CRO_Agent { double structure []; int NumHit; int indMolecule_1; int indMolecule_2; double KE; double f; E_ReactionType rType; // Метод инициализации void Init (int coords) { ArrayResize (structure, coords); NumHit = 0; indMolecule_1 = 0; indMolecule_2 = 0; f = -DBL_MAX; KE = -DBL_MAX; } };
Метод "InefCollision" класса "C_AO_CRO" представляет собой процесс неэффективного столкновения, который включает в себя создание новой молекулы путем смещения одной родительской молекулы. Вот что происходит в этом методе:
1. Метод принимает индекс родительской молекулы "index" и ссылку на счетчик молекул "molCNT". Если "molCNT" больше или равен размеру популяции "popSize", метод возвращает "false" и завершает работу.
2. Метод определяет индекс новой молекулы "index1_", которая будет создана в результате столкновения.
3. Структура родительской молекулы копируется в структуру новой молекулы.
4. Далее, для каждой координаты новой молекулы выполняется функция "N", которая генерирует новые значения координат в окрестности старых значений.
5. Новые значения координат корректируются с помощью функции "SeInDiSp", чтобы они оставались в заданном диапазоне "rangeMin" до "rangeMax".
6. Затем устанавливаются значения полей "indMolecule_1", "rType" и "NumHit" новой молекулы. "indMolecule_1" сохраняет индекс родительской молекулы, "rType" устанавливается в "inefCollision", а "NumHit" обнуляется.
7. В конце метода увеличивается счетчик молекул "molCNT" на 1, и метод возвращает "true".
//—————————————————————————————————————————————————————————————————————————————— // Неэффективное столкновение. Получение новой молекулы путём смещения одной родительской. bool C_AO_CRO::InefCollision (int index, int &molCNT) { if (molCNT >= popSize) return false; int index1_ = molCNT; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; //сохраним индекс родительской молекулы Mfilial [index1_].rType = inefCollision; Mfilial [index1_].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
Метод "PostInefCollision" в классе "C_AO_CRO" предназначен для обработки результатов неэффективного столкновения молекулы "mol" с другими молекулами в моделировании химических реакций. Метод выполняет следующие действия:
1. Объявляется переменная "ind" и инициализируется значением "indMolecule_1" из объекта "mol".
2. Условное выражение проверяет, превышает ли значение "f" объекта "mol" значение "f" родительской молекулы с индексом "ind".
3. Если условие истинно, то структура "mol" копируется в структуру родительской молекулы с индексом "ind".
4. Значение "f" родительской молекулы обновляется значением "f" из "mol".
5. Счетчик "NumHit" родительской молекулы сбрасывается до нуля.
6. Если условие "mol.f > Mparent [ind].f" ложно, то счетчик "NumHit" родительской молекулы увеличивается на единицу.
В общем, этот метод обновляет структуру и значение фитнес-функции родительской молекулы на основе результатов неэффективного столкновения. Если новая структура "mol" приводит к улучшению приспособленности, она заменяет структуру родительской молекулы и счетчик "NumHit" сбрасывается. В противном случае, увеличивается счетчик "NumHit" родительской молекулы.
//—————————————————————————————————————————————————————————————————————————————— // Обработка результатов неэффективного столкновения. void C_AO_CRO::PostInefCollision (S_CRO_Agent &mol) { int ind = mol.indMolecule_1; if (mol.f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, mol.structure); Mparent [ind].f = mol.f; Mparent [ind].NumHit = 0; } else { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
Метод "Decomposition" класса "C_AO_CRO" представляет собой процесс разложения, который включает в себя создание двух новых молекул путем разложения одной родительской молекулы. Вот что происходит в этом методе:
1. Метод принимает индекс родительской молекулы "index" и ссылку на счетчик молекул "molCNT". Если "molCNT" больше или равен "popSize - 1", метод возвращает "false" и завершает работу.
2. Затем метод определяет индексы двух новых молекул "index1_" и "index2_", которые будут созданы в результате разложения.
3. Структура родительской молекулы копируется в структуры новых молекул.
4. Далее, для каждой координаты новых молекул выполняется функция "N", которая генерирует новые значения координат в окрестности старых значений. Это делается отдельно для первой половины координат для "index1_" первой дочерней молекулы и второй половины координат для "index2_" второй молекулы соответственно.
5. Новые значения координат корректируются с помощью функции "SeInDiSp", чтобы они оставались в заданном диапазоне "rangeMin" до "rangeMax".
6. Затем устанавливаются значения полей "indMolecule_1", "indMolecule_2", "rType" и "NumHit" новых молекул. "indMolecule_1" и "indMolecule_2" сохраняют индексы родительской и сестринской молекул соответственно, "rType" устанавливается в "decomposition", а "NumHit" обнуляется.
7. В конце метода увеличивается счетчик молекул "molCNT" на 2, и метод возвращает "true".
//—————————————————————————————————————————————————————————————————————————————— // Разложение. Получение новых двух молекул путём разложения одной родительской. bool C_AO_CRO::Decomposition (int index, int &molCNT) { if (molCNT >= popSize - 1) return false; // Создание двух новых молекул M_ω'_1 и M_ω'_2 из M_ω int index1_ = molCNT; int index2_ = molCNT + 1; ArrayCopy (Mfilial [index1_].structure, Mparent [index].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index].structure); for (int c = 0; c < coords / 2; c++) { N (Mfilial [index1_].structure [c], c); Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } for (int c = coords / 2; c < coords; c++) { N (Mfilial [index2_].structure [c], c); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index; //сохраним индекс родительской молекулы Mfilial [index1_].indMolecule_2 = index2_; //сохраним индекс второй дочерней молекулы Mfilial [index1_].rType = decomposition; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index1_; //сохраним индекс первой дочерней молекулы Mfilial [index2_].indMolecule_2 = -1; //пометим молекулу, чтобы не обрабатывать её дважды Mfilial [index2_].rType = decomposition; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
Метод "PostDecomposition" класса "C_AO_CRO" обрабатывает результаты разложения молекулы. В этом методе происходит следующее:
1. Метод принимает ссылку на молекулу "mol", которая была получена в результате разложения. Если "indMolecule_2" этой молекулы равен "-1" (означающий, что молекула уже обработана), метод завершает работу.
2. Затем извлекается индекс родительской молекулы "ind" и индексы двух "дочерних" молекул "index1_" и "index2_".
3. Далее проверяется, превосходит ли значение функции приспособленности "f" первой "дочерней" молекулы значения функций "f" второй "дочерней" молекулы и родительской молекулы. Если это так, то первая "дочерняя" молекула заменяет родительскую. При этом значение "NumHit" заменяемой молекулы обнуляется и устанавливается флаг "flag".
4. Если "flag" все еще "false", то проводится аналогичная проверка для второй "дочерней" молекулы.
5. Если после всех проверок "flag" все еще "false", то значение "NumHit" родительской молекулы увеличивается на 1.
Этот метод "PostDecomposition" отвечает за обновление состояний родительской молекулы после процесса разложения в алгоритме CRO.
//—————————————————————————————————————————————————————————————————————————————— // Обработка результатов разложения. void C_AO_CRO::PostDecomposition (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind = mol.indMolecule_1; int index2_ = mol.indMolecule_2; int index1_ = Mfilial [index2_].indMolecule_1; bool flag = false; if (Mfilial [index1_].f > Mfilial [index2_].f && Mfilial [index1_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index1_].structure); Mparent [ind].f = Mfilial [index1_].f; Mparent [ind].NumHit = 0; flag = true; } if (!flag) { if (Mfilial [index2_].f > Mfilial [index1_].f && Mfilial [index2_].f > Mparent [ind].f) { ArrayCopy (Mparent [ind].structure, Mfilial [index2_].structure); Mparent [ind].f = Mfilial [index2_].f; Mparent [ind].NumHit = 0; flag = true; } } if (!flag) { Mparent [ind].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
Метод "InterMolInefColl" класса "C_AO_CRO" представляет собой процесс межмолекулярного неэффективного столкновения, который включает в себя создание двух новых молекул путем изменения двух родительских молекул. Вот что происходит в этом методе:
1. Метод принимает индексы двух родительских молекул "index1" и "index2" и ссылку на счетчик молекул "molCNT". Если "molCNT" больше или равен "popSize - 1", метод возвращает "false" и завершает работу.
2. Затем метод определяет индексы двух новых дочерних молекул "index1_" и "index2_", которые будут созданы в результате столкновения.
3. Структуры родительских молекул копируются в структуры новых молекул.
4. Далее, для каждой координаты новых молекул выполняется функция "N", которая генерирует новые значения координат в окрестности старых значений.
5. Затем новые значения координат корректируются с помощью функции "SeInDiSp", чтобы они оставались в заданном диапазоне "rangeMin" до "rangeMax".
6. Затем устанавливаются значения полей "indMolecule_1", "indMolecule_2", "rType" и "NumHit" новых молекул. "indMolecule_1" и "indMolecule_2" сохраняют индексы родительских молекул, "rType" устанавливается в "interMolecularInefColl", а "NumHit" обнуляется.
7. В конце метода увеличивается счетчик молекул "molCNT" на 2, и метод возвращает "true".
//—————————————————————————————————————————————————————————————————————————————— // Межмолекулярное неэффективное столкновение. Получение новых двух молекул путём изменения двух родительских bool C_AO_CRO::InterMolInefColl (int index1, int index2, int &molCNT) { if (molCNT >= popSize - 1) return false; int index1_ = molCNT; int index2_ = molCNT + 1; // Получение молекул ArrayCopy (Mfilial [index1_].structure, Mparent [index1].structure); ArrayCopy (Mfilial [index2_].structure, Mparent [index2].structure); // Генерация новых молекул ω'_1 = N(ω1) и ω'_2 = N(ω2) в окрестности ω1 и ω2 for (int c = 0; c < coords; c++) { N (Mfilial [index1_].structure [c], c); N (Mfilial [index2_].structure [c], c); } for (int c = 0; c < coords; c++) { Mfilial [index1_].structure [c] = u.SeInDiSp (Mfilial [index1_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); Mfilial [index2_].structure [c] = u.SeInDiSp (Mfilial [index2_].structure [c], rangeMin [c], rangeMax [c], rangeStep [c]); } Mfilial [index1_].indMolecule_1 = index1; //сохраним индекс первой родительской молекулы Mfilial [index1_].indMolecule_2 = index2_; //сохраним индекс второй дочерней молекулы Mfilial [index1_].rType = interMolecularInefColl; Mfilial [index1_].NumHit = 0; Mfilial [index2_].indMolecule_1 = index2; //сохраним индекс второй родительской молекулы Mfilial [index2_].indMolecule_2 = -1; //пометим молекулу, чтобы не обрабатывать её дважды Mfilial [index2_].rType = interMolecularInefColl; Mfilial [index2_].NumHit = 0; molCNT += 2; return true; } //——————————————————————————————————————————————————————————————————————————————
Метод "PostInterMolInefColl" класса "C_AO_CRO" обрабатывает результаты межмолекулярного неэффективного столкновения. Работа этого метода заключается в следующем:
1. Метод принимает ссылку на молекулу "mol", которая была получена в результате столкновения. Если "indMolecule_2" этой молекулы равен "-1" (означающий, что молекула уже обработана), метод завершает работу.
2. Затем извлекаются индексы двух родительских молекул "ind1" и "ind2".
3. Далее проверяется, превосходит ли сумма значений приспособленности "f" новой молекулы и ее "сестры" сумму значений приспособленности "f" обеих родительских молекул. Если это так, то новые молекулы заменяют родительские. При этом значения "NumHit" заменяемых молекул обнуляются.
4. В противном случае значения "NumHit" родительских молекул увеличиваются на 1.
Этот метод "PostInterMolInefColl" отвечает за обновление состояний родительских молекул после процесса межмолекулярного неэффективного столкновения в алгоритме CRO.
//—————————————————————————————————————————————————————————————————————————————— // Обработка результатов межмолекулярного неэффективного столкновения. void C_AO_CRO::PostInterMolInefColl (S_CRO_Agent &mol) { if (mol.indMolecule_2 == -1) return; int ind1 = mol.indMolecule_1; int ind2 = Mfilial [mol.indMolecule_2].indMolecule_1; Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; if (mol.f + Mfilial [mol.indMolecule_2].f > Mparent [ind1].f + Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; ArrayCopy (Mparent [ind2].structure, Mfilial [mol.indMolecule_2].structure); Mparent [ind2].f = Mfilial [mol.indMolecule_2].f; } } //——————————————————————————————————————————————————————————————————————————————
Последний оператор химических реакций в алгоритме - метод "Synthesis" класса "C_AO_CRO" представляет собой процесс синтеза, который включает в себя создание новой молекулы путем слияния двух родительских молекул.
1. Метод принимает индексы двух родительских молекул "index1" и "index2" и ссылку на счетчик молекул "molCNT". Если "molCNT" больше или равен размеру популяции "popSize", метод возвращает "false" и завершает работу.
2. В цикле для каждой координаты новой молекулы выполняется следующее: если случайное число меньше 0.5, то координата получает значение соответствующей координаты первой родительской молекулы "Mparent[index1].structure[i]", иначе - значение соответствующей координаты второй родительской молекулы "Mparent[index2].structure[i]".
3. Затем устанавливаются значения полей "indMolecule_1", "indMolecule_2", "rType" и "NumHit" новой молекулы. "indMolecule_1" и "indMolecule_2" сохраняют индексы родительских молекул, "rType" устанавливается в "synthesis", а "NumHit" обнуляется.
4. В конце метода увеличивается счетчик молекул "molCNT", и метод возвращает "true".
//—————————————————————————————————————————————————————————————————————————————— // Синтез. Получение новой молекулы путём слияния двух родительских bool C_AO_CRO::Synthesis (int index1, int index2, int &molCNT) { if (molCNT >= popSize) return false; // Создание новой молекулы M_ω' из M_ω1 и M_ω2 for (int i = 0; i < coords; i++) { if (u.RNDprobab () < 0.5) Mfilial [molCNT].structure [i] = Mparent [index1].structure [i]; else Mfilial [molCNT].structure [i] = Mparent [index2].structure [i]; } Mfilial [molCNT].indMolecule_1 = index1; //сохраним индекс первой родительской молекулы Mfilial [molCNT].indMolecule_2 = index2; //сохраним индекс второй родительской молекулы Mfilial [molCNT].rType = synthesis; Mfilial [molCNT].NumHit = 0; molCNT++; return true; } //——————————————————————————————————————————————————————————————————————————————
Метод "PostSynthesis" класса "C_AO_CRO" обрабатывает результаты синтеза. Вот что в нем происходит:
1. Метод принимает ссылку на молекулу "mol", которая была получена в результате синтеза. Из этой молекулы извлекаются индексы двух родительских молекул "ind1" и "ind2".
2. Затем проверяется, превосходит ли значение приспособленности "f" новой молекулы значения приспособленности "f" обеих родительских молекул. Если это так, то новая молекула заменяет ту из родительских молекул, у которой значение "f" меньше. При этом значение "NumHit" заменяемой молекулы обнуляется.
3. В противном случае, значения "NumHit" обеих родительских молекул увеличиваются на 1.
Этот метод "PostSynthesis" отвечает за обновление состояний родительских молекул после процесса синтеза в алгоритме Chemical Reaction Optimisation (CRO).
//—————————————————————————————————————————————————————————————————————————————— // Обработка результатов синтеза. void C_AO_CRO::PostSynthesis (S_CRO_Agent &mol) { int ind1 = mol.indMolecule_1; int ind2 = mol.indMolecule_2; if (mol.f > Mparent [ind1].f && mol.f > Mparent [ind2].f) { if (Mparent [ind1].f < Mparent [ind2].f) { ArrayCopy (Mparent [ind1].structure, mol.structure); Mparent [ind1].f = mol.f; Mparent [ind1].NumHit = 0; } else { ArrayCopy (Mparent [ind2].structure, mol.structure); Mparent [ind2].f = mol.f; Mparent [ind2].NumHit = 0; } } else { Mparent [ind1].NumHit++; Mparent [ind2].NumHit++; } } //——————————————————————————————————————————————————————————————————————————————
И в завершение опишем метод "N" класса "C_AO_CRO", который используется для изменения структуры молекул в операторах химических реакций. Метод используется для генерации нового значения координаты молекулы в рамках заданного диапазона:
1. Метод принимает координату молекулы "coord", которую нужно модифицировать как ссылку, и позицию этой координаты "coordPos" в структуре.
2. Затем вычисляется расстояние "dist", которое представляет собой разницу между максимальным и минимальным значениями диапазона, умноженную на параметр "molecPerturb", означающий разброс значений в окрестностях текущего значения координаты.
3. Далее определяются минимальное "min" и максимальное "max" значения новой координаты. Эти значения равны старому значению координаты плюс-минус расстояние "dist", но они не могут выходить за пределы заданного диапазона "rangeMin[coordPos]" до "rangeMax[coordPos]".
4. Наконец, новое значение координаты генерируется с помощью функции Гауссова распределения "u.GaussDistribution", которая принимает старое значение координаты, минимальное и максимальное значения стандартного отклонения, равное 8.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_CRO::N (double &coord, int coordPos) { double dist = (rangeMax [coordPos] - rangeMin [coordPos]) * molecPerturb; double min = coord - dist; if (min < rangeMin [coordPos]) min = rangeMin [coordPos]; double max = coord + dist; if (max > rangeMax [coordPos]) max = rangeMax [coordPos]; coord = u.GaussDistribution (coord, min, max, 8); } //——————————————————————————————————————————————————————————————————————————————
Итак, мы разобрали все химические операторы, изучив их функциональность и роль в процессе оптимизации. Этот анализ предоставил нам глубокое понимание механизмов, которые управляют динамикой молекул в моделировании химических реакций (CRO).
В следующей статье мы займемся сборкой алгоритма и тестированием на тестовых функциях. Мы настроим и объединим изученные операторы в полноценный алгоритм CRO, чтобы он был готов к применению на практических задачах. Затем мы проведем серию экспериментов на широком спектре тестовых функций, которые позволят нам оценить эффективность и надежность нашего подхода.
Получив результаты работы, мы сделаем выводы о сильных и слабых сторонах алгоритма, а также о возможных направлениях для дальнейших улучшений. Это позволит нам не только усовершенствовать наш метод, но и предоставить полезные рекомендации для исследователей, работающих в области оптимизации и моделирования химических процессов.
Таким образом, следующая статья будет ключевым этапом в нашем исследовании, где мы перейдем от теории к практике, тестируя и улучшая наш алгоритм для достижения наилучших результатов в решении сложных задач оптимизации.
 Разработка системы репликации (Часть 39): Прокладываем путь (III)
Разработка системы репликации (Часть 39): Прокладываем путь (III)
 Возможности Мастера MQL5, которые вам нужно знать (Часть 12): Полином Ньютона
Возможности Мастера MQL5, которые вам нужно знать (Часть 12): Полином Ньютона
 Алгорим оптимизации химическими реакциями — Chemical reaction optimisation, CRO (Часть II): Сборка и результаты
Алгорим оптимизации химическими реакциями — Chemical reaction optimisation, CRO (Часть II): Сборка и результаты
- Бесплатные приложения для трейдинга
- 8 000+ сигналов для копирования
- Экономические новости для анализа финансовых рынков
Вы принимаете политику сайта и условия использования

Вот статья о практическом применении в трейдинге. Читайте, изучайте и применяйте на практике. Желаю вам успехов в трейдинге.
Articles
Using optimization algorithms to configure EA parameters on the fly
Andrey Dik, 2024.06.07 14:30
The article discusses the practical aspects of using optimization algorithms to find the best EA parameters on the fly, as well as virtualization of trading operations and EA logic. The article can be used as an instruction for implementing optimization algorithms into an EA.Здесь статья о практическом применении в трейдинге. Читайте, изучайте и применяйте в практике. Желаю вам успеха в трейдинге.
Мне нравится ваша точка зрения, Андрей! Великие инновации рождаются из исследований. Все не всегда сводится к деньгам, и я ценю ваш вклад в развитие сообщества.