
Algoritmo de otimização baseado em brainstorming — Brain Storm Optimization (Parte II): Multimodalidade
Conteúdo:
1. Introdução
Na primeira parte do artigo, mergulhamos no mundo da otimização com o algoritmo BSO (Brain Storm Optimization), onde revelamos os princípios básicos desta metodologia inovadora, inspirada na tempestade de ideias. Nós não apenas exploramos sua estrutura lógica, mas também nos aprofundamos na discussão dos métodos de clusterização, K-Means e K-Means++. O algoritmo Brain Storm Optimization (BSO) é um método de otimização que inclui fases de geração e avaliação de ideias em atividades em grupo. Esse algoritmo contribuiu para a área de otimização em conjunto com métodos de clusterização. A clusterização permite identificar grupos de dados de elementos semelhantes, o que ajuda o BSO a encontrar soluções ótimas. A aplicação do método de mutação permite que o algoritmo contorne obstáculos no espaço de busca de soluções e encontre caminhos mais eficientes para o ótimo.
Agora é hora de partir para a ação! Na segunda parte de nossa pesquisa, mergulharemos na implementação prática do algoritmo, discutiremos a multimodalidade, testaremos o algoritmo e ansiosamente faremos um balanço final.
2. Implementação do algoritmo
Vamos resumir os pontos-chave na lógica do algoritmo BSO:
- Clusterização.
- Deslocamento de clusters.
- Seleção de ideias dos clusters: centroides dos clusters ou ideias dos clusters.
- Fusão das ideias selecionadas.
- Mutação das ideias obtidas nas etapas anteriores.
- Seleção de ideias para as etapas 2, 3, 4. Inserção de novas ideias na população pai e ordenação.
Vamos para a descrição do código do algoritmo BSO.
Escreveremos a estrutura do agente do algoritmo "S_BSO_Agent", usada para descrever cada agente no algoritmo BSO.
1. A estrutura contém os seguintes campos:
- c[] - array para armazenar as coordenadas do agente.
- f - variável para armazenar a avaliação (fitness) do agente.
- label - variável para armazenar o rótulo de pertencimento ao cluster.
2. Init - método da estrutura "S_BSO_Agent", que inicializa os campos da estrutura. Ele aceita um argumento inteiro "coords", usado para redimensionar o array de coordenadas "c" usando a função "ArrayResize".
3. f = -DBL_MAX - define o valor inicial da variável "f" como o menor valor possível para um número do tipo double.
4. label = -1 - define o valor inicial da variável "label" como -1, o que significa que o agente não pertence a nenhum cluster.
Esse código representa a estrutura básica de dados para agentes no algoritmo de otimização BSO e inicializa seus campos ao criar um novo agente.
//—————————————————————————————————————————————————————————————————————————————— struct S_BSO_Agent { double c []; //coordinates double f; //fitness int label; //cluster membership label void Init (int coords) { ArrayResize (c, coords); f = -DBL_MAX; label = -1; } }; //——————————————————————————————————————————————————————————————————————————————
O algoritmo de clusterização K-Means e K-Means++ já foi detalhado no artigo anterior e não será abordado novamente aqui.
Vamos agora para a descrição do código da classe "C_AO_BSO", que é derivada da classe base de algoritmos populacionais "C_AO" e contém os seguintes campos e métodos:
1. Campos públicos:
- ao_name - nome do algoritmo de otimização.
- ao_desc - descrição do algoritmo de otimização.
- ao_link - link para o artigo sobre o algoritmo de otimização.
- popSize - tamanho da população.
- parentPopSize - tamanho da população de pais.
- clustersNumb - número de clusters.
- p_Replace - probabilidade de substituição.
- p_One - probabilidade de seleção única.
- p_One_center - probabilidade de selecionar um centro ou indivíduo no cluster escolhido.
- p_Two_center - probabilidade de selecionar dois centros ou dois indivíduos no cluster escolhido.
- k_Mutation - coeficiente de mutação.
- distribCoeff - coeficiente de distribuição.
- agent - array de agentes.
- parents - array de pais.
- clusters - array de clusters.
- km - objeto da classe KMeans.
2. Métodos:
- SetParams - método para definir os parâmetros do algoritmo.
- Init - método para inicializar o algoritmo. Aceita intervalos mínimos e máximos de busca, passo de busca e número de épocas.
- Moving - método para mover os agentes.
- Revision - método para revisão dos agentes.
3. Campos privados:
- parentsTemp - array temporário de pais.
- epochs - número máximo de épocas.
- epochsNow - época atual.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BSO : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_BSO () { } C_AO_BSO () { ao_name = "BSO"; ao_desc = "Brain Storm Optimization"; ao_link = "https://www.mql5.com/en/articles/14622"; popSize = 25; //population size parentPopSize = 50; //parent population size; clustersNumb = 5; //number of clusters p_Replace = 0.1; //replace probability p_One = 0.5; //probability of choosing one p_One_center = 0.3; //probability of choosing one center p_Two_center = 0.2; //probability of choosing two centers k_Mutation = 20.0; //mutation coefficient distribCoeff = 1.0; //distribution coefficient ArrayResize (params, 9); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "parentPopSize"; params [1].val = parentPopSize; params [2].name = "clustersNumb"; params [2].val = clustersNumb; params [3].name = "p_Replace"; params [3].val = p_Replace; params [4].name = "p_One"; params [4].val = p_One; params [5].name = "p_One_center"; params [5].val = p_One_center; params [6].name = "p_Two_center"; params [6].val = p_Two_center; params [7].name = "k_Mutation"; params [7].val = k_Mutation; params [8].name = "distribCoeff"; params [8].val = distribCoeff; } void SetParams () { popSize = (int)params [0].val; parentPopSize = (int)params [1].val; clustersNumb = (int)params [2].val; p_Replace = params [3].val; p_One = params [4].val; p_One_center = params [5].val; p_Two_center = params [6].val; k_Mutation = params [7].val; distribCoeff = params [8].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); void Injection (const int popPos, const int coordPos, const double value); //---------------------------------------------------------------------------- int parentPopSize; //parent population size; int clustersNumb; //number of clusters double p_Replace; //replace probability double p_One; //probability of choosing one double p_One_center; //probability of choosing one center double p_Two_center; //probability of choosing two centers double k_Mutation; //mutation coefficient double distribCoeff; //distribution coefficient S_BSO_Agent agent []; S_BSO_Agent parents []; S_Clusters clusters []; S_BSO_KMeans km; private: //------------------------------------------------------------------- S_BSO_Agent parentsTemp []; int epochs; int epochsNow; }; //——————————————————————————————————————————————————————————————————————————————
O método "Init" da classe "C_AO_BSO" realiza as seguintes ações para inicializar o algoritmo de otimização:
- Verificação de inicialização. Primeiro, o método "StandardInit" é chamado com os parâmetros de intervalo de busca e passo. Se "StandardInit" retornar "false", a inicialização é interrompida, e o método "Init" retorna "false".
- Inicialização de agentes. O tamanho do array "agent" é alterado para "popSize", e para cada agente, o método "Init" é chamado com o parâmetro "coords", que define o número de coordenadas.
- Inicialização de clusters. O tamanho do array "clusters" é alterado para "clustersNumb" (número máximo de clusters), e para cada cluster o método "Init" é chamado.
- Inicialização de pais. Os tamanhos dos arrays "parents" e "parentsTemp" são ajustados para "parentPopSize + popSize", e o método "Init" é chamado para cada pai. Os arrays precisam ser grandes o suficiente para conter tanto a população de pais quanto a de filhos para posterior ordenação.
- Definição de épocas. Os valores "epochs" e "epochsNow" são definidos segundo o parâmetro fornecido "epochsP" e "0", respectivamente.
O método retorna "true" se todas as etapas de inicialização forem bem-sucedidas. Isso prepara o algoritmo para realizar a otimização para o número especificado de épocas.
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_BSO::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (agent, popSize); for (int i = 0; i < popSize; i++) agent [i].Init (coords); ArrayResize (clusters, clustersNumb); for (int i = 0; i < clustersNumb; i++) clusters [i].Init (coords); ArrayResize (parents, parentPopSize + popSize); ArrayResize (parentsTemp, parentPopSize + popSize); for (int i = 0; i < parentPopSize + popSize; i++) { parents [i].Init (coords); parentsTemp [i].Init (coords); } epochs = epochsP; epochsNow = 0; return true; } //——————————————————————————————————————————————————————————————————————————————
O método "Moving" da classe "C_AO_BSO" é usado para mover os agentes durante o processo de otimização. O método realiza as seguintes ações:
- O valor da época atual é incrementado ("epochsNow++").
- Se "revision" for igual a "false", as coordenadas dos agentes são inicializadas usando valores aleatórios nos intervalos fornecidos. O método então conclui.
- Se "revision" não for "false", para cada agente são calculadas novas coordenadas utilizando fórmulas e probabilidades.
- Vários cálculos matemáticos, números aleatórios e probabilidades são usados para determinar as novas coordenadas dos agentes.
- Novas coordenadas são calculadas segundo as condições e probabilidades.
- As novas coordenadas são definidas usando o método "SeInDiSp" para corrigir os valores conforme os intervalos de busca e os passos.
- Uma nova ideia é gerada, que substitui o centro selecionado do cluster (deslocamento do centro do cluster) se a condição "u.RNDprobab() < p_Replace" for atendida.
- Uma ideia é escolhida de um cluster se a condição "u.RNDprobab() < p_One" for atendida.
- Ideias são selecionadas de dois clusters se a condição "u.RNDprobab() < p_One" não for atendida.
- As coordenadas dos agentes passam por mutação.
- As novas coordenadas dos agentes são salvas.
Esse método é responsável por atualizar as coordenadas dos agentes no algoritmo de otimização BSO conforme a época atual e as probabilidades. Vamos destacar em cores as seções do código que descrevem os diferentes comportamentos dos agentes:
- Geração de nova ideia. A cada nova época, os agentes exploram mais detalhadamente as proximidades da solução global encontrada conforme o coeficiente "p_Replace", buscando se aproximar do ótimo global e deslocando os centroides dos clusters.
- Exploração da vizinhança de um cluster. Considerando o coeficiente "p_One", os agentes exploram as proximidades de um cluster selecionado aleatoriamente. Assim, continua-se a explorar todas as áreas onde os agentes da população estão localizados.
- Seleção de ideias de dois clusters. Se a condição "u.RNDprobab() < p_One" não for atendida, ideias são escolhidas de dois clusters selecionados aleatoriamente.
- Mutação. As coordenadas dos agentes passam por mutação, o que garante diversidade na população e ajuda a evitar a convergência precoce para ótimos locais.
- Salvamento dos agentes. Após todas as operações, as novas coordenadas dos agentes são salvas para a próxima iteração.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BSO::Moving () { epochsNow++; //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); agent [i].c [c] = a [i].c [c]; } } return; } //---------------------------------------------------------------------------- //---------------------------------------------------------------------------- int cIndx_1 = 0; //index in the list of non-empty clusters int iIndx_1 = 0; //index in the list of ideas in the cluster int cIndx_2 = 0; //index in the list of non-empty clusters int iIndx_2 = 0; //index in the list of ideas in the cluster double min = 0.0; double max = 0.0; double dist = 0.0; double val = 0.0; double X1 = 0.0; double X2 = 0.0; int clListSize = 0; int clustList []; ArrayResize (clustList, 0, clustersNumb); //---------------------------------------------------------------------------- //let's make a list of non-empty clusters for (int cl = 0; cl < clustersNumb; cl++) { if (clusters [cl].count > 0) { clListSize++; ArrayResize (clustList, clListSize); clustList [clListSize - 1] = cl; } } for (int i = 0; i < popSize; i++) { //========================================================================== //generating a new idea that replaces the selected cluster center (cluster center offset) if (u.RNDprobab () < p_Replace) { cIndx_1 = u.RNDminusOne (clListSize); for (int c = 0; c < coords; c++) { val = clusters [clustList [cIndx_1]].centroid [c]; dist = (rangeMax [c] - rangeMin [c]) * 0.8; min = val - dist; if (min < rangeMin [c]) min = rangeMin [c]; max = val + dist; if (max > rangeMax [c]) max = rangeMax [c]; val = u.GaussDistribution (val, min, max, 3); val = u.SeInDiSp (val, rangeMin [c], rangeMax [c], rangeStep [c]); clusters [clustList [cIndx_1]].centroid [c] = val; } } //========================================================================== //an idea from one cluster is selected if (u.RNDprobab () < p_One) { cIndx_1 = u.RNDminusOne (clListSize); //------------------------------------------------------------------------ if (u.RNDprobab () < p_One_center) //select cluster center { for (int c = 0; c < coords; c++) { a [i].c [c] = clusters [clustList [cIndx_1]].centroid [c]; } } //------------------------------------------------------------------------ else //random idea from the cluster { iIndx_1 = u.RNDminusOne (clusters [clustList [cIndx_1]].count); for (int c = 0; c < coords; c++) { a [i].c [c] = parents [clusters [clustList [cIndx_1]].ideasList [iIndx_1]].c [c]; } } } //========================================================================== //select ideas from two clusters else { if (clListSize == 1) { cIndx_1 = 0; cIndx_2 = 0; } else { if (clListSize == 2) { cIndx_1 = 0; cIndx_2 = 1; } else { cIndx_1 = u.RNDminusOne (clListSize); do { cIndx_2 = u.RNDminusOne (clListSize); } while (cIndx_1 == cIndx_2); } } //------------------------------------------------------------------------ if (u.RNDprobab () < p_Two_center) //two cluster centers selected { for (int c = 0; c < coords; c++) { X1 = clusters [clustList [cIndx_1]].centroid [c]; X2 = clusters [clustList [cIndx_2]].centroid [c]; a [i].c [c] = u.RNDfromCI (X1, X2); } } //------------------------------------------------------------------------ else //two ideas from two selected clusters { iIndx_1 = u.RNDminusOne (clusters [clustList [cIndx_1]].count); iIndx_2 = u.RNDminusOne (clusters [clustList [cIndx_2]].count); for (int c = 0; c < coords; c++) { X1 = parents [clusters [clustList [cIndx_1]].ideasList [iIndx_1]].c [c]; X2 = parents [clusters [clustList [cIndx_2]].ideasList [iIndx_2]].c [c]; a [i].c [c] = u.RNDfromCI (X1, X2); } } } //========================================================================== //Mutation for (int c = 0; c < coords; c++) { int x = (int)u.Scale (epochsNow, 1, epochs, 1, 200); double ξ = (1.0 / (1.0 + exp (-((100 - x) / k_Mutation))));// * u.RNDprobab (); double dist = (rangeMax [c] - rangeMin [c]) * distribCoeff * ξ; double min = a [i].c [c] - dist; if (min < rangeMin [c]) min = rangeMin [c]; double max = a [i].c [c] + dist; if (max > rangeMax [c]) max = rangeMax [c]; val = a [i].c [c]; a [i].c [c] = u.GaussDistribution (val, min, max, 8); } //Save the agent----------------------------------------------------------- for (int c = 0; c < coords; c++) { val = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].c [c] = val; agent [i].c [c] = val; } } } //——————————————————————————————————————————————————————————————————————————————
A principal tarefa do método "Revision" da classe "C_AO_BSO" é atualizar a solução global e construir os clusters das soluções. O método realiza as seguintes ações:
- Obtenção da adaptabilidade. Para cada agente da população, o valor da adaptabilidade é extraído e salvo no campo correspondente da estrutura do agente.
- Transferência de novas ideias para a população. As novas ideias (agentes) geradas durante o processo de otimização são adicionadas à população de pais.
- Ordenação da população de pais. A população de pais é ordenada pelo valor da adaptabilidade. Isso permite que apenas as melhores soluções participem da criação de novas ideias na próxima época.
- Verificação da melhor solução. Se a adaptabilidade do melhor agente na população de pais exceder a solução atual, a melhor solução e suas coordenadas são atualizadas.
- Execução da clusterização. Se esta for a primeira iteração, o algoritmo k-means é inicializado com a população de pais e os clusters. Em seguida, o algoritmo k-means é executado para realizar a clusterização da população de pais.
- Atribuição da melhor solução do cluster como centro do cluster. Para cada cluster, verifica-se se há agentes nele (os clusters podem acabar vazios). Se houver, para cada agente na população de pais, verifica-se se ele pertence ao cluster. Se a adaptabilidade do agente exceder a adaptabilidade atual do cluster, a adaptabilidade do cluster e seu centroide são atualizados (o centroide participa da criação de novas ideias).
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BSO::Revision () { //get fitness-------------------------------------------------- for (int i = 0; i < popSize; i++) { agent [i].f = a [i].f; } //pass new ideas to the population-------------------------------------------- for (int i = parentPopSize; i < parentPopSize + popSize; i++) { parents [i] = agent [i - parentPopSize]; } //sort out the parent population---------------------------------------- u.Sorting (parents, parentsTemp, parentPopSize + popSize); if (parents [0].f > fB) { fB = parents [0].f; ArrayCopy (cB, parents [0].c, 0, 0, WHOLE_ARRAY); } //perform clustering----------------------------------------------------- if (!revision) { km.KMeansInit (parents, parentPopSize, clusters); revision = true; } km.KMeansInit (parents, parentPopSize, clusters); km.KMeans (parents, parentPopSize, clusters); //Assign the best cluster solution as the cluster center-------------------------- for (int cl = 0; cl < clustersNumb; cl++) { clusters [cl].f = -DBL_MAX; if (clusters [cl].count > 0) { for (int p = 0; p < parentPopSize; p++) { if (parents [p].label == cl) { if (parents [p].f > clusters [cl].f) { clusters [cl].f = parents [p].f; ArrayCopy (clusters [cl].centroid, parents [p].c, 0, 0, WHOLE_ARRAY); } } } } } }//——————————————————————————————————————————————————————————————————————————————
Falando sobre multimodalidade, o algoritmo BSO foi inicialmente apresentado como um método de otimização para resolver problemas multimodais. No entanto, os resultados dos testes mostraram que extremos locais significativos não são suficientemente explorados por este algoritmo, e muitos deles permanecem despercebidos. Talvez minha implementação atual não seja a mais otimizada. Por isso, decidi focar mais na adaptabilidade dos agentes no contexto da clusterização K-Means. Para isso, foram necessárias algumas alterações no algoritmo de clusterização.
Lembro que a multimodalidade nos algoritmos de otimização significa que a função a ser otimizada possui vários pontos ou picos ótimos. Tais funções podem conter diversos ótimos locais, que podem estar próximos do ótimo global em termos de valor da função de fitness ou ser significativos no contexto do problema a ser resolvido. A clusterização pode ajudar a identificar diferentes regiões no espaço de busca, onde a função apresenta várias modalidades.
Portanto, vamos tentar reforçar a influência da adaptabilidade dos agentes no processo de clusterização. Vamos envolver a função de cálculo das distâncias entre os agentes em uma nova função "FitnessDistance", que terá um parâmetro adicional "alpha", atuando como um coeficiente de equilíbrio entre a importância da distância e da adaptabilidade.
A função "FitnessDistance" calcula a distância entre o agente e o centroide do cluster, levando em consideração tanto a distância quanto a diferença na função de fitness entre eles. Isso é feito calculando a soma ponderada da distância e do valor absoluto da diferença entre a função de fitness do agente e do centroide. O peso "alpha" define a importância relativa da distância em comparação com a diferença na função de fitness.
//—————————————————————————————————————————————————————————————————————————————— double FitnessDistance (S_BSO_Agent &data, S_Cluster &clust, double alpha) { double distance = VectorDistance (data.c, clust.centroid); double fitness_diff = fabs (data.f - clust.f); return alpha * distance + (1 - alpha) * fitness_diff; } //——————————————————————————————————————————————————————————————————————————————
O método "KMeans" será complementado com o parâmetro "alpha":
void KMeans (S_BSO_Agent &data [], int dataSizeClust, S_Cluster &clust [], double alpha)
Alteraremos a parte do código do método "KMeans" responsável pela atualização dos centroides, de forma que cada cluster tenha o valor máximo da adaptabilidade do indivíduo pertencente ao cluster.
// Update the centroids double sum_c []; ArrayResize (sum_c, ArraySize (data [0].c)); double sum_f = 0.0; for (int cl = 0; cl < nClusters; cl++) { ArrayInitialize (sum_c, 0.0); clust [cl].count = 0; ArrayResize (clust [cl].ideasList, 0); sum_f = -DBL_MAX; for (int d = 0; d < dataSizeClust; d++) { if (data [d].label == cl) { for (int k = 0; k < ArraySize (data [d].c); k++) { sum_c [k] += data [d].c [k]; } if (data [d].f > sum_f) sum_f = data [d].f; clust [cl].count++; ArrayResize (clust [cl].ideasList, clust [cl].count); clust [cl].ideasList [clust [cl].count - 1] = d; } } if (clust [cl].count > 0) { for (int k = 0; k < ArraySize (sum_c); k++) { clust [cl].centroid [k] = sum_c [k] / clust [cl].count; } } }
As mudanças feitas permitem considerar a função de fitness na clusterização, mas não trouxeram uma melhora significativa na identificação de modos distintos na função de fitness, nem impactaram os resultados. Isso pode estar relacionado ao fato de que o uso da função de fitness no processo de clusterização nem sempre é eficaz, pelo menos na implementação atual do BSO.
Se os métodos K-Means e K-Means++ não trouxeram os resultados desejados, pode-se considerar outros métodos de clusterização:
1. A Clusterização Baseada em Densidade para Aplicações com Ruído (DBSCAN), um método de clusterização baseado em densidade, e não em distância. Ele agrupa pontos que estão próximos uns dos outros no espaço de características e possuem uma quantidade suficiente de vizinhos. O DBSCAN é um dos algoritmos de clusterização mais utilizados.
2. Clusterização Hierárquica (Hierarchical Clustering), um método que constrói uma hierarquia de clusters, onde cada cluster está ligado aos dois clusters mais próximos. A clusterização hierárquica pode ser aglomerativa (de baixo para cima) ou divisiva (de cima para baixo).
3. Mistura de Modelos Gaussianos (GMM), este modelo estatístico assume que todos os dados observados são gerados a partir de uma mistura de várias distribuições Gaussianas, cujos parâmetros são desconhecidos. Cada cluster corresponde a uma dessas distribuições.
4. Clusterização Espectral (Spectral Clustering), o método utiliza vetores próprios da matriz de similaridade para reduzir a dimensionalidade dos dados antes de realizar a clusterização em um espaço de baixa dimensão.
Existem muitos métodos de clusterização para experimentar e continuar as pesquisas nesta área. Para quem tiver interesse em explorar, o código anexado facilita a substituição do método K-Means por qualquer outro.
3. Resultados dos testes
Saída impressa dos resultados da execução do algoritmo BSO:
BSO|Brain Storm Optimization|25.0|50.0|5.0|0.1|0.5|0.3|0.2|20.0|1.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.9301770731803266
25 Hilly's; Func runs: 10000; result: 0.5801719580773876
500 Hilly's; Func runs: 10000; result: 0.30916005647304245
=============================
5 Forest's; Func runs: 10000; result: 0.929981802038364
25 Forest's; Func runs: 10000; result: 0.5907047167619348
500 Forest's; Func runs: 10000; result: 0.2477599978259004
=============================
5 Megacity's; Func runs: 10000; result: 0.5246153846153847
25 Megacity's; Func runs: 10000; result: 0.2784615384615384
500 Megacity's; Func runs: 10000; result: 0.1253384615384627
=============================
All score: 4.51637 (50.18%)
Os resultados dos testes do algoritmo em funções de teste (o resultado geral (All score) de 4,51637 corresponde a 50,18% do valor máximo possível) mostram que, ao utilizar os parâmetros indicados na primeira linha das saídas impressas, são obtidos resultados bastante satisfatórios. Os valores dos resultados das funções variam de 0,125 para 1000 parâmetros otimizáveis até 0,93 para 10, o que indica o sucesso do algoritmo na busca de soluções ótimas.
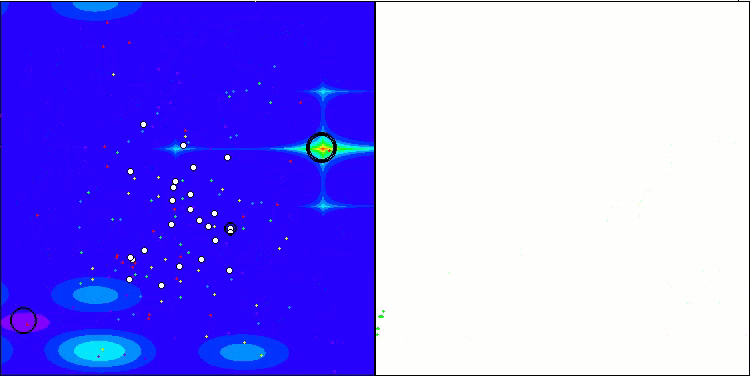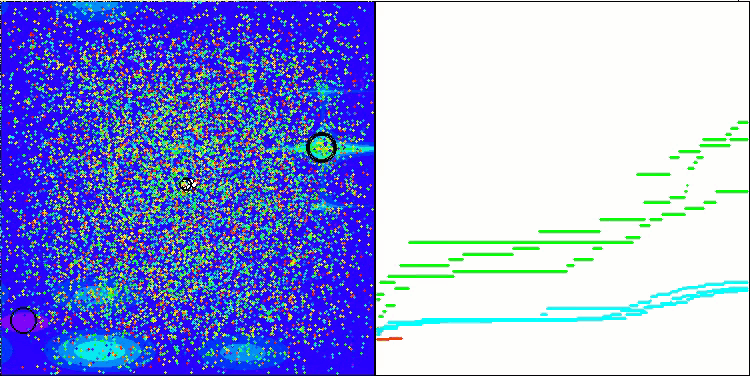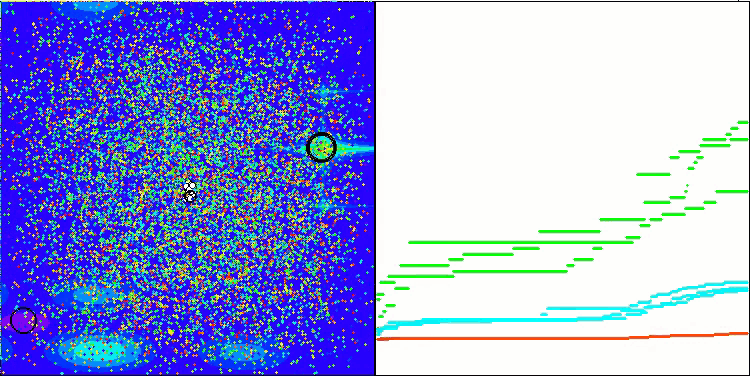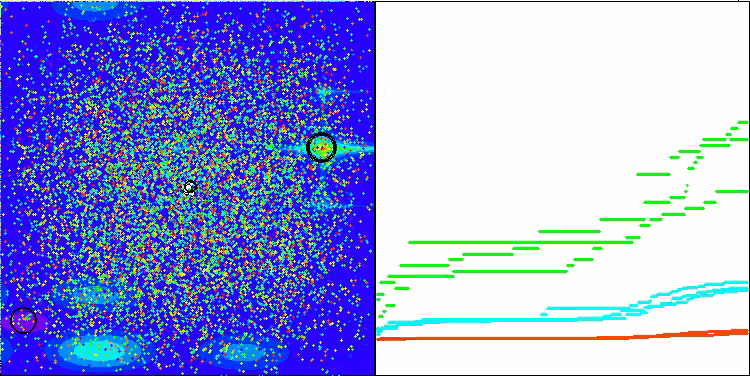
Gostaria de destacar como a visualização da clusterização das soluções aparece, especialmente nas funções com o maior número de parâmetros, onde o processo é claramente perceptível a cada iteração, à medida que as áreas características dos clusters começam a emergir do caos inicial.
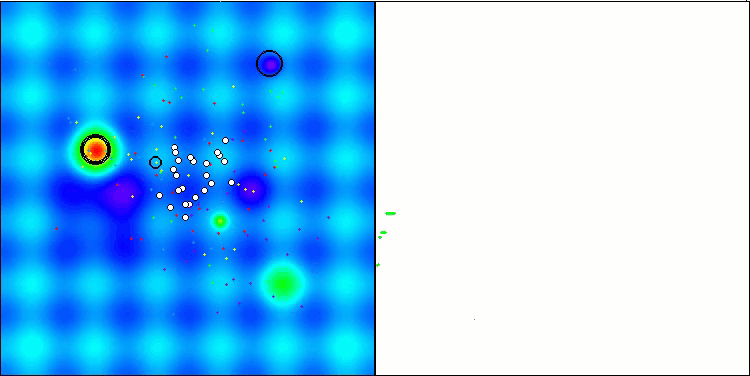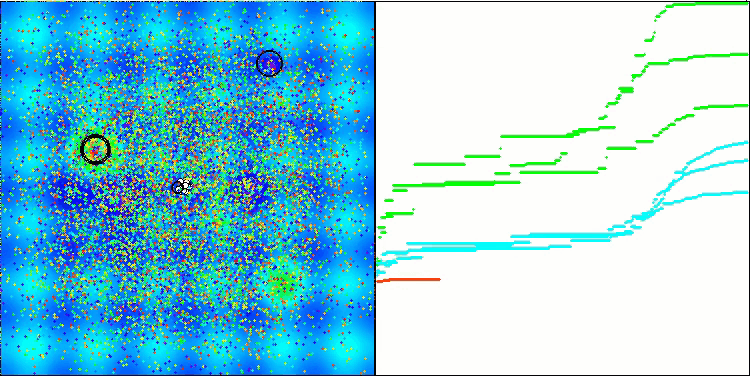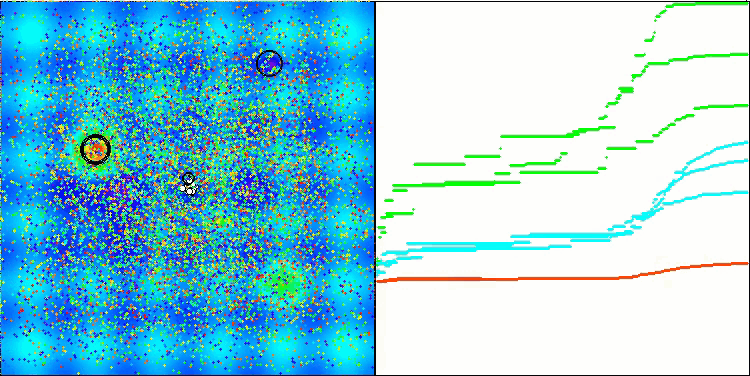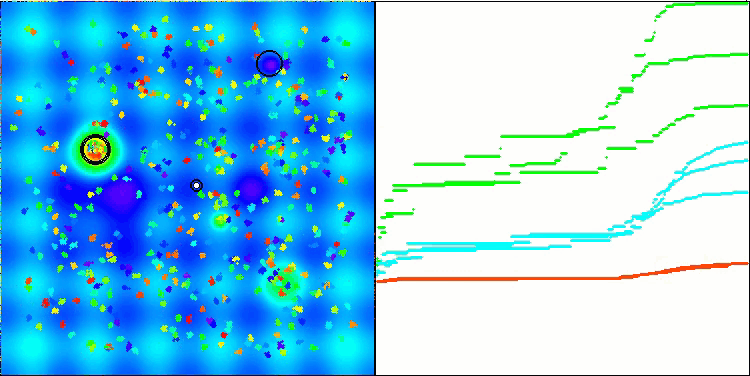
BSO na função de teste Hilly.
BSO na função de teste Forest.
BSO na função de teste Megacity.
Havia grandes expectativas para este algoritmo, e eu esperava vê-lo na primeira posição da tabela de classificação. Afinal, foi a primeira vez que apliquei o método de clusterização em conjunto com o método de mutação, que deveria apresentar resultados únicos para o algoritmo. Fiquei um pouco desapontado ao ver que o algoritmo ficou apenas na parte superior da tabela de classificação, mas não entre os líderes. Gostaria de salientar que o BSO apresentou excelentes resultados nas funções Forest e Megacity com 1000 parâmetros, dignos de aparecer entre os líderes da tabela.
№ | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | BGA | binary genetic algorithm | 0,99992 | 0,99484 | 0,50483 | 2,49959 | 1,00000 | 0,99975 | 0,32054 | 2,32029 | 0,90667 | 0,96400 | 0,23035 | 2,10102 | 6,921 | 76,90 |
| 2 | (P+O)ES | (P+O) evolution strategies | 0,99934 | 0,91895 | 0,56297 | 2,48127 | 1,00000 | 0,93522 | 0,39179 | 2,32701 | 0,83167 | 0,64433 | 0,21155 | 1,68755 | 6,496 | 72,18 |
| 3 | SDSm | stochastic diffusion search M | 0,93066 | 0,85445 | 0,39476 | 2,17988 | 0,99983 | 0,89244 | 0,19619 | 2,08846 | 0,72333 | 0,61100 | 0,10670 | 1,44103 | 5,709 | 63,44 |
| 4 | ESG | evolution of social groups | 0,99906 | 0,79654 | 0,35056 | 2,14616 | 1,00000 | 0,82863 | 0,13102 | 1,95965 | 0,82333 | 0,55300 | 0,04725 | 1,42358 | 5,529 | 61,44 |
| 5 | SIA | simulated isotropic annealing | 0,95784 | 0,84264 | 0,41465 | 2,21513 | 0,98239 | 0,79586 | 0,20507 | 1,98332 | 0,68667 | 0,49300 | 0,09053 | 1,27020 | 5,469 | 60,76 |
| 6 | DE | differential evolution | 0,95044 | 0,61674 | 0,30308 | 1,87026 | 0,95317 | 0,78896 | 0,16652 | 1,90865 | 0,78667 | 0,36033 | 0,02953 | 1,17653 | 4,955 | 55,06 |
| 7 | BSA | bird swarm algorithm | 0,90857 | 0,73661 | 0,25767 | 1,90285 | 0,90437 | 0,81619 | 0,16401 | 1,88457 | 0,61692 | 0,54154 | 0,10951 | 1,26797 | 5,055 | 56,17 |
| 8 | HS | harmony search | 0,86509 | 0,68782 | 0,32527 | 1,87818 | 0,99999 | 0,68002 | 0,09590 | 1,77592 | 0,62000 | 0,42267 | 0,05458 | 1,09725 | 4,751 | 52,79 |
| 9 | SSG | saplings sowing and growing | 0,77839 | 0,64925 | 0,39543 | 1,82308 | 0,85973 | 0,62467 | 0,17429 | 1,65869 | 0,64667 | 0,44133 | 0,10598 | 1,19398 | 4,676 | 51,95 |
| 10 | (PO)ES | (PO) evolution strategies | 0,79025 | 0,62647 | 0,42935 | 1,84606 | 0,87616 | 0,60943 | 0,19591 | 1,68151 | 0,59000 | 0,37933 | 0,11322 | 1,08255 | 4,610 | 51,22 |
| 11 | BSO | brain storm optimization | 0,91301 | 0,56222 | 0,30047 | 1,77570 | 0,97162 | 0,57162 | 0,23449 | 1,77772 | 0,60462 | 0,27138 | 0,12011 | 0,99611 | 4,550 | 50,55 |
| 12 | WOAm | wale optimization algorithm M | 0,84521 | 0,56298 | 0,26263 | 1,67081 | 0,93100 | 0,52278 | 0,16365 | 1,61743 | 0,66308 | 0,41138 | 0,11357 | 1,18803 | 4,476 | 49,74 |
| 13 | ACOm | ant colony optimization M | 0,88190 | 0,66127 | 0,30377 | 1,84693 | 0,85873 | 0,58680 | 0,15051 | 1,59604 | 0,59667 | 0,37333 | 0,02472 | 0,99472 | 4,438 | 49,31 |
| 14 | BFO-GA | bacterial foraging optimization - ga | 0,89150 | 0,55111 | 0,31529 | 1,75790 | 0,96982 | 0,39612 | 0,06305 | 1,42899 | 0,72667 | 0,27500 | 0,03525 | 1,03692 | 4,224 | 46,93 |
| 15 | MEC | mind evolutionary computation | 0,69533 | 0,53376 | 0,32661 | 1,55569 | 0,72464 | 0,33036 | 0,07198 | 1,12698 | 0,52500 | 0,22000 | 0,04198 | 0,78698 | 3,470 | 38,55 |
| 16 | IWO | invasive weed optimization | 0,72679 | 0,52256 | 0,33123 | 1,58058 | 0,70756 | 0,33955 | 0,07484 | 1,12196 | 0,42333 | 0,23067 | 0,04617 | 0,70017 | 3,403 | 37,81 |
| 17 | Micro-AIS | micro artificial immune system | 0,79547 | 0,51922 | 0,30861 | 1,62330 | 0,72956 | 0,36879 | 0,09398 | 1,19233 | 0,37667 | 0,15867 | 0,02802 | 0,56335 | 3,379 | 37,54 |
| 18 | COAm | cuckoo optimization algorithm M | 0,75820 | 0,48652 | 0,31369 | 1,55841 | 0,74054 | 0,28051 | 0,05599 | 1,07704 | 0,50500 | 0,17467 | 0,03380 | 0,71347 | 3,349 | 37,21 |
| 19 | SDOm | spiral dynamics optimization M | 0,74601 | 0,44623 | 0,29687 | 1,48912 | 0,70204 | 0,34678 | 0,10944 | 1,15826 | 0,42833 | 0,16767 | 0,03663 | 0,63263 | 3,280 | 36,44 |
| 20 | NMm | Nelder-Mead method M | 0,73807 | 0,50598 | 0,31342 | 1,55747 | 0,63674 | 0,28302 | 0,08221 | 1,00197 | 0,44667 | 0,18667 | 0,04028 | 0,67362 | 3,233 | 35,92 |
| 21 | FAm | firefly algorithm M | 0,58634 | 0,47228 | 0,32276 | 1,38138 | 0,68467 | 0,37439 | 0,10908 | 1,16814 | 0,28667 | 0,16467 | 0,04722 | 0,49855 | 3,048 | 33,87 |
| 22 | GSA | gravitational search algorithm | 0,64757 | 0,49197 | 0,30062 | 1,44016 | 0,53962 | 0,36353 | 0,09945 | 1,00260 | 0,32667 | 0,12200 | 0,01917 | 0,46783 | 2,911 | 32,34 |
| 23 | BFO | bacterial foraging optimization | 0,61171 | 0,43270 | 0,31318 | 1,35759 | 0,54410 | 0,21511 | 0,05676 | 0,81597 | 0,42167 | 0,13800 | 0,03195 | 0,59162 | 2,765 | 30,72 |
| 24 | ABC | artificial bee colony | 0,63377 | 0,42402 | 0,30892 | 1,36671 | 0,55103 | 0,21874 | 0,05623 | 0,82600 | 0,34000 | 0,14200 | 0,03102 | 0,51302 | 2,706 | 30,06 |
| 25 | BA | bat algorithm | 0,59761 | 0,45911 | 0,35242 | 1,40915 | 0,40321 | 0,19313 | 0,07175 | 0,66810 | 0,21000 | 0,10100 | 0,03517 | 0,34617 | 2,423 | 26,93 |
| 26 | SA | simulated annealing | 0,55787 | 0,42177 | 0,31549 | 1,29513 | 0,34998 | 0,15259 | 0,05023 | 0,55280 | 0,31167 | 0,10033 | 0,02883 | 0,44083 | 2,289 | 25,43 |
| 27 | IWDm | intelligent water drops M | 0,54501 | 0,37897 | 0,30124 | 1,22522 | 0,46104 | 0,14704 | 0,04369 | 0,65177 | 0,25833 | 0,09700 | 0,02308 | 0,37842 | 2,255 | 25,06 |
| 28 | PSO | particle swarm optimisation | 0,59726 | 0,36923 | 0,29928 | 1,26577 | 0,37237 | 0,16324 | 0,07010 | 0,60572 | 0,25667 | 0,08000 | 0,02157 | 0,35823 | 2,230 | 24,77 |
| 29 | Boids | boids algorithm | 0,43340 | 0,30581 | 0,25425 | 0,99346 | 0,35718 | 0,20160 | 0,15708 | 0,71586 | 0,27846 | 0,14277 | 0,09834 | 0,51957 | 2,229 | 24,77 |
| 30 | MA | monkey algorithm | 0,59107 | 0,42681 | 0,31816 | 1,33604 | 0,31138 | 0,14069 | 0,06612 | 0,51819 | 0,22833 | 0,08567 | 0,02790 | 0,34190 | 2,196 | 24,40 |
| 31 | SFL | shuffled frog-leaping | 0,53925 | 0,35816 | 0,29809 | 1,19551 | 0,37141 | 0,11427 | 0,04051 | 0,52618 | 0,27167 | 0,08667 | 0,02402 | 0,38235 | 2,104 | 23,38 |
| 32 | FSS | fish school search | 0,55669 | 0,39992 | 0,31172 | 1,26833 | 0,31009 | 0,11889 | 0,04569 | 0,47467 | 0,21167 | 0,07633 | 0,02488 | 0,31288 | 2,056 | 22,84 |
| 33 | RND | random | 0,52033 | 0,36068 | 0,30133 | 1,18234 | 0,31335 | 0,11787 | 0,04354 | 0,47476 | 0,25333 | 0,07933 | 0,02382 | 0,35648 | 2,014 | 22,37 |
| 34 | GWO | grey wolf optimizer | 0,59169 | 0,36561 | 0,29595 | 1,25326 | 0,24499 | 0,09047 | 0,03612 | 0,37158 | 0,27667 | 0,08567 | 0,02170 | 0,38403 | 2,009 | 22,32 |
| 35 | CSS | charged system search | 0,44252 | 0,35454 | 0,35201 | 1,14907 | 0,24140 | 0,11345 | 0,06814 | 0,42299 | 0,18333 | 0,06300 | 0,02322 | 0,26955 | 1,842 | 20,46 |
| 36 | EM | electroMagnetism-like algorithm | 0,46250 | 0,34594 | 0,32285 | 1,13129 | 0,21245 | 0,09783 | 0,10057 | 0,41085 | 0,15667 | 0,06033 | 0,02712 | 0,24412 | 1,786 | 19,85 |
Considerações finais
O algoritmo BSO tem várias vantagens, incluindo flexibilidade, equilíbrio entre exploração e exploração de soluções, além de adaptabilidade a diferentes tarefas de otimização.
No entanto, deve-se observar que a eficácia do algoritmo depende fortemente das configurações dos parâmetros externos (o grande número de parâmetros externos é a principal desvantagem do BSO), portanto, é necessário realizar pesquisas e experimentos rigorosos para determinar as melhores configurações para cada tarefa específica.
Convido todos os entusiastas da otimização a se juntarem aos experimentos e explorarem as possibilidades do algoritmo na solução de problemas práticos. Se alguém encontrar resultados mais interessantes e melhores parâmetros externos, por favor, compartilhe nos comentários do artigo.
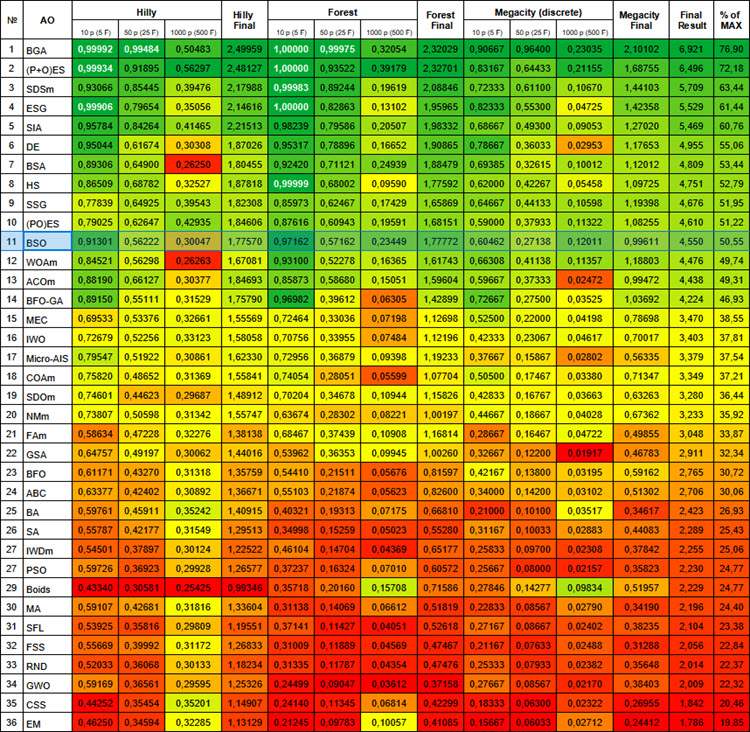
Figura 1. Gradiente de cores dos algoritmos conforme os respectivos testes. Os resultados maiores ou iguais a 0,99 são destacados em branco.
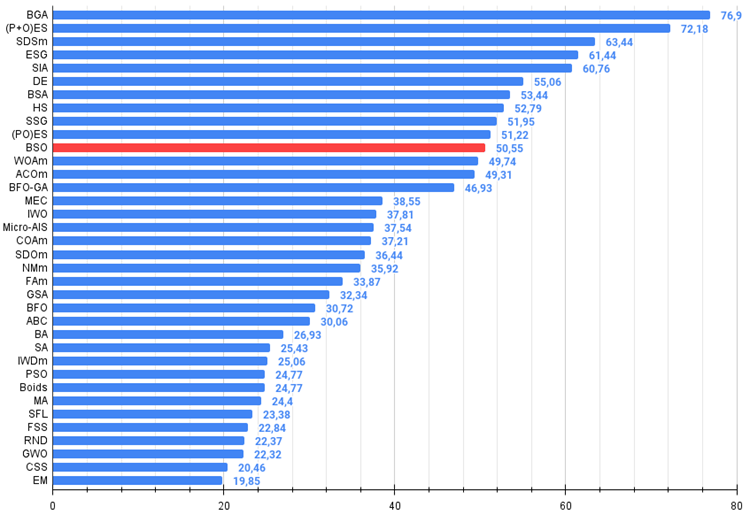
Figura 2. Histograma dos resultados dos testes dos algoritmos (na escala de 0 a 100, quanto maior, melhor,
onde 100 é o resultado teórico máximo possível, no arquivo há um script para calcular a tabela de classificação).
Vantagens e desvantagens do algoritmo de otimização baseado em brainstorming (BSO):
Vantagens:
- Bons resultados na função aguda Forest e na função discreta Megacity com alta dimensionalidade.
Desvantagens:
- Número muito grande de parâmetros externos.
- Arquitetura e implementação complexas.
- Alta exigência de recursos computacionais.
Um arquivo com as versões atualizadas dos códigos dos algoritmos está anexado ao artigo. O autor do artigo não se responsabiliza pela precisão absoluta na descrição dos algoritmos canônicos, já que muitas modificações foram feitas para melhorar as capacidades de busca. As conclusões e julgamentos apresentados nos artigos baseiam-se nos resultados dos experimentos realizados.
Traduzido do russo pela MetaQuotes Ltd.
Artigo original: https://www.mql5.com/ru/articles/14622
 Desenvolvendo um EA multimoeda (Parte 10): Criação de objetos a partir de uma string
Desenvolvendo um EA multimoeda (Parte 10): Criação de objetos a partir de uma string
 Técnicas do MQL5 Wizard que você deve conhecer (Parte 19): Inferência Bayesiana
Técnicas do MQL5 Wizard que você deve conhecer (Parte 19): Inferência Bayesiana
 Arbitragem triangular com previsões
Arbitragem triangular com previsões
 Modificação do Grid-Hedge EA em MQL5 (Parte IV): Otimizando a Estratégia de Grid Simples (I)
Modificação do Grid-Hedge EA em MQL5 (Parte IV): Otimizando a Estratégia de Grid Simples (I)
- Aplicativos de negociação gratuitos
- 8 000+ sinais para cópia
- Notícias econômicas para análise dos mercados financeiros
Você concorda com a política do site e com os termos de uso