Algoritmos de otimização populacionais: algoritmo híbrido de otimização de forrageamento bacteriano com algoritmo genético (Bacterial Foraging Optimization - Genetic Algorithm, BFO-GA)
Conteúdo:
1. Introdução
2. Descrição do algoritmo
3. Resultados dos testes
1. Introdução
O algoritmo híbrido de otimização BFO-GA combina dois algoritmos de otimização distintos: o algoritmo de otimização de forrageamento bacteriano (BFO) e o algoritmo genético (GA). Este algoritmo híbrido foi criado com o objetivo de melhorar o desempenho da otimização e superar algumas limitações de cada um dos algoritmos individuais.
O BFO (Bacterial Foraging Optimization) é um algoritmo de otimização inspirado no comportamento das bactérias ao buscar alimento. Ele foi proposto em 2002 por Rahul K. Kujur. O BFO modela o movimento das bactérias usando três mecanismos principais: transições, difusão e atualização de posição. Cada bactéria no algoritmo representa uma solução para o problema de otimização, e o alimento corresponde à solução ótima. As bactérias se movem no espaço de busca para encontrar o melhor alimento.
O algoritmo genético (GA) é um algoritmo de otimização inspirado nos princípios da seleção natural e genética. Ele foi desenvolvido por John Holland na década de 1970. O GA trabalha com uma população de indivíduos que representam soluções para o problema de otimização. Os indivíduos são submetidos a operações de cruzamento (combinação de informações genéticas) e mutação (alterações aleatórias na informação genética) para criar novas gerações. Após várias gerações, o GA busca encontrar a solução ótima.
O algoritmo híbrido BFO-GA combina as vantagens de ambos os algoritmos. O BFO tem uma boa capacidade de busca local e rápida convergência, mas pode ter dificuldades na busca global. Por outro lado, o GA tem uma boa capacidade de busca global, mas pode ser lento na convergência e propenso a ficar preso em ótimos locais. O algoritmo híbrido BFO-GA tenta superar essas limitações, utilizando o BFO para a busca global e o GA para refinar os ótimos locais.
O algoritmo híbrido BFO-GA (Bacterial Foraging Optimization - Genetic Algorithm) foi proposto em 2007 no trabalho de D. N. Kim, A. Abraham e Choo. Este algoritmo combina princípios de otimização baseados no comportamento de enxameamento bacteriano com operadores genéticos de seleção, cruzamento e mutação.
O BFO-GA usa o algoritmo bacteriano como base, mas o expande com operadores genéticos de seleção, cruzamento e mutação. Este algoritmo utiliza o enxameamento bacteriano para a busca da solução ótima.
Como operador de seleção, o BFO-GA usa um algoritmo baseado no método da roleta. Esse método seleciona bactérias parentais com probabilidades proporcionais à sua adaptação. Assim, bactérias mais adaptadas têm maior probabilidade de serem escolhidas para o cruzamento.
O cruzamento no algoritmo BFO-GA é realizado com o operador de cruzamento aritmético. Ele combina as informações genéticas de duas bactérias parentais, criando uma nova prole com novas combinações de genes.
A mutação no BFO-GA é feita com o mutador não uniforme de Michalewicz. Este mutador altera a informação genética dentro das bactérias, introduzindo mudanças aleatórias. A não uniformidade da mutação significa que a probabilidade de mutação pode variar dependendo de diferentes fatores.
Os operadores modificadores mencionados são executados após a conclusão dos procedimentos de quimiotaxia e reprodução do algoritmo de forrageamento bacteriano, antes dos procedimentos de eliminação e dispersão deste algoritmo. Ou seja, depois que as bactérias se movem em direção à solução ótima e produzem descendentes, os operadores genéticos são aplicados para diversificar e melhorar as soluções.
2. Descrição do algoritmo
O algoritmo BFO-GA utiliza a modelagem do comportamento de enxameamento das bactérias para buscar a solução ótima do problema de otimização. Ele imita o movimento das bactérias no espaço de parâmetros, onde cada bactéria representa uma potencial solução para o problema. As bactérias se movem no espaço de parâmetros realizando dois tipos de movimentos: movimento na direção do gradiente alimentar e movimento aleatório.
O algoritmo BFO-GA segue os seguintes passos:
- Inicialização: Criação da população inicial de bactérias, cada uma representando uma potencial solução para o problema. Cada bactéria possui seus próprios parâmetros, que podem ser modificados durante o processo de otimização.
- Avaliação do gradiente alimentar: Cálculo do valor da função de adaptação para cada bactéria na população e comparação dos dois últimos valores, onde essa diferença de valores determina a qualidade da solução representada por cada bactéria.
- Movimento na direção do gradiente: Cada bactéria se move na direção do gradiente alimentar, permitindo que se aproxime de soluções mais ótimas com um vetor constante de mudança de posição.
- Movimento aleatório: Parte das bactérias também realiza movimento aleatório para evitar ficar presa em ótimos locais. Esse movimento é baseado na alteração aleatória dos parâmetros da bactéria no caso de um movimento anterior malsucedido.
- Operadores genéticos: Aplicação dos operadores genéticos - seleção, cruzamento e mutação - à população de bactérias. Isso permite criar novas combinações de parâmetros e melhorar a qualidade das soluções.
- Repetição dos passos 2-5: Repetição dos passos de movimento na direção do gradiente, movimento aleatório e aplicação dos operadores genéticos até que um critério de parada seja alcançado, como o número máximo de iterações ou um valor específico da função de adaptação.
Teoricamente, o algoritmo híbrido BFO-GA deve ter várias vantagens sobre o BFO, que vale a pena mencionar:
- Exploração e aproveitamento: O BFO-GA possui a capacidade de explorar o espaço de parâmetros, graças ao movimento aleatório das bactérias, e de aproveitar os recursos, graças ao movimento na direção do gradiente alimentar. Isso permite que o algoritmo encontre soluções ótimas, evitando ótimos locais e convergindo para o global.
- Adaptabilidade: O BFO-GA tem a capacidade de se adaptar a condições de otimização mutáveis. Durante a execução do algoritmo, os parâmetros das bactérias podem ser ajustados, permitindo adaptação a novas condições e encontrando soluções mais ótimas.
- Ausência de necessidade de ajuste de parâmetros: Como qualquer algoritmo de otimização, o BFO-GA possui um conjunto de parâmetros que podem ser ajustados para alcançar melhores resultados. Isso inclui parâmetros relacionados ao tamanho da população de bactérias, passos de movimento na direção do gradiente e movimento aleatório, probabilidades dos operadores genéticos e outros. A alteração desses parâmetros não tem um impacto significativo no resultado.

Figura 1. Herança de genes parentais pela bactéria filha.
Passando das bases teóricas para a implementação prática.
Primeiramente, abandonei a seleção pelo método da roleta (método usado no algoritmo da colônia artificial de abelhas). Experimentalmente, obtive melhores resultados no BFO-GA usando uma seleção aleatória simples entre a população de pais.
Em segundo lugar, decidi substituir o mutador não uniforme de Michalewicz por uma mutação pela lei de potência (em algumas fontes mencionada como uma variação do mutador de potência), que é essencialmente a geração de um número aleatório com uma distribuição de potência, mencionada no artigo sobre o Cabeçudinho Inteligente.
Em terceiro lugar, o operador de cruzamento aritmético foi substituído por uma simples herança aleatória de genes de diferentes indivíduos parentais, escolhidos aleatoriamente com uma distribuição uniforme.
Para descrever a bactéria, vamos definir a estrutura `S_Bacteria`, contendo os seguintes campos:
- c: array de coordenadas da bactéria, que representa as coordenadas atuais da bactéria no espaço de parâmetros. O tamanho do array `c` depende do número de variáveis no problema de otimização.
- cLast: array das coordenadas anteriores da bactéria, usado para armazenar a posição anterior da bactéria no espaço de parâmetros, a partir das quais o novo posicionamento é calculado.
- v: array de vetores da bactéria, que representa a direção atual de movimento da bactéria no espaço de parâmetros. O tamanho do array `v` depende do número de variáveis no problema de otimização.
- f: valor atual da saúde (adaptação) da bactéria, que determina a qualidade da posição atual da bactéria no espaço de parâmetros. Quanto maior o valor de `f`, melhor.
- fLast: valor anterior da saúde da bactéria, usado para rastrear a qualidade anterior da posição da bactéria e para a determinação do gradiente alimentar.
- lifeCNT: contador de vida da bactéria, que rastreia o número de iterações que a bactéria passou na posição atual, usado para limitar o número de passos de movimentação das bactérias em uma direção.
//—————————————————————————————————————————————————————————————————————————————— struct S_Bacteria { double c []; //coordinates double cLast []; //coordinates double v []; //vector double f; //current health double fLast; //previous health double lifeCNT; //life counter }; //——————————————————————————————————————————————————————————————————————————————
Vamos declarar a classe `C_AO_BFO_GA`, que implementa o algoritmo BFO-GA (Bacterial Foraging Optimization - Genetic Algorithm). Vamos analisar cada campo e método da classe:
Campos da classe:
- b: um array de bactérias, onde cada elemento é um objeto do tipo "S_Bacteria", que representa uma bactéria no algoritmo.
- rangeMax: um array de valores máximos do intervalo de busca para cada variável no problema de otimização.
- rangeMin: um array de valores mínimos do intervalo de busca para cada variável.
- rangeStep: um array de passos de busca para cada variável.
- cB: um array com as melhores coordenadas encontradas pelo algoritmo.
- fB: o valor da função de adaptação para as melhores coordenadas.
Métodos da classe:
- Init: Inicializa os parâmetros do algoritmo BFO-GA, como a quantidade de parâmetros a serem otimizados "paramsP", o tamanho da população "populationSizeP", o parâmetro "lambda", que determina o comprimento do deslocamento das bactérias, a probabilidade de reprodução "reproductionP", o contador de vida "lifeCounterP", que especifica que após um número específico de ciclos de vida, as bactérias realizam uma reviravolta, e a força da mutação "powerMutP".
- Swimming: realiza o movimento das bactérias no espaço dos parâmetros.
- Evaluation: executa a revisão da população e a atualização da solução global.
Métodos privados da classe:
- NewVector: gera um novo vetor para o parâmetro "paramInd" da bactéria.
- PowerDistribution: aplica uma distribuição de potência ao valor de entrada "In" com os parâmetros fornecidos "outMin", "outMax", "power".
- Sorting: ordena as bactérias na população de acordo com seus valores na função de adaptação em ordem decrescente.
- SeInDiSp: normaliza o valor de entrada "In" no intervalo InMin, InMax com o passo "Step".
- RNDfromCI: gera um número aleatório no intervalo especificado min, max.
- Scale: escala o valor de entrada "In" do intervalo InMIN, InMAX para o intervalo OutMIN, OutMAX.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BFO_GA { //---------------------------------------------------------------------------- public: S_Bacteria b []; //bacteria public: double rangeMax []; //maximum search range public: double rangeMin []; //manimum search range public: double rangeStep []; //step search public: double cB []; //best coordinates public: double fB; //FF of the best coordinates public: void Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP, //life counter const double powerMutP); //mutation power public: void Swimming (); public: void Evaluation (); //---------------------------------------------------------------------------- private: double NewVector (int paramInd); private: S_Bacteria bT []; //bacteria private: double v []; private: int ind []; private: double val []; private: int populationSize; //population size private: int parameters; //number of optimized parameters private: double lambda; //lambda private: double reproduction; //probability of reproduction private: int lifeCounter; //life counter private: double powerMut; //mutation power private: bool evaluation; private: double PowerDistribution (const double In, const double outMin, const double outMax, const double power); private: void Sorting (); private: double SeInDiSp (double In, double InMin, double InMax, double Step); private: double RNDfromCI (double min, double max); private: double Scale (double In, double InMIN, double InMAX, double OutMIN, double OutMAX, bool revers); }; //——————————————————————————————————————————————————————————————————————————————
O método "Init" é usado para inicializar a classe, que configura os parâmetros do algoritmo BFO-GA.
Parâmetros de entrada do método:
- paramsP: número de parâmetros a serem otimizados.
- populationSizeP: tamanho da população.
- lambdaP: parâmetro lambda, que determina a distância de deslocamento das bactérias em cada passo.
- reproductionP: probabilidade de reprodução.
- lifeCounterP: contador de vida.
- powerMutP: força da mutação.
Dentro do método, os campos da classe C_AO_BFO_GA são inicializados com valores iniciais e os tamanhos dos arrays são definidos.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO_GA::Init (const int paramsP, //number of opt. parameters const int populationSizeP, //population size const double lambdaP, //lambda const double reproductionP, //probability of reproduction const int lifeCounterP, //life counter const double powerMutP) //mutation power { fB = -DBL_MAX; evaluation = false; parameters = paramsP; populationSize = populationSizeP; lambda = lambdaP; reproduction = reproductionP; lifeCounter = lifeCounterP; powerMut = powerMutP; ArrayResize (rangeMax, parameters); ArrayResize (rangeMin, parameters); ArrayResize (rangeStep, parameters); ArrayResize (v, parameters); ArrayResize (ind, populationSize); ArrayResize (val, populationSize); ArrayResize (b, populationSize); ArrayResize (bT, populationSize); for (int i = 0; i < populationSize; i++) { ArrayResize (b [i].c, parameters); ArrayResize (b [i].cLast, parameters); ArrayResize (b [i].v, parameters); b [i].f = -DBL_MAX; b [i].fLast = -DBL_MAX; ArrayResize (bT [i].c, parameters); ArrayResize (bT [i].cLast, parameters); ArrayResize (bT [i].v, parameters); } ArrayResize (cB, parameters); } //——————————————————————————————————————————————————————————————————————————————
Para realizar a quimiotaxia das bactérias, sua replicação, seleção, herança de características e mutação, vamos escrever o método "Swimming":
void Swimming ()
{
} Na primeira iteração, a flag "evaluation" é "false", distribuímos as bactérias aleatoriamente por todo o espaço de busca com uma distribuição uniforme. Nesta etapa, a saúde atual e anterior não são conhecidas, então atribuiremos às variáveis correspondentes o valor "-DBL_MAX", além de adicionar um vetor de deslocamento aleatório às bactérias recém-criadas.
As coordenadas obtidas das bactérias são verificadas para garantir que não saiam dos limites e aplicamos o passo de mudança dos parâmetros a serem otimizados. O vetor de movimento de cada bactéria permite que elas nadem de maneira uniforme e progressiva.
if (!evaluation) { fB = -DBL_MAX; for (int s = 0; s < populationSize; s++) { for (int k = 0; k < parameters; k++) { b [s].c [k] = RNDfromCI (rangeMin [k], rangeMax [k]); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); v [k] = rangeMax [k] - rangeMin [k]; b [s].v [k] = NewVector (k); b [s].f = -DBL_MAX; b [s].fLast = -DBL_MAX; b [s].lifeCNT = 0; } } evaluation = true; return; }
Nas iterações subsequentes, primeiro verificamos a probabilidade de reprodução (replicação) das bactérias. Se a probabilidade de reprodução for atendida, ocorre o seguinte:
- "bacterium original": a melhor metade da população ordenada continua seu movimento na mesma direção; para isso, adicionamos às coordenadas da posição anterior o vetor de movimento individual de cada bactéria.
- "bacterium clone": a segunda metade da população é preenchida por bactérias filhas, cada uma resultante dos "genes" (coordenadas) de diferentes bactérias parentais, promovendo assim a herança de características e a capacidade combinatória do algoritmo, conforme mostrado na Figura 1 acima.
- As bactérias clonadas, que herdaram genes de diferentes pais, também herdam o componente do vetor de movimento do progenitor, e o gene herdado sofre mutação de acordo com a lei de distribuição de potência.
Assim, as bactérias clonadas herdam características de vários pais. Os genes são modificados com alta probabilidade nas suas proximidades e, com uma probabilidade não nula, em valores distantes do gene parental. Isso permite combinar as melhores características dos pais, uma vez que os genes só podem ser transmitidos para os descendentes pelos melhores membros da população. Além disso, os componentes herdados do vetor de movimento fazem com que as bactérias filhas comecem a se mover na próxima iteração conforme os melhores componentes dos vetores de seus pais.
Idealmente, isso deve se assemelhar ao movimento de um cardume de peixes, mas essa analogia nem sempre se confirma devido a muitos fatores aleatórios que afetam o movimento das bactérias. Entre esses fatores estão o cenário da função de adaptação, a substituição por membros mais bem-sucedidos da população e a inevitável alteração do vetor de movimento devido à limitação do número de ciclos de vida.
O contador de vidas das bactérias na melhor metade da colônia é incrementado em um, enquanto o das clonadas é resetado.
double r = RNDfromCI (0.0, 1.0); if (r < reproduction) { int st = populationSize / 2; //bacterium original-------------------------------------------------------- for (int s = 0; s < st; s++) { for (int k = 0; k < parameters; k++) { b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++; } //bacterium clone----------------------------------------------------------- for (int s = st; s < populationSize; s++) { for (int k = 0; k < parameters; k++) { int i = (int)RNDfromCI (0, st); if (i >= st) i = st - 1; b [s].c [k] = b [i].cLast [k]; b [s].c [k] = PowerDistribution (b [s].c [k], rangeMin [k], rangeMax [k], powerMut); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT = 0; } }
Então, se a probabilidade de reprodução não for atendida, no loop para toda a população:
for (int s = 0; s < populationSize; s++)
Primeiro, verificamos se a bactéria atingiu o limite de vidas. As bactérias que atingiram o limite fazem uma reviravolta e começam a se mover em uma nova direção, alterando seu vetor de movimento. O contador de vidas é resetado.
Após verificar o limite de vidas, aquelas bactérias que o atingiram sofrem mutação e na próxima iteração começam a se mover em uma nova direção, alterando seu vetor de movimento e resetando o contador de vidas.
if (b [s].lifeCNT >= lifeCounter) { for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = PowerDistribution (b [s].cLast [k], rangeMin [k], rangeMax [k], powerMut); b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT = 0; }
Se a bactéria ainda não atingiu o limite de vidas, verificamos a presença de quimiotaxia positiva, ou seja, se a bactéria está se movendo na direção de aumento do gradiente de alimento. O movimento da bactéria é considerado correto se os valores de adaptação nas duas iterações anteriores forem iguais. Por que iguais? Porque na próxima etapa, no método "Evaluation", verificamos a mudança positiva na saúde das bactérias. Se as mudanças forem positivas, o estado atual é atribuído ao estado anterior. Portanto, se os estados de saúde nas duas últimas iterações forem iguais, isso indica a direção correta do movimento da bactéria na busca por mais alimento. Dessa forma, a bactéria só pode se mover em direção a ainda mais alimento. Caso contrário, a bactéria começa a se agitar no local. No entanto, isso não é um problema, pois, mais cedo ou mais tarde, a bactéria encalhada sofrerá uma mutação para um novo estado ou herdará os genes de pais mais afortunados e seu vetor de movimento.
O contador de vidas bacterianas que se movem na direção certa aumenta gradualmente. Isso significa que, eventualmente, até mesmo as bactérias bem-sucedidas sofrerão mutações, conforme mencionado no bloco de código anterior.
if (b [s].f == b [s].fLast) { for (int k = 0; k < parameters; k++) { b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++; }
E, se a quimiotaxia positiva não for observada em uma bactéria, essa bactéria mudará seu vetor de movimento dando uma cambalhota. O contador de vida dessas bactérias também continua correndo, então surgiu a ideia de aumentar mais intensamente o valor do contador de vida dessas bactérias malsucedidas, embora eu não tenha testado essa hipótese.
Se não houver quimiotaxia positiva na bactéria, ela muda seu vetor de movimento, fazendo uma cambalhota. O contador de vidas dessas bactérias continua funcionando. Surgiu uma ideia de aumentar o valor do contador de vidas dessas bactérias menos bem-sucedidas de forma mais intensa, embora eu não tenha verificado essa hipótese.
for (int k = 0; k < parameters; k++) { b [s].v [k] = NewVector (k); b [s].c [k] = b [s].cLast [k] + b [s].v [k]; b [s].c [k] = SeInDiSp (b [s].c [k], rangeMin [k], rangeMax [k], rangeStep [k]); } b [s].lifeCNT++;
Para realizar a cambalhota das bactérias, ou seja, mudar o vetor de movimento, usa-se o método "NewVector", que é executado para o parâmetro otimizável com o índice "paramInd":
- gera-se um número aleatório "r" com distribuição uniforme no intervalo de -1.0 a 1.0 usando a função "RNDfromCI".
- calcula-se o novo valor do componente do vetor multiplicando o intervalo permitido de valores "v" do parâmetro otimizável pelo coeficiente "lambda" e pelo número aleatório "r".
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BFO_GA::NewVector (int paramInd) { double r = RNDfromCI (-1.0, 1.0); return lambda * v [paramInd] * r; } //——————————————————————————————————————————————————————————————————————————————Para executar o processo de competição entre as bactérias e atualizar a solução global, utiliza-se o método "Evaluation".
No início deste método, cada bactéria na população é verificada quanto à presença de quimiotaxia positiva: se o valor atual da função de adaptação "bs.f" for maior que o valor anterior "bs.fLast", ocorre a atualização da adaptação anterior e das coordenadas anteriores, das quais as bactérias se moverão no futuro.
Em seguida, a população é ordenada pelo método "Sorting" em ordem decrescente dos valores da função de adaptação, usando o valor "fLast" das bactérias.
Depois disso, ocorre a atualização da solução global.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BFO_GA::Evaluation () { for (int s = 0; s < populationSize; s++) { if (b [s].f > b [s].fLast) { b [s].fLast = b [s].f; ArrayCopy (b [s].cLast, b [s].c, 0, 0, WHOLE_ARRAY); } } Sorting (); if (b [0].fLast > fB) { fB = b [0].fLast; ArrayCopy (cB, b [0].cLast, 0, 0, WHOLE_ARRAY); } } //——————————————————————————————————————————————————————————————————————————————
3. Resultados dos testes
Impressão do desempenho do algoritmo híbrido BFO-GA no banco de testes:
C_AO_BFO_GA:50;0.01;0.8;50;10.0
=============================
5 Hilly's; Func runs: 10000; result: 0.8914957961234459
25 Hilly's; Func runs: 10000; result: 0.5511088047221568
500 Hilly's; Func runs: 10000; result: 0.31529201642392934
=============================
5 Forest's; Func runs: 10000; result: 0.9698155977381523
25 Forest's; Func runs: 10000; result: 0.39612322805995287
500 Forest's; Func runs: 10000; result: 0.06304955092899359
=============================
5 Megacity's; Func runs: 10000; result: 0.7266666666666667
25 Megacity's; Func runs: 10000; result: 0.275
500 Megacity's; Func runs: 10000; result: 0.035250000000000004
=============================
All score: 4.22380 (46.93%)
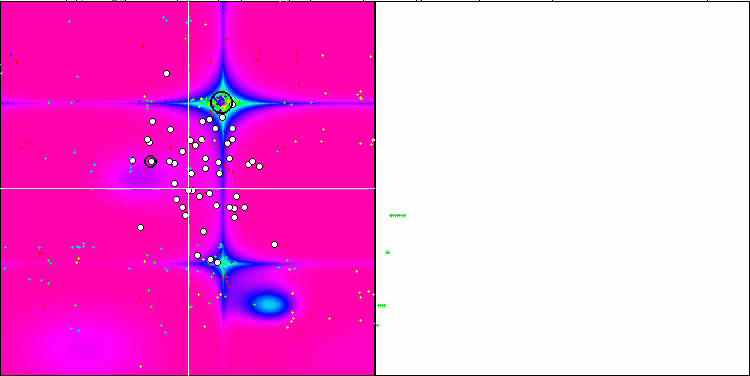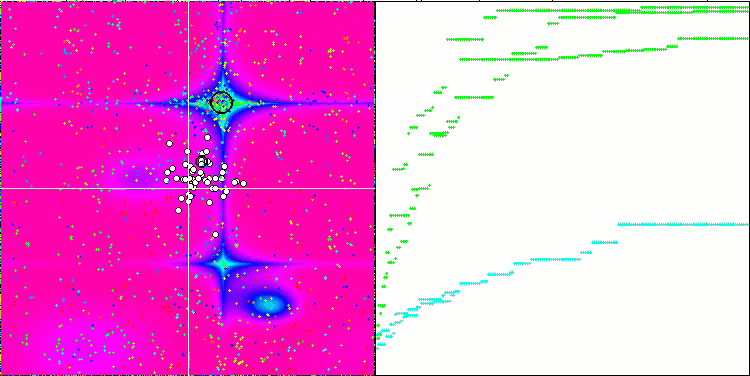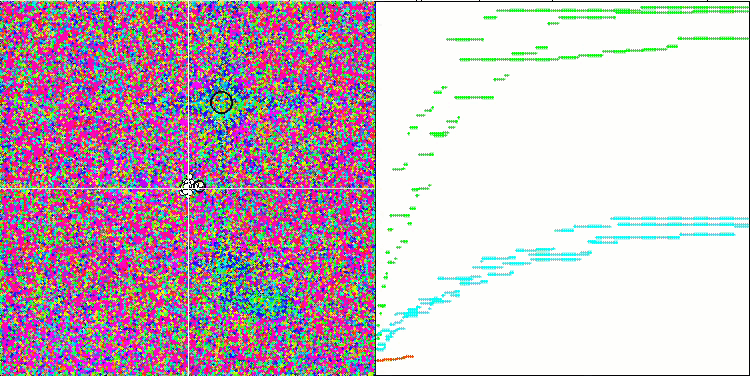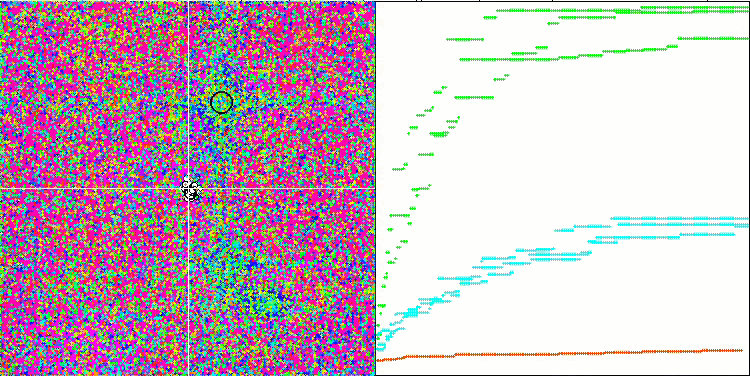
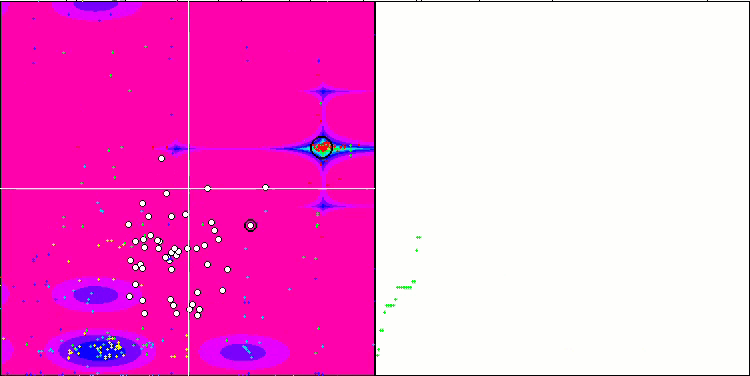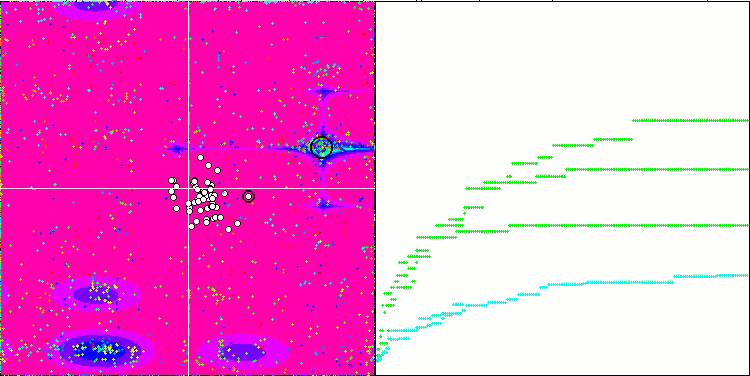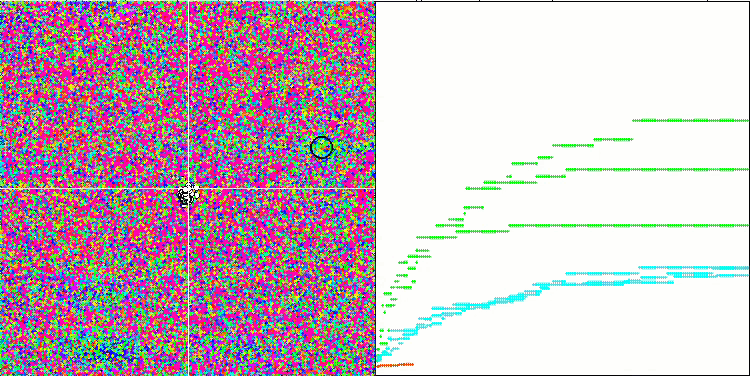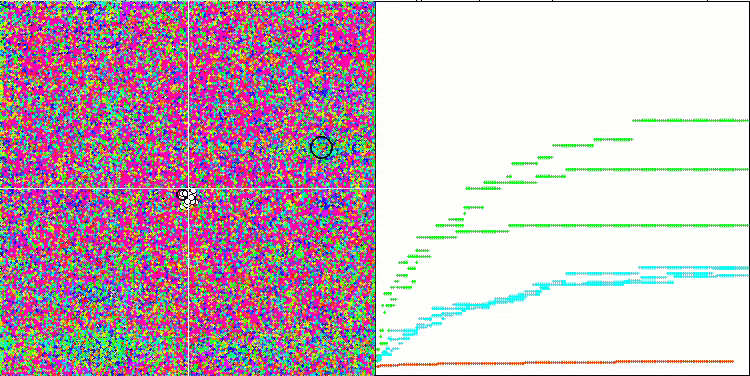
A análise visual do funcionamento do banco de testes mostra que o algoritmo BFO-GA rapidamente encontra a região do extremo global, dedicando menos atenção aos extremos locais, o que pode levar a um bloqueio se o extremo global for ilusório (errôneo). No geral, o algoritmo converge de forma bastante confiável, embora um pouco lentamente. Observa-se um certo "enxameamento", o que é um bom sinal, mas não há conexão total entre as bactérias, e elas se comportam como unidades independentes da população.
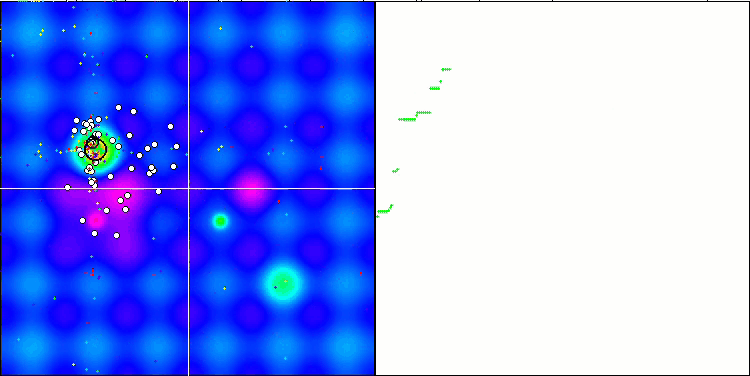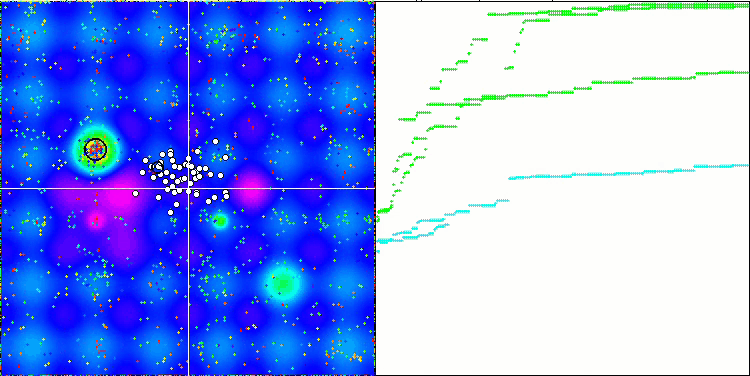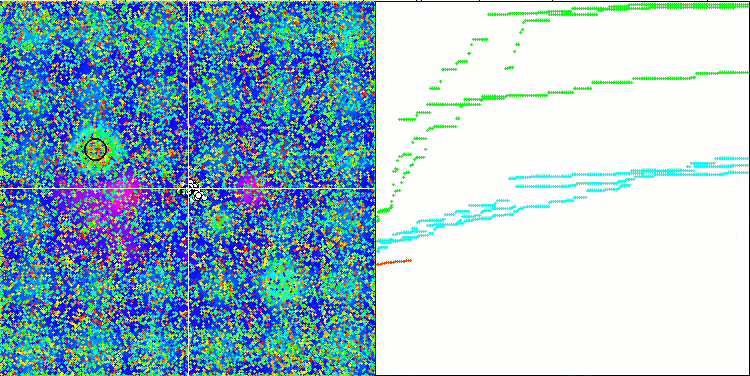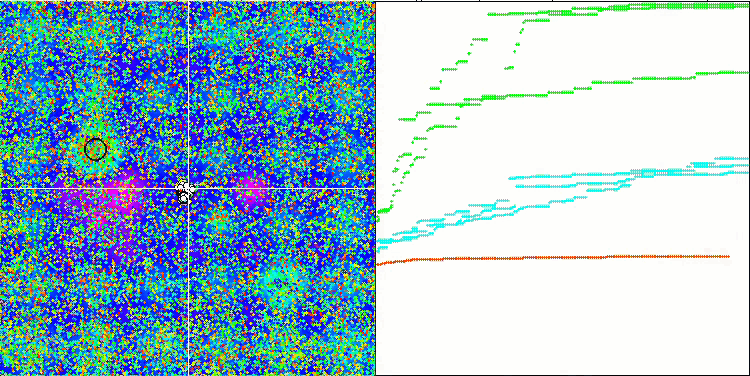
BFO-GA na função de teste Hilly.
BFO-GA na função de teste Forest.
BFO-GA na função de teste Megacity.
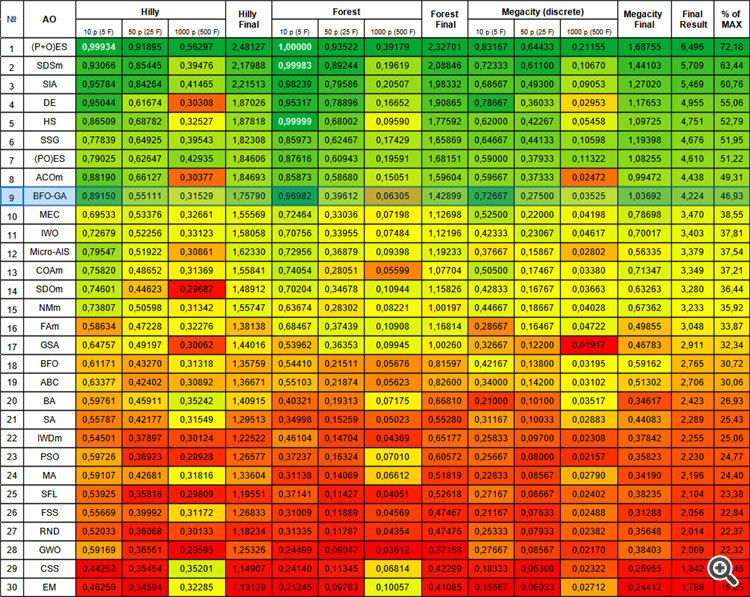
Na tabela de classificação geral, o algoritmo BFO-GA aparece em 9º lugar, o que indica um resultado bastante alto. Isso mostra que as modificações feitas ao adicionar operadores de algoritmo genético ao algoritmo BFO original não foram em vão e levaram a resultados correspondentes. Os melhores resultados deste algoritmo foram demonstrados principalmente em funções de teste com um pequeno número de variáveis, superando a maioria dos outros algoritmos.
| № | AO | Description | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | (P+O)ES | (P+O) evolution strategies | 0,99934 | 0,91895 | 0,56297 | 2,48127 | 1,00000 | 0,93522 | 0,39179 | 2,32701 | 0,83167 | 0,64433 | 0,21155 | 1,68755 | 6,496 | 72,18 |
| 2 | SDSm | stochastic diffusion search M | 0,93066 | 0,85445 | 0,39476 | 2,17988 | 0,99983 | 0,89244 | 0,19619 | 2,08846 | 0,72333 | 0,61100 | 0,10670 | 1,44103 | 5,709 | 63,44 |
| 3 | SIA | simulated isotropic annealing | 0,95784 | 0,84264 | 0,41465 | 2,21513 | 0,98239 | 0,79586 | 0,20507 | 1,98332 | 0,68667 | 0,49300 | 0,09053 | 1,27020 | 5,469 | 60,76 |
| 4 | DE | differential evolution | 0,95044 | 0,61674 | 0,30308 | 1,87026 | 0,95317 | 0,78896 | 0,16652 | 1,90865 | 0,78667 | 0,36033 | 0,02953 | 1,17653 | 4,955 | 55,06 |
| 5 | HS | harmony search | 0,86509 | 0,68782 | 0,32527 | 1,87818 | 0,99999 | 0,68002 | 0,09590 | 1,77592 | 0,62000 | 0,42267 | 0,05458 | 1,09725 | 4,751 | 52,79 |
| 6 | SSG | saplings sowing and growing | 0,77839 | 0,64925 | 0,39543 | 1,82308 | 0,85973 | 0,62467 | 0,17429 | 1,65869 | 0,64667 | 0,44133 | 0,10598 | 1,19398 | 4,676 | 51,95 |
| 7 | (PO)ES | (PO) evolution strategies | 0,79025 | 0,62647 | 0,42935 | 1,84606 | 0,87616 | 0,60943 | 0,19591 | 1,68151 | 0,59000 | 0,37933 | 0,11322 | 1,08255 | 4,610 | 51,22 |
| 8 | ACOm | ant colony optimization M | 0,88190 | 0,66127 | 0,30377 | 1,84693 | 0,85873 | 0,58680 | 0,15051 | 1,59604 | 0,59667 | 0,37333 | 0,02472 | 0,99472 | 4,438 | 49,31 |
| 9 | BFO-GA | bacterial foraging optimization - ga | 0,89150 | 0,55111 | 0,31529 | 1,75790 | 0,96982 | 0,39612 | 0,06305 | 1,42899 | 0,72667 | 0,27500 | 0,03525 | 1,03692 | 4,224 | 46,93 |
| 10 | MEC | mind evolutionary computation | 0,69533 | 0,53376 | 0,32661 | 1,55569 | 0,72464 | 0,33036 | 0,07198 | 1,12698 | 0,52500 | 0,22000 | 0,04198 | 0,78698 | 3,470 | 38,55 |
| 11 | IWO | invasive weed optimization | 0,72679 | 0,52256 | 0,33123 | 1,58058 | 0,70756 | 0,33955 | 0,07484 | 1,12196 | 0,42333 | 0,23067 | 0,04617 | 0,70017 | 3,403 | 37,81 |
| 12 | Micro-AIS | micro artificial immune system | 0,79547 | 0,51922 | 0,30861 | 1,62330 | 0,72956 | 0,36879 | 0,09398 | 1,19233 | 0,37667 | 0,15867 | 0,02802 | 0,56335 | 3,379 | 37,54 |
| 13 | COAm | cuckoo optimization algorithm M | 0,75820 | 0,48652 | 0,31369 | 1,55841 | 0,74054 | 0,28051 | 0,05599 | 1,07704 | 0,50500 | 0,17467 | 0,03380 | 0,71347 | 3,349 | 37,21 |
| 14 | SDOm | spiral dynamics optimization M | 0,74601 | 0,44623 | 0,29687 | 1,48912 | 0,70204 | 0,34678 | 0,10944 | 1,15826 | 0,42833 | 0,16767 | 0,03663 | 0,63263 | 3,280 | 36,44 |
| 15 | NMm | Nelder-Mead method M | 0,73807 | 0,50598 | 0,31342 | 1,55747 | 0,63674 | 0,28302 | 0,08221 | 1,00197 | 0,44667 | 0,18667 | 0,04028 | 0,67362 | 3,233 | 35,92 |
| 16 | FAm | firefly algorithm M | 0,58634 | 0,47228 | 0,32276 | 1,38138 | 0,68467 | 0,37439 | 0,10908 | 1,16814 | 0,28667 | 0,16467 | 0,04722 | 0,49855 | 3,048 | 33,87 |
| 17 | GSA | gravitational search algorithm | 0,64757 | 0,49197 | 0,30062 | 1,44016 | 0,53962 | 0,36353 | 0,09945 | 1,00260 | 0,32667 | 0,12200 | 0,01917 | 0,46783 | 2,911 | 32,34 |
| 18 | BFO | bacterial foraging optimization | 0,61171 | 0,43270 | 0,31318 | 1,35759 | 0,54410 | 0,21511 | 0,05676 | 0,81597 | 0,42167 | 0,13800 | 0,03195 | 0,59162 | 2,765 | 30,72 |
| 19 | ABC | artificial bee colony | 0,63377 | 0,42402 | 0,30892 | 1,36671 | 0,55103 | 0,21874 | 0,05623 | 0,82600 | 0,34000 | 0,14200 | 0,03102 | 0,51302 | 2,706 | 30,06 |
| 20 | BA | bat algorithm | 0,59761 | 0,45911 | 0,35242 | 1,40915 | 0,40321 | 0,19313 | 0,07175 | 0,66810 | 0,21000 | 0,10100 | 0,03517 | 0,34617 | 2,423 | 26,93 |
| 21 | SA | simulated annealing | 0,55787 | 0,42177 | 0,31549 | 1,29513 | 0,34998 | 0,15259 | 0,05023 | 0,55280 | 0,31167 | 0,10033 | 0,02883 | 0,44083 | 2,289 | 25,43 |
| 22 | IWDm | intelligent water drops M | 0,54501 | 0,37897 | 0,30124 | 1,22522 | 0,46104 | 0,14704 | 0,04369 | 0,65177 | 0,25833 | 0,09700 | 0,02308 | 0,37842 | 2,255 | 25,06 |
| 23 | PSO | particle swarm optimisation | 0,59726 | 0,36923 | 0,29928 | 1,26577 | 0,37237 | 0,16324 | 0,07010 | 0,60572 | 0,25667 | 0,08000 | 0,02157 | 0,35823 | 2,230 | 24,77 |
| 24 | MA | monkey algorithm | 0,59107 | 0,42681 | 0,31816 | 1,33604 | 0,31138 | 0,14069 | 0,06612 | 0,51819 | 0,22833 | 0,08567 | 0,02790 | 0,34190 | 2,196 | 24,40 |
| 25 | SFL | shuffled frog-leaping | 0,53925 | 0,35816 | 0,29809 | 1,19551 | 0,37141 | 0,11427 | 0,04051 | 0,52618 | 0,27167 | 0,08667 | 0,02402 | 0,38235 | 2,104 | 23,38 |
| 26 | FSS | fish school search | 0,55669 | 0,39992 | 0,31172 | 1,26833 | 0,31009 | 0,11889 | 0,04569 | 0,47467 | 0,21167 | 0,07633 | 0,02488 | 0,31288 | 2,056 | 22,84 |
| 27 | RND | random | 0,52033 | 0,36068 | 0,30133 | 1,18234 | 0,31335 | 0,11787 | 0,04354 | 0,47476 | 0,25333 | 0,07933 | 0,02382 | 0,35648 | 2,014 | 22,37 |
| 28 | GWO | grey wolf optimizer | 0,59169 | 0,36561 | 0,29595 | 1,25326 | 0,24499 | 0,09047 | 0,03612 | 0,37158 | 0,27667 | 0,08567 | 0,02170 | 0,38403 | 2,009 | 22,32 |
| 29 | CSS | charged system search | 0,44252 | 0,35454 | 0,35201 | 1,14907 | 0,24140 | 0,11345 | 0,06814 | 0,42299 | 0,18333 | 0,06300 | 0,02322 | 0,26955 | 1,842 | 20,46 |
| 30 | EM | electroMagnetism-like algorithm | 0,46250 | 0,34594 | 0,32285 | 1,13129 | 0,21245 | 0,09783 | 0,10057 | 0,41085 | 0,15667 | 0,06033 | 0,02712 | 0,24412 | 1,786 | 19,85 |
Considerações finais
Observamos melhorias claras nos resultados do BFO-GA em comparação com o algoritmo BFO original, devido ao uso dos operadores do algoritmo genético: seleção, mutação e herança. O BFO anteriormente não possuía mecanismos para escapar de extremos locais, e agora esse problema foi resolvido. O algoritmo oferece enormes possibilidades para experimentação, combinando a ordem dos componentes da estratégia de forrageamento bacteriano com os operadores de GA, muitos dos quais ainda não testei.
Anteriormente, cometi um erro no algoritmo BFO ao contar o número de vidas das bactérias, mas esse erro foi corrigido. O BFO melhorou um pouco os resultados e subiu uma posição acima do ABC. Gostaria de destacar que, ao escrever algoritmos de otimização, é difícil detectar erros no código e na lógica da estratégia de busca. Mesmo com erros, o algoritmo quase sempre continua a busca. Os erros nem sempre pioram os resultados; às vezes, os resultados melhoram significativamente, e o "erro" se torna parte da estratégia. É importante lembrar que, nos algoritmos de otimização, grandes mudanças na lógica nem sempre levam a melhorias significativas nos resultados, enquanto pequenas mudanças podem levar a melhorias drásticas. A versão corrigida do BFO está incluída no arquivo anexado ao artigo.
Figura 2. Graduação de cores dos algoritmos nos testes correspondentes.
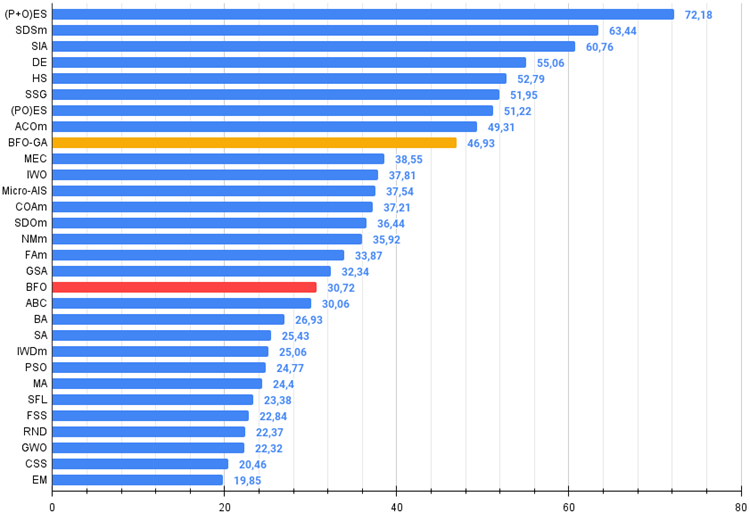
Figura 3. Histograma dos resultados dos testes dos algoritmos (em uma escala de 0 a 100, quanto maior, melhor,
onde 100 é o resultado teórico máximo possível, com o script para cálculo da tabela de classificação incluído no arquivo).
Prós e contras do algoritmo híbrido de otimização de forrageamento bacteriano com algoritmo genético (BFO-GA):
- Alta velocidade de execução do algoritmo.
- Implementação simples.
- Boa convergência em funções com poucos parâmetros.
- Baixa convergência em problemas com grande espaço de busca.
O artigo inclui um arquivo com as versões atualizadas dos códigos dos algoritmos descritos em artigos anteriores. O autor do artigo não se responsabiliza pela precisão absoluta na descrição dos algoritmos canônicos, pois muitos deles foram modificados para melhorar as capacidades de busca. As conclusões e julgamentos apresentados nos artigos se baseiam nos resultados dos experimentos realizados.
Traduzido do russo pela MetaQuotes Ltd.
Artigo original: https://www.mql5.com/ru/articles/14011
 Redes neurais de maneira fácil (Parte 71): Previsão de estados futuros com base em objetivos (GCPC)
Redes neurais de maneira fácil (Parte 71): Previsão de estados futuros com base em objetivos (GCPC)
 Redes neurais de maneira fácil (Parte 70): melhorando a política usando operadores de forma fechada (CFPI)
Redes neurais de maneira fácil (Parte 70): melhorando a política usando operadores de forma fechada (CFPI)
 Desenvolvendo um sistema de Replay (Parte 53): Complicando as coisas (V)
Desenvolvendo um sistema de Replay (Parte 53): Complicando as coisas (V)
 Redes neurais de maneira fácil (Parte 69): restrição de política comportamental com base na densidade de dados off-line (SPOT)
Redes neurais de maneira fácil (Parte 69): restrição de política comportamental com base na densidade de dados off-line (SPOT)
- Aplicativos de negociação gratuitos
- 8 000+ sinais para cópia
- Notícias econômicas para análise dos mercados financeiros
Você concorda com a política do site e com os termos de uso