
Der Optimierungsalgorithmus Brain Storm (Teil II): Multimodalität
Inhalt
1. Einführung
Im ersten Teil des Artikels haben wir uns mit dem Algorithmus Brain Storm Optimization (BSO) in die Welt der Optimierung begeben und die Grundprinzipien dieser innovativen, vom Brainstorming (Ideenfindung) inspirierten Methode vorgestellt. Neben der Untersuchung seiner logischen Struktur haben wir uns auch mit den Clustering-Methoden K-Means und K-Means++ befasst. Brain Storm Optimization (BSO) ist eine Optimierungsmethode, die die Phasen der Ideengenerierung und -bewertung in Gruppenaktivitäten einschließt. Dieser Algorithmus trug zum Bereich der Optimierung in Verbindung mit Clustermethoden bei. Durch Clustering können wir Gruppen ähnlicher Datenelemente identifizieren, was BSO hilft, optimale Lösungen zu finden. Durch den Einsatz der Mutationsmethode kann der Algorithmus Hindernisse im Lösungssuchraum umgehen und nach effizienteren Wegen zum Optimum suchen.
Jetzt ist es an der Zeit, zur Tat zu schreiten! Im zweiten Teil werden wir uns mit der praktischen Umsetzung des Algorithmus befassen, über Multimodalität sprechen, den Algorithmus testen und die Ergebnisse zusammenfassen.
2. Implementierung des Algorithmus
Lassen Sie uns kurz die wichtigsten Punkte der Logik des BSO-Algorithmus skizzieren:
- Clustering.
- Cluster-Verschiebung.
- Auswahl von Ideen aus Clustern: Clusterschwerpunkte oder Ideen aus Clustern.
- Zusammenführung ausgewählter Ideen.
- Abwandlung der in den vorangegangenen Phasen gewonnenen Ideen.
- Auswahl der Ideen für die Stufen 2, 3 und 4. Neue Ideen werden in die Grundgesamtheit eingeordnet und sortiert.
Wir fahren fort mit der Beschreibung des BSO-Algorithmus.
Implementieren wir nun die Struktur des Agentenalgorithmus S_BSO_Agent. Die Struktur wird verwendet, um jeden Agenten im BSO-Algorithmus zu beschreiben.
1. Die Struktur enthält folgende Felder:
- c[] - Array zum Speichern der Koordinaten des Agenten.
- f - Variable zum Speichern der Agentenbewertung (Fitness).
- label - Variable zum Speichern des Clustermitgliedslabels.
2. Init - S_BSO_Agent Strukturmethode, die die Felder der Struktur initialisiert. Sie nimmt das ganzzahlige Argument „coords“, das zur Größenänderung des Koordinatenarrays „c“ mit der Funktion ArrayResize verwendet wird.
3. f = -DBL_MAX - setzt den Anfangswert der Variablen „f“ gleich dem kleinstmöglichen Wert einer Double-Zahl.
4. label = -1 - setzt den Anfangswert der Variable „label“ auf -1, was bedeutet, dass der Agent keinem Cluster angehört.
Dieser Code stellt die grundlegende Datenstruktur für Agenten im BSO-Optimierungsalgorithmus dar und initialisiert ihre Felder, wenn ein neuer Agent erstellt wird.
//—————————————————————————————————————————————————————————————————————————————— struct S_BSO_Agent { double c []; //coordinates double f; //fitness int label; //cluster membership label void Init (int coords) { ArrayResize (c, coords); f = -DBL_MAX; label = -1; } }; //——————————————————————————————————————————————————————————————————————————————
Wir haben die Clustering-Algorithmen K-Means und K-Means++ bereits im vorangegangenen Artikel ausführlich besprochen, sodass wir hier nicht weiter darauf eingehen werden.
Fahren wir mit der Beschreibung der Klasse C_AO_BSO fort, die von der Basisklasse der C_AO-Populationsalgorithmen erbt und die folgenden Felder und Methoden enthält:
1. Öffentliche Felder:
- ao_name - Name des Optimierungsalgorithmus.
- ao_desc - Beschreibung des Optimierungsalgorithmus.
- ao_link - Link zu dem Artikel über den Optimierungsalgorithmus.
- popSize - Größe der Population.
- parentPopSize - Größe der Elternpopulation.
- clustersNumb - Anzahl von Clustern.
- p_Replace - Ersetzungswahrscheinlichkeit.
- p_One - Wahrscheinlichkeit einer einzigen Wahl.
- p_One_center - Wahrscheinlichkeit der Auswahl eines Zentrums oder Individuums im ausgewählten Cluster.
- p_Two_center - Wahrscheinlichkeit der Auswahl von zwei Zentren oder zwei Individuen im ausgewählten Cluster.
- k_Mutation - Mutationsverhältnis.
- distribCoeff - Verteilungsverhältnis.
- agent - Array der Agent.
- parents - Array der Eltern.
- clusters - Array der Cluster.
- km - Objekt der Klasse KMeans.
2. Die verfügbaren Optionen sind:
- SetParams - Methode zur Einstellung der Algorithmusparameter.
- Init - Methode zur Initialisierung des Algorithmus. Die Methode akzeptiert minimale und maximale Suchbereiche, Suchschritte und die Anzahl der Epochen.
- Moving - Methode für die Bewegung der Agenten.
- Revision - Methode zur Revision der Agenten.
3. Private Felder:
- parentsTemp - temporäres Array der Eltern.
- epochs - maximale Anzahl von Epochen.
- epochsNow - aktuelle Epoche.
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BSO : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_BSO () { } C_AO_BSO () { ao_name = "BSO"; ao_desc = "Brain Storm Optimization"; ao_link = "https://www.mql5.com/en/articles/14622"; popSize = 25; //population size parentPopSize = 50; //parent population size; clustersNumb = 5; //number of clusters p_Replace = 0.1; //replace probability p_One = 0.5; //probability of choosing one p_One_center = 0.3; //probability of choosing one center p_Two_center = 0.2; //probability of choosing two centers k_Mutation = 20.0; //mutation coefficient distribCoeff = 1.0; //distribution coefficient ArrayResize (params, 9); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "parentPopSize"; params [1].val = parentPopSize; params [2].name = "clustersNumb"; params [2].val = clustersNumb; params [3].name = "p_Replace"; params [3].val = p_Replace; params [4].name = "p_One"; params [4].val = p_One; params [5].name = "p_One_center"; params [5].val = p_One_center; params [6].name = "p_Two_center"; params [6].val = p_Two_center; params [7].name = "k_Mutation"; params [7].val = k_Mutation; params [8].name = "distribCoeff"; params [8].val = distribCoeff; } void SetParams () { popSize = (int)params [0].val; parentPopSize = (int)params [1].val; clustersNumb = (int)params [2].val; p_Replace = params [3].val; p_One = params [4].val; p_One_center = params [5].val; p_Two_center = params [6].val; k_Mutation = params [7].val; distribCoeff = params [8].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); void Injection (const int popPos, const int coordPos, const double value); //---------------------------------------------------------------------------- int parentPopSize; //parent population size; int clustersNumb; //number of clusters double p_Replace; //replace probability double p_One; //probability of choosing one double p_One_center; //probability of choosing one center double p_Two_center; //probability of choosing two centers double k_Mutation; //mutation coefficient double distribCoeff; //distribution coefficient S_BSO_Agent agent []; S_BSO_Agent parents []; S_Clusters clusters []; S_BSO_KMeans km; private: //------------------------------------------------------------------- S_BSO_Agent parentsTemp []; int epochs; int epochsNow; }; //——————————————————————————————————————————————————————————————————————————————
Die Init-Methode der Klasse C_AO_BSO führt die folgenden Aktionen zur Initialisierung des Optimierungsalgorithmus durch:
- Überprüfung der Initialisierung. Zunächst wird die Methode StandardInit mit den Parametern Suchbereich und Schritt aufgerufen. Wenn StandardInit „false“ zurückgibt, wird die Initialisierung abgebrochen und die Init-Methode gibt „false“ zurück.
- Initialisierung des Agenten. Die Größe des Arrays „agent“ wird auf den Wert „popSize“ geändert. Die Methode Init wird für jeden Agenten aufgerufen, wobei der Parameter „coords“ die Anzahl der Koordinaten angibt.
- Cluster-Initialisierung. Das Array „clusters“ wird auf „clustersNumb“ (die maximale Anzahl von Clustern) verkleinert, und die Methode Init wird für jeden Cluster aufgerufen.
- Initialisierung der Eltern. Die Arrays „parents“ und „parentsTemp“ werden auf „parentPopSize + popSize“ skaliert und die Init-Methode wird für jedes Elternteil aufgerufen. Die Arrays sollten so groß sein, dass sie sowohl die Eltern- als auch die Kindpopulation für die anschließende Sortierung aufnehmen können.
- Einstellung der Epochen. Die Werte „epochs“ und „epochsNow“ werden entsprechend dem übergebenen Parameter „epochsP“ bzw. „0“ gesetzt.
Die Methode gibt „true“ zurück, wenn alle Initialisierungsschritte erfolgreich abgeschlossen sind. Dadurch wird der Algorithmus darauf vorbereitet, die Optimierung für eine bestimmte Anzahl von Epochen durchzuführen.
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_BSO::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (agent, popSize); for (int i = 0; i < popSize; i++) agent [i].Init (coords); ArrayResize (clusters, clustersNumb); for (int i = 0; i < clustersNumb; i++) clusters [i].Init (coords); ArrayResize (parents, parentPopSize + popSize); ArrayResize (parentsTemp, parentPopSize + popSize); for (int i = 0; i < parentPopSize + popSize; i++) { parents [i].Init (coords); parentsTemp [i].Init (coords); } epochs = epochsP; epochsNow = 0; return true; } //——————————————————————————————————————————————————————————————————————————————
Die Methode Moving der Klasse C_AO_BSO wird verwendet, um Agenten während der Optimierung zu bewegen. Die Methode geht folgendermaßen vor:
- Der Wert der aktuellen Epoche („epochsNow++“) wird erhöht.
- Wenn „revision“ „false“ ist, werden die Koordinaten der Agenten mit Zufallswerten in den angegebenen Bereichen initialisiert. Die Methode wird dann beendet.
- Wenn „revision“ nicht „false“ ist, werden für jeden Agenten neue Koordinaten anhand von Gleichungen und Wahrscheinlichkeiten berechnet.
- Verschiedene mathematische Berechnungen, Zufallszahlen und Wahrscheinlichkeiten werden verwendet, um die neuen Koordinaten der Agenten zu bestimmen.
- Die neuen Koordinaten werden entsprechend den Bedingungen und Wahrscheinlichkeiten berechnet.
- Die neuen Koordinaten werden mit der Methode SeInDiSp festgelegt, um die Werte entsprechend den Suchbereichen und -schritten anzupassen.
- Es wird eine neue Idee erzeugt, die das ausgewählte Clusterzentrum (Clusterzentrum-Offset) ersetzt, wenn die Bedingung „u.RNDprobab () < p_Replace“ erfüllt ist.
- Eine Idee aus einem Cluster wird ausgewählt, wenn die Bedingung „u.RNDprobab () < p_One“ erfüllt ist.
- Ideen aus zwei Clustern werden ausgewählt, wenn die Bedingung „u.RNDprobab () < p_One“ nicht erfüllt ist.
- Es kommt zu einer Mutation der Koordinaten des Agenten.
- Neue Koordinaten von Bearbeitern werden gespeichert.
Diese Methode ist für die Aktualisierung der Koordinaten der Agenten im BSO-Optimierungsalgorithmus entsprechend der aktuellen Epoche und Wahrscheinlichkeiten verantwortlich. Wir wollen die entsprechenden Codeabschnitte, die verschiedene Modelle des Agentenverhaltens beschreiben, farblich hervorheben:
- Eine neue Idee entwickeln. Mit jeder neuen Epoche erkunden die Agenten die Nachbarschaft der gefundenen globalen Lösung entsprechend dem „p_Replace“-Verhältnis gründlicher und versuchen, sich dem globalen Optimum zu nähern und die Zentroide zu verschieben.
- Erkundung der Nachbarschaft eines einzelnen Clusters. Unter Berücksichtigung des Verhältnisses „p_One“ erkunden die Agenten die Nachbarschaften eines zufällig ausgewählten Clusters. Die Erkundung aller Bereiche, in denen sich Agenten in der Population befinden, geht also weiter.
- Auswahl von Ideen aus zwei Clustern. Wenn die Bedingung „u.RNDprobab () < p_One“ nicht erfüllt ist, werden Ideen aus zwei zufällig ausgewählten Clustern ausgewählt.
- Mutation. Die Koordinaten der Agenten unterliegen der Mutation, was die Vielfalt der Population gewährleistet und dazu beiträgt, eine vorzeitige Konvergenz zu lokalen Optima zu vermeiden.
- Sicher der Agenten. Nach allen Operationen werden die neuen Agentenkoordinaten für die nächste Iteration gespeichert.
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BSO::Moving () { epochsNow++; //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); agent [i].c [c] = a [i].c [c]; } } return; } //---------------------------------------------------------------------------- //---------------------------------------------------------------------------- int cIndx_1 = 0; //index in the list of non-empty clusters int iIndx_1 = 0; //index in the list of ideas in the cluster int cIndx_2 = 0; //index in the list of non-empty clusters int iIndx_2 = 0; //index in the list of ideas in the cluster double min = 0.0; double max = 0.0; double dist = 0.0; double val = 0.0; double X1 = 0.0; double X2 = 0.0; int clListSize = 0; int clustList []; ArrayResize (clustList, 0, clustersNumb); //---------------------------------------------------------------------------- //let's make a list of non-empty clusters for (int cl = 0; cl < clustersNumb; cl++) { if (clusters [cl].count > 0) { clListSize++; ArrayResize (clustList, clListSize); clustList [clListSize - 1] = cl; } } for (int i = 0; i < popSize; i++) { //========================================================================== //generating a new idea that replaces the selected cluster center (cluster center offset) if (u.RNDprobab () < p_Replace) { cIndx_1 = u.RNDminusOne (clListSize); for (int c = 0; c < coords; c++) { val = clusters [clustList [cIndx_1]].centroid [c]; dist = (rangeMax [c] - rangeMin [c]) * 0.8; min = val - dist; if (min < rangeMin [c]) min = rangeMin [c]; max = val + dist; if (max > rangeMax [c]) max = rangeMax [c]; val = u.GaussDistribution (val, min, max, 3); val = u.SeInDiSp (val, rangeMin [c], rangeMax [c], rangeStep [c]); clusters [clustList [cIndx_1]].centroid [c] = val; } } //========================================================================== //an idea from one cluster is selected if (u.RNDprobab () < p_One) { cIndx_1 = u.RNDminusOne (clListSize); //------------------------------------------------------------------------ if (u.RNDprobab () < p_One_center) //select cluster center { for (int c = 0; c < coords; c++) { a [i].c [c] = clusters [clustList [cIndx_1]].centroid [c]; } } //------------------------------------------------------------------------ else //random idea from the cluster { iIndx_1 = u.RNDminusOne (clusters [clustList [cIndx_1]].count); for (int c = 0; c < coords; c++) { a [i].c [c] = parents [clusters [clustList [cIndx_1]].ideasList [iIndx_1]].c [c]; } } } //========================================================================== //select ideas from two clusters else { if (clListSize == 1) { cIndx_1 = 0; cIndx_2 = 0; } else { if (clListSize == 2) { cIndx_1 = 0; cIndx_2 = 1; } else { cIndx_1 = u.RNDminusOne (clListSize); do { cIndx_2 = u.RNDminusOne (clListSize); } while (cIndx_1 == cIndx_2); } } //------------------------------------------------------------------------ if (u.RNDprobab () < p_Two_center) //two cluster centers selected { for (int c = 0; c < coords; c++) { X1 = clusters [clustList [cIndx_1]].centroid [c]; X2 = clusters [clustList [cIndx_2]].centroid [c]; a [i].c [c] = u.RNDfromCI (X1, X2); } } //------------------------------------------------------------------------ else //two ideas from two selected clusters { iIndx_1 = u.RNDminusOne (clusters [clustList [cIndx_1]].count); iIndx_2 = u.RNDminusOne (clusters [clustList [cIndx_2]].count); for (int c = 0; c < coords; c++) { X1 = parents [clusters [clustList [cIndx_1]].ideasList [iIndx_1]].c [c]; X2 = parents [clusters [clustList [cIndx_2]].ideasList [iIndx_2]].c [c]; a [i].c [c] = u.RNDfromCI (X1, X2); } } } //========================================================================== //Mutation for (int c = 0; c < coords; c++) { int x = (int)u.Scale (epochsNow, 1, epochs, 1, 200); double ξ = (1.0 / (1.0 + exp (-((100 - x) / k_Mutation))));// * u.RNDprobab (); double dist = (rangeMax [c] - rangeMin [c]) * distribCoeff * ξ; double min = a [i].c [c] - dist; if (min < rangeMin [c]) min = rangeMin [c]; double max = a [i].c [c] + dist; if (max > rangeMax [c]) max = rangeMax [c]; val = a [i].c [c]; a [i].c [c] = u.GaussDistribution (val, min, max, 8); } //Save the agent----------------------------------------------------------- for (int c = 0; c < coords; c++) { val = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); a [i].c [c] = val; agent [i].c [c] = val; } } } //——————————————————————————————————————————————————————————————————————————————
Die Hauptaufgabe der Methode Revision der Klasse C_AO_BSO besteht darin, die globale Lösung zu aktualisieren und Lösungscluster zu bilden. Die Methode geht folgendermaßen vor:
- Fit werden. Für jeden Agenten in der Population wird ein Fitnesswert extrahiert und in dem entsprechenden Feld der Agentenstruktur gespeichert.
- Neue Ideen an die Population weitergeben. Neue Ideen (Agenten), die während des Optimierungsprozesses entstehen, werden der Grundgesamtheit hinzugefügt.
- Sortierung der Grundgesamtheit. Die Elternpopulation wird nach Fitness sortiert. So können nur die besten Lösungen an der Schaffung neuer Ideen für die nächste Epoche teilnehmen.
- Suche nach der besten Lösung. Wenn die Fitness des besten Agenten in der Elternpopulation die aktuelle beste Lösung übersteigt, werden die beste Lösung und ihre Koordinaten aktualisiert.
- Durchführen des Clustering. Wenn dies die erste Iteration ist, wird der K-Means-Algorithmus mit der übergeordneten Population und den Clustern initialisiert. Anschließend wird der K-Means-Algorithmus zum Clustern der Grundgesamtheit eingesetzt.
- Zuweisung der besten Clusterlösung als Clusterzentrum. Für jeden Cluster wird geprüft, ob er Agenten hat (Cluster können leer sein). Wenn dies der Fall ist, wird geprüft, ob jeder Agent in der Elternpopulation zu dem Cluster gehört. Wenn die Agentenfitness die aktuelle Clusterfitness übersteigt, werden die Clusterfitness und der Clusterschwerpunkt aktualisiert (der Schwerpunkt nimmt an der Erstellung neuer Ideen teil).
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BSO::Revision () { //get fitness-------------------------------------------------- for (int i = 0; i < popSize; i++) { agent [i].f = a [i].f; } //pass new ideas to the population-------------------------------------------- for (int i = parentPopSize; i < parentPopSize + popSize; i++) { parents [i] = agent [i - parentPopSize]; } //sort out the parent population---------------------------------------- u.Sorting (parents, parentsTemp, parentPopSize + popSize); if (parents [0].f > fB) { fB = parents [0].f; ArrayCopy (cB, parents [0].c, 0, 0, WHOLE_ARRAY); } //perform clustering----------------------------------------------------- if (!revision) { km.KMeansInit (parents, parentPopSize, clusters); revision = true; } km.KMeansInit (parents, parentPopSize, clusters); km.KMeans (parents, parentPopSize, clusters); //Assign the best cluster solution as the cluster center-------------------------- for (int cl = 0; cl < clustersNumb; cl++) { clusters [cl].f = -DBL_MAX; if (clusters [cl].count > 0) { for (int p = 0; p < parentPopSize; p++) { if (parents [p].label == cl) { if (parents [p].f > clusters [cl].f) { clusters [cl].f = parents [p].f; ArrayCopy (clusters [cl].centroid, parents [p].c, 0, 0, WHOLE_ARRAY); } } } } } }//——————————————————————————————————————————————————————————————————————————————
Was die Multimodalität betrifft, so wurde der BSO-Algorithmus ursprünglich als Optimierungsmethode für die Lösung multimodaler Probleme eingeführt. Die Testergebnisse zeigten jedoch, dass signifikante lokale Extremwerte von diesem Algorithmus nicht ausreichend untersucht werden und viele von ihnen unbemerkt bleiben. Meine derzeitige Umsetzung ist vielleicht nicht die optimalste. Daher habe ich beschlossen, der Anpassungsfähigkeit von Agenten im Zusammenhang mit K-Means-Clustering mehr Aufmerksamkeit zu schenken. Dies erforderte einige Änderungen am Clustering-Algorithmus.
Wie Sie sich vielleicht erinnern, bedeutet Multimodalität im Rahmen von Optimierungsalgorithmen, dass die zu optimierende Funktion mehrere optimale Punkte oder Spitzenwerte aufweist. Solche Funktionen können mehrere lokale Optima enthalten, die entweder in der Nähe des globalen Optimums in Bezug auf den Wert der Fitnessfunktion liegen oder im Rahmen des zu lösenden Problems von Bedeutung sind. Clustering kann helfen, verschiedene Regionen im Suchraum hervorzuheben, in denen das Merkmal unterschiedliche Modalitäten aufweist.
Versuchen wir also, den Einfluss der Agentenfitness auf die Clusterbildung zu verstärken. Wir verpacken die Funktion zur Berechnung der Entfernungen zwischen Agenten in die neue Funktion FitnessDistance. Es wird einen zusätzlichen Parameter „alpha“ geben, der als Verhältnis der Signifikanz zwischen Entfernung und Fitness fungiert.
Die Funktion FitnessDistance berechnet den Abstand zwischen einem Agenten und einem Clusterschwerpunkt, wobei sowohl der Abstand als auch der Unterschied in der Fitnessfunktion zwischen ihnen berücksichtigt wird. Dazu wird eine gewichtete Summe aus dem Abstand und dem Absolutwert der Differenz zwischen der Fitnessfunktion des Agenten und dem Schwerpunkt berechnet. Das Gewicht „Alpha“ bestimmt die relative Bedeutung des Abstands im Vergleich zum Unterschied in der Fitnessfunktion.
//—————————————————————————————————————————————————————————————————————————————— double FitnessDistance (S_BSO_Agent &data, S_Cluster &clust, double alpha) { double distance = VectorDistance (data.c, clust.centroid); double fitness_diff = fabs (data.f - clust.f); return alpha * distance + (1 - alpha) * fitness_diff; } //——————————————————————————————————————————————————————————————————————————————
Die KMeans-Methode wird durch den Parameter „alpha“ ergänzt:
void KMeans (S_BSO_Agent &data [], int dataSizeClust, S_Cluster &clust [], double alpha)
Ändern wir den Codeabschnitt der KMeans-Methode, der für die Aktualisierung der Zentroide zuständig ist, sodass jeder Cluster den maximalen Fitnesswert eines Individuums aufweist, das Teil des Clusters ist.
// Update the centroids double sum_c []; ArrayResize (sum_c, ArraySize (data [0].c)); double sum_f = 0.0; for (int cl = 0; cl < nClusters; cl++) { ArrayInitialize (sum_c, 0.0); clust [cl].count = 0; ArrayResize (clust [cl].ideasList, 0); sum_f = -DBL_MAX; for (int d = 0; d < dataSizeClust; d++) { if (data [d].label == cl) { for (int k = 0; k < ArraySize (data [d].c); k++) { sum_c [k] += data [d].c [k]; } if (data [d].f > sum_f) sum_f = data [d].f; clust [cl].count++; ArrayResize (clust [cl].ideasList, clust [cl].count); clust [cl].ideasList [clust [cl].count - 1] = d; } } if (clust [cl].count > 0) { for (int k = 0; k < ArraySize (sum_c); k++) { clust [cl].centroid [k] = sum_c [k] / clust [cl].count; } } }
Die vorgenommenen Änderungen ermöglichen die Berücksichtigung der Fitnessfunktion bei der Clusterbildung, führten aber nicht zu einer spürbaren Verbesserung der Zuordnung der einzelnen Modi in der Fitnessfunktion und hatten keinen Einfluss auf die Ergebnisse. Dies könnte darauf zurückzuführen sein, dass die Verwendung einer Fitnessfunktion im Clustering-Prozess nicht immer effizient ist, zumindest nicht in dieser Implementierung von BSO.
Wenn K-Means und K-Means++ nicht die gewünschten Ergebnisse liefern, können wir andere Clustermethoden ausprobieren:
1. Dichtebasiertes, räumliches Clustering für verrauschte Anwendungen (DBSCAN) - die Clustermethode basiert auf der Dichte und nicht auf der Entfernung. Es fasst Punkte zusammen, die im Merkmalsraum nahe beieinander liegen und eine ausreichende Anzahl von Nachbarn haben. DBSCAN ist einer der am häufigsten verwendeten Clustering-Algorithmen.
2. Hierarchisches Clustering baut eine Hierarchie von Clustern auf, wobei jedes Cluster mit den beiden nächstgelegenen Clustern verbunden ist. Hierarchisches Clustering kann agglomerativ (bottom-up) oder divisional (top-down) sein.
3. Gaussian mixture Modell (GMM) - dieses statistische Modell geht davon aus, dass alle beobachteten Daten aus einer Mischung mehrerer Gauß'scher Verteilungen erzeugt werden, deren Parameter unbekannt sind. Jedes Cluster entspricht einer dieser Verteilungen.
4. Das spektrale Clustering verwendet die Eigenvektoren der Ähnlichkeitsmatrix, um die Dimensionalität der Daten zu reduzieren, bevor sie in einem niedrigdimensionalen Raum geclustert werden.
Es gibt eine ganze Reihe von Clustermethoden, mit denen man versuchen kann, weitere Forschungen in diesem Bereich durchzuführen. Wenn Sie experimentierfreudig sind, können Sie die K-Means-Methode im beigefügten Code durch eine beliebige andere Methode ersetzen.
3. Testergebnisse
Ergebnisse des BSO-Algorithmus:
BSO|Brain Storm Optimization|25.0|50.0|5.0|0.1|0.5|0.3|0.2|20.0|1.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.9301770731803266
25 Hilly's; Func runs: 10000; result: 0.5801719580773876
500 Hilly's; Func runs: 10000; result: 0.30916005647304245
=============================
5 Forest's; Func runs: 10000; result: 0.929981802038364
25 Forest's; Func runs: 10000; result: 0.5907047167619348
500 Forest's; Func runs: 10000; result: 0.2477599978259004
=============================
5 Megacity's; Func runs: 10000; result: 0.5246153846153847
25 Megacity's; Func runs: 10000; result: 0.2784615384615384
500 Megacity's; Func runs: 10000; result: 0.1253384615384627
=============================
All score: 4.51637 (50.18%)
Die Ergebnisse der Prüfung des Algorithmus auf Testfunktionen (die Gesamtpunktzahl von 4,51637 entspricht 50,18 % des maximal möglichen Wertes) zeigen, dass die Verwendung der in der ersten Zeile der Ausdrucke angegebenen Parameter recht gute Ergebnisse liefert. Die Werte der Funktionsergebnisse liegen im Bereich von 0,125 für 1000 optimierte Parameter bzw. bis zu 0,93 für 10, was darauf hindeutet, dass der Algorithmus bei der Suche nach optimalen Lösungen recht erfolgreich ist.
Ich möchte gesondert darauf hinweisen, wie das Clustering der Lösungen in der Visualisierung aussieht. Dieser Prozess ist besonders bei Funktionen mit einer maximalen Anzahl von Parametern zu beobachten, da sich aus dem anfänglichen Chaos mit jeder abgeschlossenen Iteration die charakteristischen Abschnitte der Cluster immer deutlicher abzeichnen.
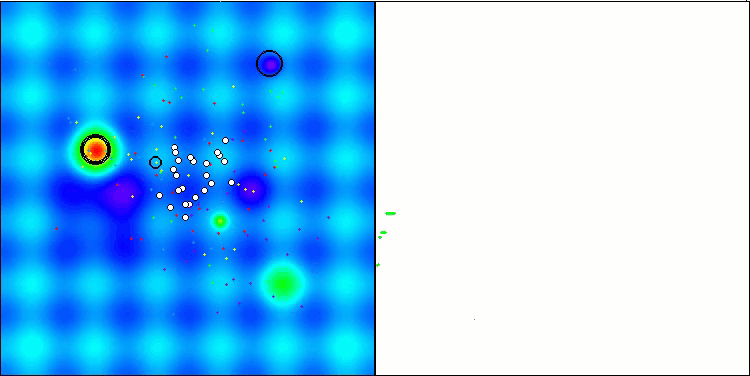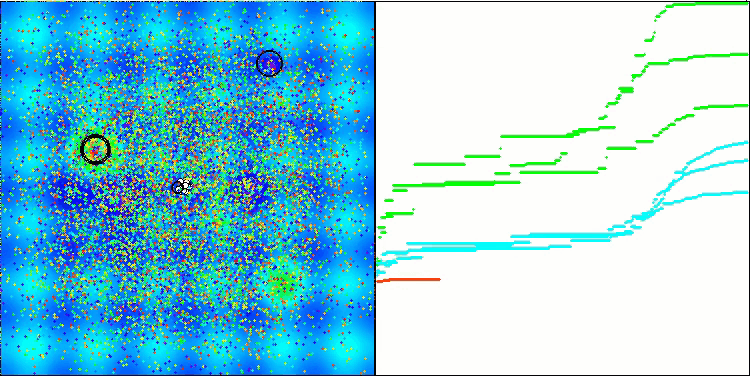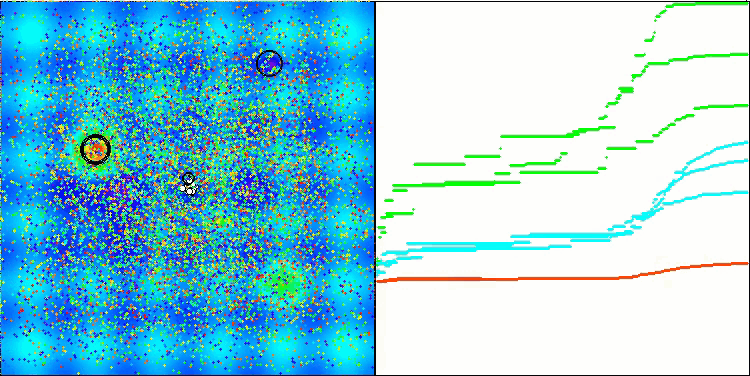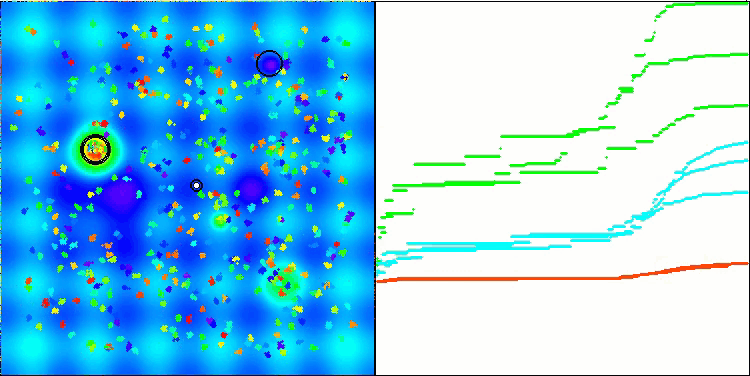
BSO mit der Testfunktion Hilly.
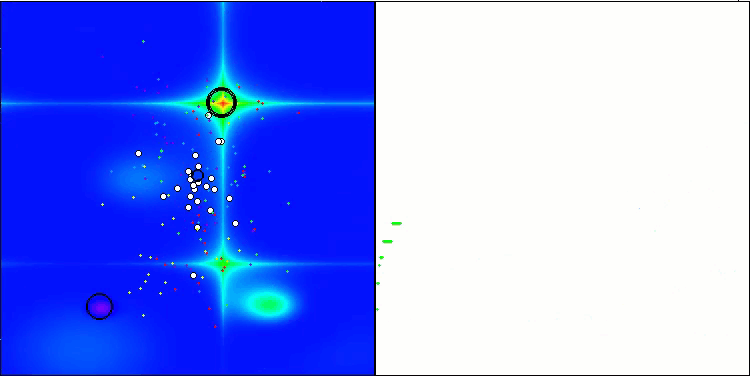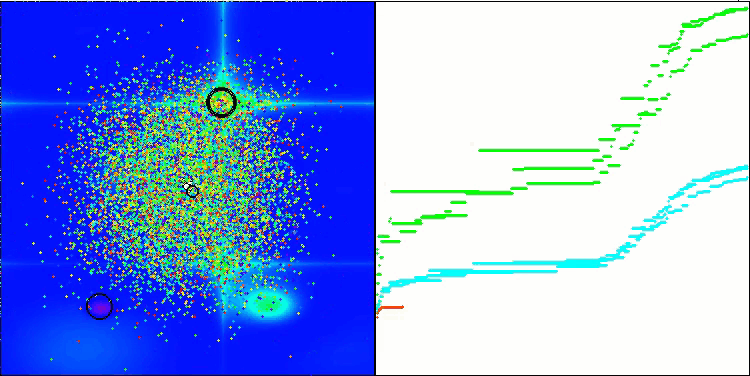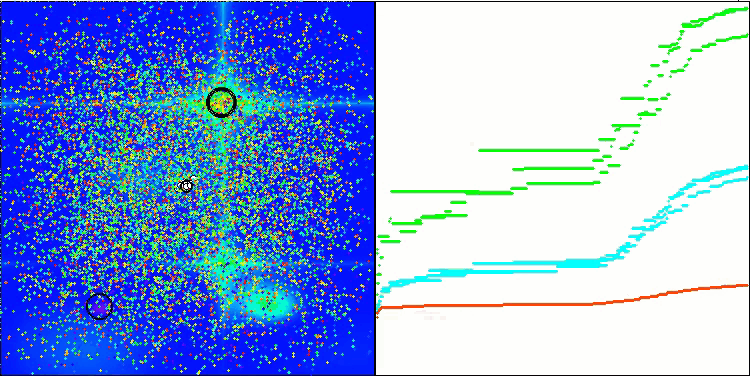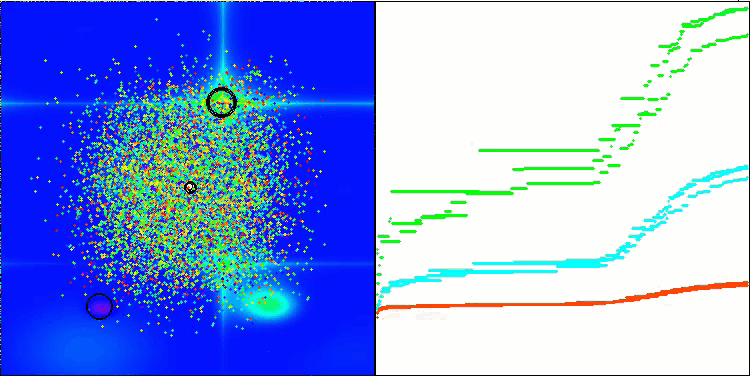
BSO mit der Testfunktion Forest.
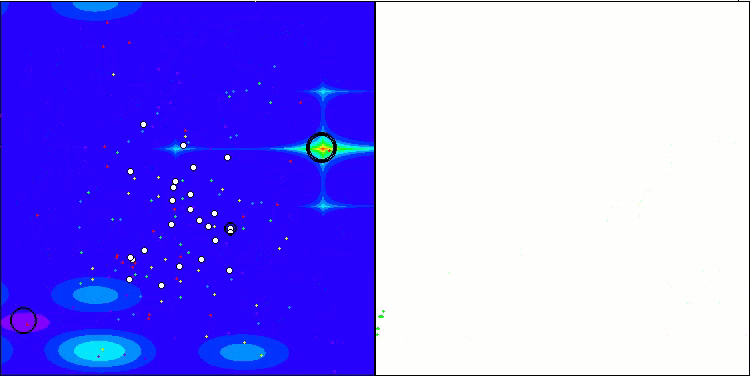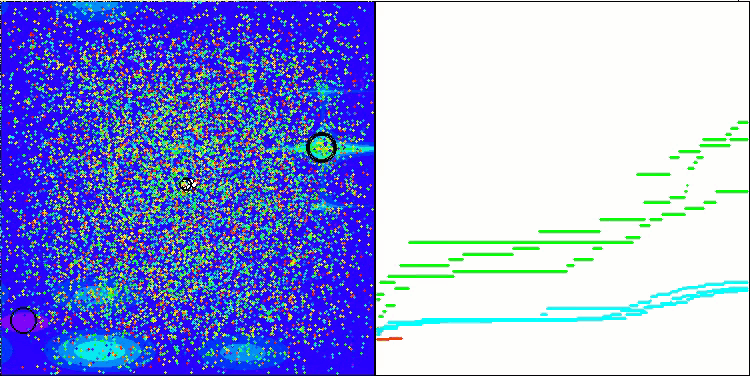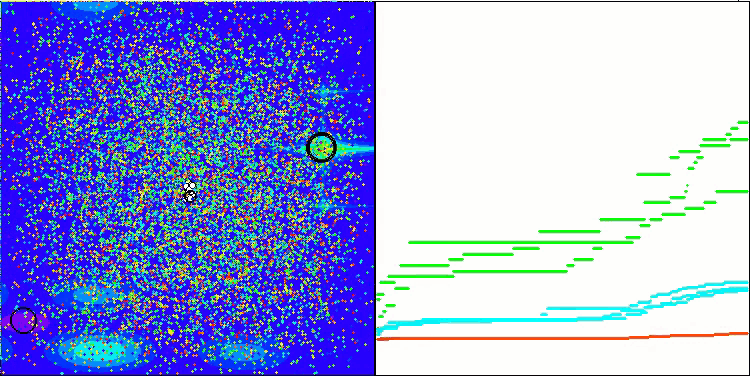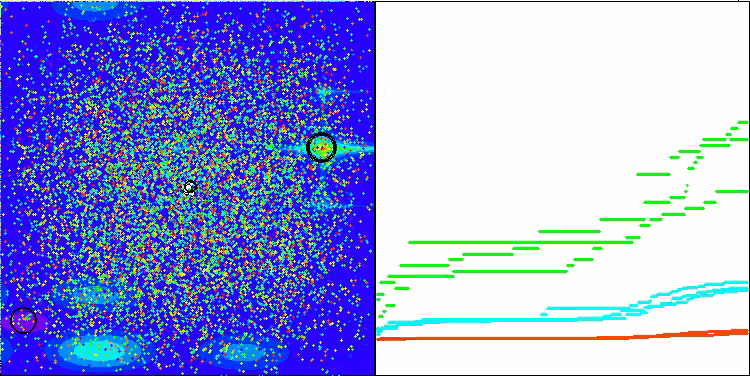
BSO mit der Testfunktion Megacity.
Ich hatte hohe Erwartungen an diesen Algorithmus und hoffte, ihn an der Spitze der Rangliste zu sehen. Schließlich ist es das erste Mal, dass ich die Clustermethode in Kombination mit der Mutationsmethode verwendet habe, die einzigartige Ergebnisse liefern sollte. Ich war etwas enttäuscht, als ich sah, dass der Algorithmus nur an der Spitze der Rangliste stand, aber nicht an der Spitze. BSO zeigt bei den Funktionen Wald und Megacity mit 1000 Parametern hervorragende Ergebnisse, die den Tabellenführern durchaus würdig sind.
# | AO | Beschreibung | Hilly | Hilly final | Forest | Forest final | Megacity (discrete) | Megacity final | Final result | % of MAX | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | BGA | binärer genetischer Algorithmus | 0.99992 | 0.99484 | 0.50483 | 2.49959 | 1.00000 | 0.99975 | 0.32054 | 2.32029 | 0.90667 | 0.96400 | 0.23035 | 2.10102 | 6.921 | 76.90 |
| 2 | (P+O)ES | (P+O) Entwicklungsstrategien | 0.99934 | 0.91895 | 0.56297 | 2.48127 | 1.00000 | 0.93522 | 0.39179 | 2.32701 | 0.83167 | 0.64433 | 0.21155 | 1.68755 | 6.496 | 72.18 |
| 3 | SDSm | stochastische Diffusionssuche M | 0.93066 | 0.85445 | 0.39476 | 2.17988 | 0.99983 | 0.89244 | 0.19619 | 2.08846 | 0.72333 | 0.61100 | 0.10670 | 1.44103 | 5.709 | 63.44 |
| 4 | ESG | Entwicklung der sozialen Gruppen | 0.99906 | 0.79654 | 0.35056 | 2.14616 | 1.00000 | 0.82863 | 0.13102 | 1.95965 | 0.82333 | 0.55300 | 0.04725 | 1.42358 | 5.529 | 61.44 |
| 5 | SIA | Simuliertes isotropes Abkühlen | 0.95784 | 0.84264 | 0.41465 | 2.21513 | 0.98239 | 0.79586 | 0.20507 | 1.98332 | 0.68667 | 0.49300 | 0.09053 | 1.27020 | 5.469 | 60.76 |
| 6 | DE | differentielle Evolution | 0.95044 | 0.61674 | 0.30308 | 1.87026 | 0.95317 | 0.78896 | 0.16652 | 1.90865 | 0.78667 | 0.36033 | 0.02953 | 1.17653 | 4.955 | 55.06 |
| 7 | BSA | Vogelschwarm-Algorithmus | 0.90857 | 0.73661 | 0.25767 | 1.90285 | 0.90437 | 0.81619 | 0.16401 | 1.88457 | 0.61692 | 0.54154 | 0.10951 | 1.26797 | 5.055 | 56.17 |
| 8 | HS | Harmoniesuche | 0.86509 | 0.68782 | 0.32527 | 1.87818 | 0.99999 | 0.68002 | 0.09590 | 1.77592 | 0.62000 | 0.42267 | 0.05458 | 1.09725 | 4.751 | 52.79 |
| 9 | SSG | Setzen, Säen und Wachsen | 0.77839 | 0.64925 | 0.39543 | 1.82308 | 0.85973 | 0.62467 | 0.17429 | 1.65869 | 0.64667 | 0.44133 | 0.10598 | 1.19398 | 4.676 | 51.95 |
| 10 | (PO)ES | (PO) Entwicklungsstrategien | 0.79025 | 0.62647 | 0.42935 | 1.84606 | 0.87616 | 0.60943 | 0.19591 | 1.68151 | 0.59000 | 0.37933 | 0.11322 | 1.08255 | 4.610 | 51.22 |
| 11 | BSO | Brainstorming-Optimierung | 0.91301 | 0.56222 | 0.30047 | 1.77570 | 0.97162 | 0.57162 | 0.23449 | 1,77772 | 0.60462 | 0.27138 | 0.12011 | 0.99611 | 4.550 | 50.55 |
| 12 | WOAm | Wal-Optimierungsalgorithmus M | 0.84521 | 0.56298 | 0.26263 | 1.67081 | 0.93100 | 0.52278 | 0.16365 | 1.61743 | 0.66308 | 0.41138 | 0.11357 | 1.18803 | 4.476 | 49.74 |
| 13 | ACOm | Ameisen-Kolonie-Optimierung M | 0.88190 | 0.66127 | 0.30377 | 1.84693 | 0.85873 | 0.58680 | 0.15051 | 1.59604 | 0.59667 | 0.37333 | 0.02472 | 0.99472 | 4.438 | 49.31 |
| 14 | BFO-GA | Optimierung der bakteriellen Futtersuche - ga | 0.89150 | 0.55111 | 0.31529 | 1.75790 | 0.96982 | 0.39612 | 0.06305 | 1.42899 | 0.72667 | 0.27500 | 0.03525 | 1.03692 | 4.224 | 46.93 |
| 15 | MEC | Evolutionäre Berechnung des Geistes | 0.69533 | 0.53376 | 0.32661 | 1.55569 | 0.72464 | 0.33036 | 0.07198 | 1.12698 | 0.52500 | 0.22000 | 0.04198 | 0.78698 | 3.470 | 38.55 |
| 16 | IWO | Optimierung mit invasiven Unkräutern | 0.72679 | 0.52256 | 0.33123 | 1.58058 | 0.70756 | 0.33955 | 0.07484 | 1.12196 | 0.42333 | 0.23067 | 0.04617 | 0.70017 | 3.403 | 37.81 |
| 17 | Micro-AIS | Künstliches Mikro-Immunsystem | 0.79547 | 0.51922 | 0.30861 | 1.62330 | 0.72956 | 0.36879 | 0.09398 | 1.19233 | 0.37667 | 0.15867 | 0.02802 | 0.56335 | 3.379 | 37.54 |
| 18 | COAm | Kuckuck-Optimierungsalgorithmus M | 0.75820 | 0.48652 | 0.31369 | 1.55841 | 0.74054 | 0.28051 | 0.05599 | 1.07704 | 0.50500 | 0.17467 | 0.03380 | 0.71347 | 3.349 | 37.21 |
| 19 | SDOm | Optimierung der Spiraldynamik M | 0.74601 | 0.44623 | 0.29687 | 1.48912 | 0.70204 | 0.34678 | 0.10944 | 1.15826 | 0.42833 | 0.16767 | 0.03663 | 0.63263 | 3.280 | 36.44 |
| 20 | NMm | Nelder-Mead-Verfahren M | 0.73807 | 0.50598 | 0.31342 | 1.55747 | 0.63674 | 0.28302 | 0.08221 | 1.00197 | 0.44667 | 0.18667 | 0.04028 | 0.67362 | 3.233 | 35.92 |
| 21 | FAm | Firefly-Algorithmus M | 0.58634 | 0.47228 | 0.32276 | 1.38138 | 0.68467 | 0.37439 | 0.10908 | 1.16814 | 0.28667 | 0.16467 | 0.04722 | 0.49855 | 3.048 | 33.87 |
| 22 | GSA | Algorithmus für die Schwerkraftsuche | 0.64757 | 0.49197 | 0.30062 | 1.44016 | 0.53962 | 0.36353 | 0.09945 | 1.00260 | 0.32667 | 0.12200 | 0.01917 | 0.46783 | 2.911 | 32.34 |
| 23 | BFO | Optimierung der bakteriellen Futtersuche | 0.61171 | 0.43270 | 0.31318 | 1.35759 | 0.54410 | 0.21511 | 0.05676 | 0.81597 | 0.42167 | 0.13800 | 0.03195 | 0.59162 | 2.765 | 30.72 |
| 24 | ABC | Künstliches Bienenvolk (Artificial Bee Colony, ABC) | 0.63377 | 0.42402 | 0.30892 | 1.36671 | 0.55103 | 0.21874 | 0.05623 | 0.82600 | 0.34000 | 0.14200 | 0.03102 | 0.51302 | 2.706 | 30.06 |
| 25 | BA | Fledermaus-Algorithmus | 0.59761 | 0.45911 | 0.35242 | 1.40915 | 0.40321 | 0.19313 | 0.07175 | 0.66810 | 0.21000 | 0.10100 | 0.03517 | 0.34617 | 2.423 | 26.93 |
| 26 | SA | simuliertes Abkühlen | 0.55787 | 0.42177 | 0.31549 | 1.29513 | 0.34998 | 0.15259 | 0.05023 | 0.55280 | 0.31167 | 0.10033 | 0.02883 | 0.44083 | 2.289 | 25.43 |
| 27 | IWDm | intelligente Wassertropfen M | 0.54501 | 0.37897 | 0.30124 | 1.22522 | 0.46104 | 0.14704 | 0.04369 | 0.65177 | 0.25833 | 0.09700 | 0.02308 | 0.37842 | 2.255 | 25.06 |
| 28 | PSO | Partikelschwarmoptimierung | 0.59726 | 0.36923 | 0.29928 | 1.26577 | 0.37237 | 0.16324 | 0.07010 | 0.60572 | 0.25667 | 0.08000 | 0.02157 | 0.35823 | 2.230 | 24.77 |
| 29 | Gebote | Boids-Algorithmus | 0.43340 | 0.30581 | 0.25425 | 0.99346 | 0.35718 | 0.20160 | 0.15708 | 0.71586 | 0.27846 | 0.14277 | 0.09834 | 0.51957 | 2.229 | 24.77 |
| 30 | MA | Affen-Algorithmus | 0.59107 | 0.42681 | 0.31816 | 1.33604 | 0.31138 | 0.14069 | 0.06612 | 0.51819 | 0.22833 | 0.08567 | 0.02790 | 0.34190 | 2.196 | 24.40 |
| 31 | SFL | schlurfender Froschsprung | 0.53925 | 0.35816 | 0.29809 | 1.19551 | 0.37141 | 0.11427 | 0.04051 | 0.52618 | 0.27167 | 0.08667 | 0.02402 | 0.38235 | 2.104 | 23.38 |
| 32 | FSS | Fischschulsuche | 0.55669 | 0.39992 | 0.31172 | 1.26833 | 0.31009 | 0.11889 | 0.04569 | 0.47467 | 0.21167 | 0.07633 | 0.02488 | 0.31288 | 2.056 | 22.84 |
| 33 | RND | zufällig | 0.52033 | 0.36068 | 0.30133 | 1.18234 | 0.31335 | 0.11787 | 0.04354 | 0.47476 | 0.25333 | 0.07933 | 0.02382 | 0.35648 | 2.014 | 22.37 |
| 34 | GWO | Grauer-Wolf-Optimierung | 0.59169 | 0.36561 | 0.29595 | 1.25326 | 0.24499 | 0.09047 | 0.03612 | 0.37158 | 0.27667 | 0.08567 | 0.02170 | 0.38403 | 2.009 | 22.32 |
| 35 | CSS | Suche geladener Systeme | 0.44252 | 0.35454 | 0.35201 | 1.14907 | 0.24140 | 0.11345 | 0.06814 | 0.42299 | 0.18333 | 0.06300 | 0.02322 | 0.26955 | 1.842 | 20.46 |
| 36 | EM | elektromagnetismusähnlicher Algorithmus | 0.46250 | 0.34594 | 0.32285 | 1.13129 | 0.21245 | 0.09783 | 0.10057 | 0.41085 | 0.15667 | 0.06033 | 0.02712 | 0.24412 | 1.786 | 19.85 |
Zusammenfassung
Der BSO-Algorithmus hat mehrere Vorteile, darunter Flexibilität, Ausgewogenheit zwischen Erkundung und Ausbeutung und Anpassungsfähigkeit an verschiedene Optimierungsprobleme.
Die Effizienz des Algorithmus hängt jedoch stark von den Einstellungen der externen Parameter ab (die Anzahl der externen Parameter ist der größte Nachteil von BSO), sodass sorgfältige Untersuchungen und Experimente erforderlich sind, um die optimalen Einstellungen für jede spezifische Aufgabe zu ermitteln.
Ich ermutige alle Optimierungsbegeisterten, sich an den Experimenten zu beteiligen und gemeinsam die Fähigkeiten des Algorithmus bei der Lösung praktischer Probleme zu erkunden. Wenn jemand weitere interessante Ergebnisse und bessere externe Parameter findet, kann er sie in den Kommentaren zu diesem Artikel mitteilen.
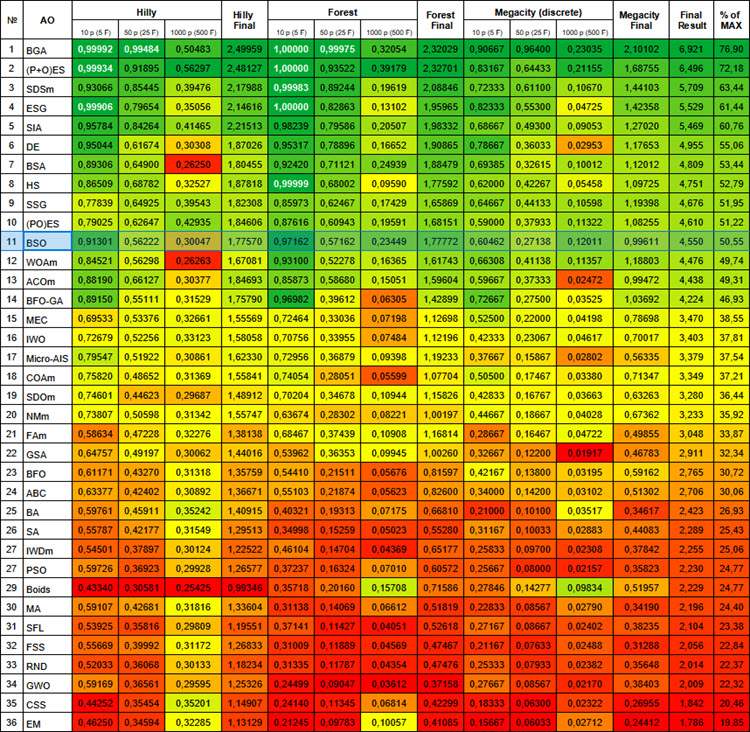
Abbildung 1. Die farblichen Abstufungen der Algorithmen nach relevanten Tests Ergebnisse größer oder gleich 0,99 sind weiß hervorgehoben
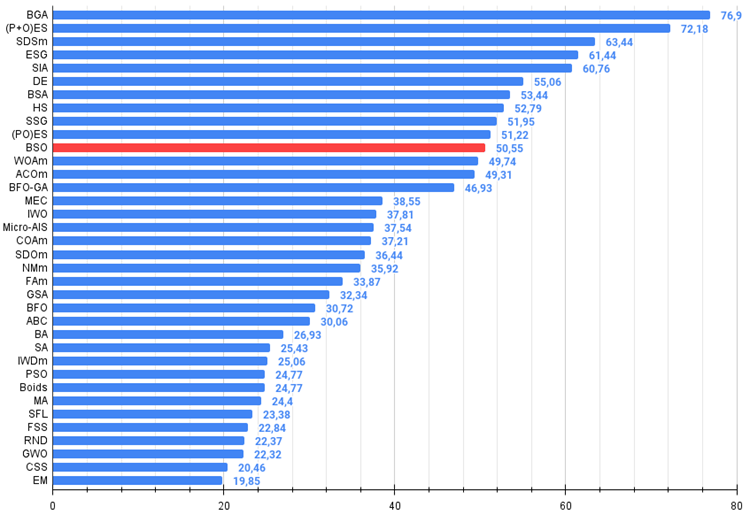
Bild 2. Das Histogramm der Algorithmus-Testergebnisse (auf einer Skala von 0 bis 100, größer ist besser,
wobei 100 das maximal mögliche theoretische Ergebnis ist; das Archiv enthält ein Skript zur Berechnung der Bewertungstabelle)
Vor- und Nachteile von BSO:
Vorteile:
- Gute Ergebnisse für die scharfe Forest-Funktion und die diskrete Megacity mit großer Dimension.
Nachteile
- Eine sehr große Anzahl von externen Parametern.
- Komplexe Architektur und Implementierung.
- Hohe Belastung der Computerressourcen.
Der Artikel wird von einem Archiv mit den aktuellen Versionen der Algorithmuscodes begleitet. Der Autor des Artikels übernimmt keine Verantwortung für die absolute Richtigkeit der Beschreibung der kanonischen Algorithmen. An vielen von ihnen wurden Änderungen vorgenommen, um die Suchmöglichkeiten zu verbessern. Die in den Artikeln dargelegten Schlussfolgerungen und Urteile beruhen auf den Ergebnissen der Versuche.
Übersetzt aus dem Russischen von MetaQuotes Ltd.
Originalartikel: https://www.mql5.com/ru/articles/14622
 Die Übertragung der Trading-Signale in einem universalen Expert Advisor.
Die Übertragung der Trading-Signale in einem universalen Expert Advisor.
 Neuronale Netze leicht gemacht (Teil 84): Umkehrbare Normalisierung (RevIN)
Neuronale Netze leicht gemacht (Teil 84): Umkehrbare Normalisierung (RevIN)
 Eine alternative Log-datei mit der Verwendung der HTML und CSS
Eine alternative Log-datei mit der Verwendung der HTML und CSS
 Brain Storm Optimierungsalgorithmus (Teil I): Clustering
Brain Storm Optimierungsalgorithmus (Teil I): Clustering
- Freie Handelsapplikationen
- Über 8.000 Signale zum Kopieren
- Wirtschaftsnachrichten für die Lage an den Finanzmärkte
Sie stimmen der Website-Richtlinie und den Nutzungsbedingungen zu.