细菌趋化优化(BCO)
内容
引言
在优化领域,许多研究人员和开发者从自然界的生物过程汲取灵感,例如进化、社会互动或生物体觅食行为。这促使了全新创新优化方法的开发。研究表明,这些方法在解决多模态、不可微和离散问题时,往往优于经典的启发式和基于梯度的方法。其中一种方法是趋化算法,最初由Bremermann等人提出。我们已经讨论过用于优化细菌觅食行为的算法(BFO)。它模拟细菌对浓度梯度中趋化因子的响应。Bremermann分析了三维梯度中的趋化现象,并将其应用于训练神经网络。尽管该算法基于细菌趋化原理,但新的生物学发现使其能够创建更详细的模型。
在本文中,我们将尝试将这一模型视为一种新的优化策略。新模型与传统趋化算法的主要区别如下:
- 新模型中的细菌利用浓度值信息。
- 它们不会随着趋化因子浓度的增加而持续朝一个方向移动。
细菌是单细胞生物,是地球上最简单的生命形式之一。尽管结构简单,但它们能够感知环境信息、导航并有效利用这些信息以求生存。近年来,细菌对环境变化的响应一直是密集研究的主题,也吸引了优化领域科学家的关注。优化算法可以被视为收集函数相关信息并利用其达到最优的系统。细菌趋化的简单性使其颇具吸引力,成为构建此类算法的起点。
各种研究表明,细菌会相互交流信息,尽管其通信机制尚不清楚。通常,细菌被视为个体,模型中未考虑其社交互动。这使它们与描述社会性昆虫(如蚂蚁、蜜蜂、黄蜂或白蚁)行为的互动模型不同,后者作为具有集体智能的系统,为解决各种问题提供了不同的可能性。
适应性是趋化的另一个重要方面。细菌能够改变对恒定化学条件的敏感性,从而有效响应环境变化。这一特性不仅使它们具有很强的适应性,还使其在寻找资源方面极为高效。
在本研究中,作者专注于微观模型,关注单个细菌的趋化行为,而不是分析群体运动的宏观模型。该算法由S. D. Müller和P. Koumatsakas开发,其主要思想于2002年提出并发表。
算法的实现
细菌趋化优化算法(BCO)的核心思想是利用细菌行为中观察到的生物学原理来解决优化问题。该算法模拟细菌对其环境中化学梯度的响应,使其能够找到更适宜的生存条件。算法的主要思想如下:
- 算法将细菌的运动描述为一系列由瞬间转弯连接的直线轨迹。每次运动都具有速度、方向和持续时间的特征。
- 细菌旋转的方向由概率分布决定,这使得运动中的随机变化得以被考虑进来。
- 算法利用关于函数梯度的信息引导细菌朝最优解前进,从而减少达到目标所需的迭代次数。
细菌趋化优化(BCO)算法的详细伪代码描述了算法的主要步骤,包括初始化、包含运动和修正步骤的主要优化循环以及辅助函数。
初始化。
1. 设置参数:
- popSize — 种群大小(细菌数量)
- hs — 用于计算平均变化的步数
- T0 — 初始温度
- b — 参数
- tau_c — 参数
- v — 速度
2. 创建一个包含popSize个细菌的数组。
3. 对于每个索引为i的细菌(从0到popSize-1):
- 将fPrev初始化为-DBL_MAX
- 创建一个大小为hs的fHistory数组,并用零填充。
基本优化循环。重复执行,直到满足停止条件:
每个索引为i的细菌的运动阶段(从0到popSize - 1):
1. 获取当前目标函数f_tr的值
2. 从细菌[i].fPrev中获取目标函数f_pr的前一个值
3. 如果f_tr <= f_pr:T = T0
否则:计算b_corr = CalculateCorrectedB(f_tr, f_pr, i)
- T = T0 * exp (b_corr * (f_tr - f_pr))
4. 从参数为T的指数分布中生成“tau”。
5. 为coords - 1维计算新的角度new_angles[]:对于每个角度j(从0到coords - 2):
- theta = CalculateRotationAngle ()
- mu = 62 * (1 - cos (theta)) * π / 180
- sigma = 26 * (1 - cos (theta)) * π / 180
- 从参数为(mu, mu-π, mu+π)的高斯分布中生成new_angles[j]
6. 计算新位置:
- l = v * tau
- CalculateNewPosition (l, new_angles, new_position, current_position)
7. 更新细菌位置,将值限制在rangeMin和rangeMax之间
8. 用f_tr的值更新细菌[i].fPrev
修正阶段。
1. 用fB适应度值更新全局最优解cB
2. 对于每个索引为i的细菌(从0到popSize-1):更新目标函数值的历史记录fHistory:
- 将所有值向左移动一个位置
- 将目标函数的当前值添加到历史记录的末尾
辅助函数:
CalculateCorrectedB (f_tr, f_pr, bacteriumIndex)
1. 计算delta_f_tr = f_tr - f_pr
2. 计算delta_f_pr = 最近hs步的平均变化
3. 如果|delta_f_pr| < epsilon:返回b
否则:返回b * (1 / (|delta_f_tr / delta_f_pr| + 1) + 1 / (|f_pr| + 1))
CalculateRotationAngle ()
1. 计算cos_theta = exp(-tau_c / T0)
2. 返回arccos (cos_theta)
CalculateNewPosition (l, angles, new_position, current_position)
1. 根据所有角度计算new_position[0]
2. 对于每个坐标i(从1到coords - 1):
根据对应角度计算new_position[i]
3. 为每个坐标应用随机方向(1或-1)
GenerateFromExponentialDistribution (T)
返回-T * ln(0到1之间的随机数)
接下来,我们开始编写算法代码。为了将细菌表示为优化问题的解,描述S_BCO_Bacterium结构。
1. 结构字段:
- fPrev - 目标函数的前一个值。
- fHistory [] - 目标函数值历史记录数组。
2. Init - 初始化方法执行以下操作:
- 将 fHistory 数组的大小调整为 historySize。
- 将 fHistory 数组的所有元素初始化为 0.0。
- 将 fPrev - 目标函数的前一个值设置为可能的最小值。
以结构体形式表示的细菌,用于跟踪目标函数值在给定迭代次数(单独移动)内的变化。
//—————————————————————————————————————————————————————————————————————————————— struct S_BCO_Bacterium { double fPrev; // previous value of the objective function double fHistory []; // history of objective function values void Init (int coords, int historySize) { ArrayResize (fHistory, historySize); ArrayInitialize (fHistory, 0.0); fPrev = -DBL_MAX; } }; //——————————————————————————————————————————————————————————————————————————————
描述 C_AO_BCO 算法类。让我们一步步分解。
1. 算法的外部参数在构造函数中初始化。
2. SetParams 方法从 params 数组中更新参数值。
3. Moving 和 Revision 方法负责细菌的移动及其位置的修正。
4. 该类定义了几个用于算法相关各种计算的私有方法。"CalculateAverageDeltaFpr", "CalculateNewAngles", "CalculateNewPosition", "GenerateFromExponentialDistribution", "CalculateCorrectedB", "CalculateRotationAngle", "RNDdir", bacterium - array of bacteria (population). 类的参数:
- hs — 用于计算平均变化的步数。
- T0 — 初始温度。
- b - 参数。
- tau_c - 参数。
- v - 速度。
//—————————————————————————————————————————————————————————————————————————————— class C_AO_BCO : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_BCO () { } C_AO_BCO () { ao_name = "BCO"; ao_desc = "Bacterial Chemotaxis Optimization"; ao_link = "https://www.mql5.com/en/articles/15711"; popSize = 50; // population size (number of bacteria) hs = 10; // number of steps to calculate average change T0 = 1.0; // initial temperature b = 0.5; // parameter b tau_c = 1.0; // parameter tau_c v = 1.0; // velocity ArrayResize (params, 6); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "hs"; params [1].val = hs; params [2].name = "T0"; params [2].val = T0; params [3].name = "b"; params [3].val = b; params [4].name = "tau_c"; params [4].val = tau_c; params [5].name = "v"; params [5].val = v; } void SetParams () { popSize = (int)params [0].val; hs = (int)params [1].val; T0 = params [2].val; b = params [3].val; tau_c = params [4].val; v = params [5].val; } bool Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0); //number of epochs void Moving (); void Revision (); //---------------------------------------------------------------------------- int hs; double T0; double b; double tau_c; double v; S_BCO_Bacterium bacterium []; private: //------------------------------------------------------------------- double CalculateAverageDeltaFpr (int bacteriumIndex); void CalculateNewAngles (double &angles []); void CalculateNewPosition (double l, const double &angles [], double &new_position [], const double ¤t_position []); double GenerateFromExponentialDistribution (double T); double CalculateCorrectedB (double f_tr, double f_pr, int bacteriumIndex); double CalculateRotationAngle (); int RNDdir (); }; //—————————————————————————————————————————————————————————————————————————————— bool C_AO_BCO::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (bacterium, popSize); for (int i = 0; i < popSize; i++) bacterium [i].Init (coords, hs); return true; } //——————————————————————————————————————————————————————————————————————————————
Moving 方法是 C_AO_BCO 类中负责细菌在搜索空间中移动的关键部分。让我们看看它是如何工作的:
1. 如果 revision 为 false,这意味着细菌处于初始位置。
- 对于每个 a[i] 细菌,会在给定范围内生成随机坐标。
- u.RNDfromCI 函数生成随机值,而 u.SeInDiSp 函数会根据指定的步长对其进行调整。
2. 如果 revision 为 true,则进入基本移动循环。定义 T 温度:
- 如果当前目标函数值 f_tr 不低于之前的值 f_pr,则使用初始温度 T0。
- 否则,通过 CalculateCorrectedB 函数调整温度,该函数会考虑适应度函数当前值与前一个值之间的差异。
- 生成 tau 移动时间:使用指数分布来生成移动时间。
- 根据移动长度 l 和新角度计算新的移动角度和新位置。
- 新位置会根据指定的范围和步长进行调整。
- 在循环结束时,会更新每个细菌的适应度函数前一个值。
Moving 方法实现了细菌在搜索空间中移动的基本逻辑,根据前一次迭代的结果调整其行为,以寻找最优解。它结合了随机元素和自适应机制。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCO::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } revision = true; return; } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) { double f_tr = a [i].f; double f_pr = bacterium [i].fPrev; double T; if (f_tr <= f_pr) { T = T0; } else { double b_corr = CalculateCorrectedB (f_tr, f_pr, i); T = T0 * exp (b_corr * (f_tr - f_pr)); } double tau = GenerateFromExponentialDistribution (T); double new_angles []; ArrayResize (new_angles, coords - 1); CalculateNewAngles (new_angles); double l = v * tau; double new_position []; ArrayResize (new_position, coords); CalculateNewPosition (l, new_angles, new_position, a [i].c); for (int c = 0; c < coords; c++) { a [i].c [c] = u.SeInDiSp (new_position [c], rangeMin [c], rangeMax [c], rangeStep [c]); } bacterium [i].fPrev = a [i].f; } } //——————————————————————————————————————————————————————————————————————————————
Revision 方法是 C_AO_BCO 类中负责更新关于找到的最佳解的信息以及每个细菌适应度函数值历史记录的部分。
1. ind 变量被初始化为 -1。它将被用来存储找到的最佳函数值的细菌的索引。
2. 寻找最佳函数值:
- 该方法遍历 popSize 种群中的所有细菌,寻找函数值 f 大于当前最佳值 fB 的细菌。
- 如果找到具有更高函数值的细菌,则更新 fB,并将该细菌的索引保存到 ind 中。
3. 如果存在索引为 ind 的细菌(“ind”不等于 -1),则将该细菌的坐标复制到 cB 数组中,该数组表示当前最佳解的坐标。
4. 对于每个细菌,更新其函数值的历史记录。该方法遍历每个 fHistory 元素,将值向左移动一个位置,为新值腾出空间。在每次迭代的末尾,fHistory 数组的最后一个元素将获得每个细菌的当前适应度值 a[i].f。
因此,Revision 方法执行了两个主要动作:
- 更新最佳适应度函数值及其对应的坐标。
- 更新每个细菌的适应度函数值历史记录,允许在其移动历史过程中跟踪其状态的变化。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCO::Revision () { int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) { ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); } for (int i = 0; i < popSize; i++) { for (int j = 1; j < hs; j++) { bacterium [i].fHistory [j - 1] = bacterium [i].fHistory [j]; } bacterium [i].fHistory [hs - 1] = a [i].f; } } //——————————————————————————————————————————————————————————————————————————————
CalculateAverageDeltaFpr 方法是 C_AO_BCO 类中用于根据适应度值的历史记录,计算特定细菌在其两个相邻位置之间适应度函数值变化(delta)的平均值。
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BCO::CalculateAverageDeltaFpr (int bacteriumIndex) { double sum = 0; for (int i = 1; i < hs; i++) { sum += bacterium [bacteriumIndex].fHistory [i] - bacterium [bacteriumIndex].fHistory [i - 1]; } return sum / (hs - 1); } //——————————————————————————————————————————————————————————————————————————————
CalculateNewAngles 方法是 C_AO_BCO 类中用于根据与旋转和分布相关的逻辑计算新角度的部分,并执行以下操作:
- 遍历新角度数组并为每个角度计算一个新值。
- 使用依赖于角度余弦的参数来生成使用高斯分布的值。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCO::CalculateNewAngles (double &angles []) { for (int i = 0; i < coords - 1; i++) { double theta = CalculateRotationAngle (); double mu = 62 * (1 - MathCos (theta)) * M_PI / 180.0; double sigma = 26 * (1 - MathCos (theta)) * M_PI / 180.0; angles [i] = u.GaussDistribution (mu, mu - M_PI, mu + M_PI, 8); } } //——————————————————————————————————————————————————————————————————————————————
CalculateNewPosition 方法是 C_AO_BCO 类中用于根据当前坐标、角度和参数 l 计算新位置坐标的部分。
1. 方法的输入参数:
- l - 影响位置变化的比例因子。
- angles [] - 用于计算新位置的角度数组。
- new_position [] - 设置新坐标的数组。
- current_position [] - 当前坐标的数组。
2. 第一个坐标 new_position[0] 被计算为当前坐标 current_position[0] 与 l 乘以 rangeMax[0] 和 rangeMin[0] 之差的乘积之和。
3. 然后,第一个坐标乘以从 angles 数组的第一个到倒数第二个角度的余弦值。
4. 结果乘以 RNDdir() 函数返回的值,该函数生成随机方向“-1”或“1”。
5. 对于每个后续坐标 new_position[i](其中 i 从 1 到 coords - 2),根据当前位置和对应角度的正弦值计算新位置。
6. 每个新坐标也乘以从当前索引 i 开始到倒数第二个角度的余弦值。
7. 对于剩余的坐标,结果也乘以 RNDdir() 返回的值,以确定随机方向。
8. 处理最后一个坐标,如果坐标数量大于 1,则对于最后一个坐标 new_position[coords - 1],根据当前位置和最后一个角度的正弦值计算新位置。
因此,CalculateNewPosition 方法执行以下操作:
- 根据当前坐标和角度计算新坐标。
- 考虑每个坐标上的随机方向的影响。
- 使用三角函数(正弦和余弦)来考虑角度。
因此,该方法用于模拟细菌在空间中的运动,考虑其当前位置和给定的角度。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCO::CalculateNewPosition (double l, const double &angles [], double &new_position [], const double ¤t_position []) { new_position [0] = current_position [0] + l * (rangeMax [0] - rangeMin [0]); for (int k = 0; k < coords - 1; k++) { new_position [0] *= MathCos (angles [k]); } new_position [0] *= RNDdir (); for (int i = 1; i < coords - 1; i++) { new_position [i] = current_position [i] + l * MathSin (angles [i - 1]) * (rangeMax [0] - rangeMin [0]); for (int k = i; k < coords - 1; k++) { new_position [i] *= MathCos (angles [k]); } new_position [i] *= RNDdir (); } if (coords > 1) { new_position [coords - 1] = current_position [coords - 1] + l * MathSin (angles [coords - 2]); } } //——————————————————————————————————————————————————————————————————————————————
接下来,我们将简要描述 C_AO_BCO 类中的 RNDdir 方法,该方法用于生成随机方向,其值可以是 -1 或 1。
//—————————————————————————————————————————————————————————————————————————————— int C_AO_BCO::RNDdir () { if (u.RNDbool () < 0.5) return -1; return 1; } //——————————————————————————————————————————————————————————————————————————————
让我们快速了解一下 C_AO_BCO 类的方法。
GenerateFromExponentialDistribution 方法:
- 使用参数为 T 的指数分布生成随机数。
- 然后,它使用范围在 (0, 1) 内的随机数,计算其对数并乘以 -T。
- 我们得到一个符合指数分布的随机数。
CalculateCorrectedB 方法:
- 根据 f_tr 和 f_pr(当前和前一个适应度)之间的差异计算调整后的 b 值。
- 计算 f_tr 和 f_pr 之间的差异,获取细菌的平均值,并返回调整后的 b 值。
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BCO::GenerateFromExponentialDistribution (double T) { return -T * MathLog (u.RNDprobab ()); } double C_AO_BCO::CalculateCorrectedB (double f_tr, double f_pr, int bacteriumIndex) { double delta_f_tr = f_tr - f_pr; double delta_f_pr = CalculateAverageDeltaFpr (bacteriumIndex); if (MathAbs (delta_f_pr) < DBL_EPSILON) { return b; } else { return b * (1 / (MathAbs (delta_f_tr / delta_f_pr) + 1) + 1 / (MathAbs (f_pr) + 1)); } } //——————————————————————————————————————————————————————————————————————————————
CalculateRotationAngle 方法是 C_AO_BCO 类中的最后一个方法。该方法根据给定参数计算旋转角度,并以弧度返回值。
//—————————————————————————————————————————————————————————————————————————————— double C_AO_BCO::CalculateRotationAngle () { double cos_theta = MathExp (-tau_c / T0); return MathArccos (cos_theta); } //——————————————————————————————————————————————————————————————————————————————
让我们测试算法的原始版本,并查看结果:
=============================
5 Hilly's; Func runs: 10000; result: 0.42924491510564006
25 Hilly's; Func runs: 10000; result: 0.282259866768426
500 Hilly's; Func runs: 10000; result: 0.2515386629014219
=============================
5 Forest's; Func runs: 10000; result: 0.2476662231845009
25 Forest's; Func runs: 10000; result: 0.17824381036550777
500 Forest's; Func runs: 10000; result: 0.15324081202657283
=============================
5 Megacity's; Func runs: 10000; result: 0.2430769230769231
25 Megacity's; Func runs: 10000; result: 0.11415384615384619
500 Megacity's; Func runs: 10000; result: 0.09444615384615461
=============================
All score: 1.99387 (22.15%)
获得的结果如此之糟,以至于没有将其包含在评级表中的必要。在根据作者的文字描述设计算法和编写代码时,我不得不调整一些方程:有些方程在数学上没有意义(例如,用一个数字除以一个向量),而另一些则退化为极小或极大的值,这些值与问题的量纲无法比较。在这样做的过程中,我打印出了每个呈现的方程的函数结果,并对其进行了分析。因此,该算法无法以作者描述的形式运行。
此外,使用正态分布的操作角度可以大大简化,因为这些操作的物理意义归结为在每个坐标上,可以从细菌的当前位置简单地设置一个新的增量。因此,我决定开发自己的细菌趋化算法实现,尽可能贴近算法的基本概念和思想。
以下是算法的伪代码:
初始化:
1. 创建一个包含 popSize 个细菌的种群
2. 对于每个 i 细菌:
初始化目标函数值的历史记录 f_history[i],大小为 hs
将初始值 f_prev[i] 设置为 -DBL_MAX
主循环:
直到满足停止条件:
1. 如果这是第一次迭代:
对于每个 i 细菌:
在搜索空间中随机初始化 x[i] 位置
对于每个 j 坐标 x[i].[j] ∈ [x_min[j], x_max[j]]
2. 否则:
对于每个 i 细菌:
a. 计算目标函数的平均变化:
delta_ave [i] = (1 / (hs - 1)) * sum (f_history [i].[k] - f_history [i].[k-1] for k in 1..hs-1) + epsilon
b. 计算适应度的相对变化:
delta [i] = 1 - |f (x [i]) - f_prev [i]| / delta_ave [i]
delta [i] = max (delta [i], 0.0001)
c. 对于每个 j 坐标:
以 0.5 的概率:
dist [j] = (x_max [j] - x_min [j]) * delta [i]
x [i].[j] = N (x [i].[j], x [i].[j] - dist [j], x [i].[j] + dist [j])
将 x[i].[j] 限制在 [x_min[j], x_max[j]] 范围内
否则:
x [i].[j] = x_best [j]
d. 更新 f_prev[i] = f(x[i])
3. 对每个细菌评估目标函数 f(x[i])
4. 更新找到的最佳解:
如果存在 i:f(x[i]) > f(x_best),则 x_best = x[i]
5. 更新每个细菌的目标函数值历史记录:
在 f_history[i] 中移动值
添加新值:f_history[i].[hs - 1] = f(x[i])
完成:
返回找到的最佳解 x_best
其中:
- x[i] - 第 i 个细菌的位置
- f(x) - 目标函数
- hs - 历史记录大小
- epsilon - 一个小常数,用于防止除以零
- N(μ, a, b) - 均值为 μ,范围为 [a, b] 的截断正态分布
因此,我的修改后的伪代码反映了 BCO 算法的基本结构和逻辑。让我们关注主要要点:
- 该算法使用一个细菌种群,每个细菌代表一个潜在的解。
- 在每次迭代中,细菌利用其先前结果的信息在搜索空间中移动。
- 移动基于目标函数的相对变化,这使得算法能够适应优化情形。
- 该算法为每个细菌存储目标函数值的历史记录,用于计算平均变化。
- 有一定概率细菌会朝已知的最佳解移动,而不是探索新的区域(类似于遗传学中性状的遗传)。
- 在每次移动后,评估新位置并更新找到的最佳解。
BCOm 版本结合了局部搜索(单个细菌的移动)和全局搜索(通过已知的最佳解交换信息)的元素。
让我们看看两个版本算法的主要区别。首先,细菌移动机制已简化。我放弃了复杂的旋转角度和温度系统。其次,新版本较少依赖变化历史来调整细菌行为,采用更直接的方法更新位置。代替指数分布和正态分布的组合,新算法使用正态分布来更新坐标。这些变化的结果是将控制算法行为的参数数量减少到一个(不包括种群大小),这改变了优化过程的配置方式,并简化了管理。
总体而言,新的伪代码假设在每个优化步骤中进行更简单的计算,这也应该对算法执行任务的速度产生积极影响(原始版本对每个细菌的每个坐标执行多次旋转角度的正弦和余弦计算)。我的方法稍有不同,在探索新的解空间和利用已找到的良好解之间取得平衡。
这些逻辑上的变化导致了一个更简单且(待测试结果确认)更高效的算法。让我们继续编写代码。
代表每个细菌的 S_BCO_Bacterium 结构保持不变。它旨在存储有关细菌及其目标函数值历史记录的信息。
在负责初始化算法参数的 C_AO_BCOm 类的 Init 方法中,我们将添加沿每个坐标可接受移动距离的定义。
因此,C_AO_BCOm 类的 Init 方法负责初始化优化算法的参数。它检查标准初始化条件,创建必要的数组并用值填充它们。
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_BCOm::Init (const double &rangeMinP [], //minimum search range const double &rangeMaxP [], //maximum search range const double &rangeStepP [], //step search const int epochsP = 0) //number of epochs { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (bacterium, popSize); for (int i = 0; i < popSize; i++) bacterium [i].Init (coords, hs); ArrayResize (allowedDispl, coords); for (int c = 0; c < coords; c++) allowedDispl [c] = rangeMax [c] - rangeMin [c]; return true; } //——————————————————————————————————————————————————————————————————————————————
让我们看看 C_AO_BCOm 类中负责细菌在搜索空间中移动的 Moving 方法。让我们一步步分解。
1. 在第一次迭代中,方法的行动没有改变:用给定范围内的随机值初始化细菌坐标。
2. 移动的基本算法是声明变量以存储值,例如 Δ、ΔAve 和 dist,以及种群中的每个个体。
- 使用 CalculateAverageDeltaFpr(i) 函数计算平均值 ΔAve。
- 计算用于确定细菌位置变化程度的相对变化 Δ。
- 如果 Δ 太小,则将其设置为 0.0001。
3. 改变坐标:
- 对每个坐标检查随机概率(50%)。
- 如果满足条件,则根据 allowedDispl[c] 和 Δ 计算 dist。
- 使用函数 GaussDistribution 并考虑 xMin 和 xMax 边界来计算新值 x。
- 如果 x 超出范围,则使用 RNDfromCI 进行修正。
- 最后,存储考虑 rangeStep 的新坐标值。
4. 在 bacterium 数组中为每个个体保存适应度函数的前一个值 f。使用了 a 和 bacterium 数组。RNDfromCI、SeInDiSp 和 GaussDistribution 函数负责生成随机数和分布,以及归一化坐标值。
因此,Moving() 函数负责在算法中初始化和更新种群中个体的位置。它使用随机概率和分布来控制细菌的移动。然而,与原始版本的关键区别在于以更简单、更高效的方式实现适应度函数的梯度。当细菌在当前步骤的养料与前一步相比减少时,其移动速度加快。相反,在有利的外部环境中,细菌会减速。这与细菌的自然行为相矛盾,因为在恶劣环境中,细菌会变得沮丧并进入休眠状态。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_BCOm::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } revision = true; return; } //---------------------------------------------------------------------------- double x = 0.0; double xMin = 0.0; double xMax = 0.0; double Δ = 0.0; double ΔAve = 0.0; double dist = 0.0; for (int i = 0; i < popSize; i++) { ΔAve = CalculateAverageDeltaFpr (i) + DBL_EPSILON; Δ = fabs (a [i].f - bacterium [i].fPrev) / ΔAve; Δ = 1.0 - Δ; if (Δ < 0.0001) Δ = 0.0001; for (int c = 0; c < coords; c++) { if (u.RNDprobab () < 0.5) { dist = allowedDispl [c] * Δ; x = a [i].c [c]; xMin = x - dist; xMax = x + dist; x = u.GaussDistribution (x, xMin, xMax, 8); if (x > rangeMax [c]) x = u.RNDfromCI (xMin, rangeMax [c]); if (x < rangeMin [c]) x = u.RNDfromCI (rangeMin [c], xMax); a [i].c [c] = u.SeInDiSp (x, rangeMin [c], rangeMax [c], rangeStep [c]); } else a [i].c [c] = cB [c]; } bacterium [i].fPrev = a [i].f; } } //——————————————————————————————————————————————————————————————————————————————
C_AO_BCOm 类中负责更新种群信息和目标函数值历史记录的 Revision() 方法没有改变。
测试结果
现在让我们看看 BCOm 算法新版本的表现:
BCO|Bacterial Chemotaxis Optimization|50.0|10.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.759526049526603
25 Hilly's; Func runs: 10000; result: 0.6226756163411526
500 Hilly's; Func runs: 10000; result: 0.31483373090540534
=============================
5 Forest's; Func runs: 10000; result: 0.8937814268120954
25 Forest's; Func runs: 10000; result: 0.6133909133246214
500 Forest's; Func runs: 10000; result: 0.22541842289630293
=============================
5 Megacity's; Func runs: 10000; result: 0.653846153846154
25 Megacity's; Func runs: 10000; result: 0.42092307692307684
500 Megacity's; Func runs: 10000; result: 0.14435384615384755
=============================
All score: 4.64875 (51.65%)
显然,对原始版本算法的经验积累与现有信息的审慎分析发挥了关键作用,使得新版算法在实际应用场景中展现出比原版更强大的性能。
在BCOm算法的可视化结果中,可以清晰看到其对超空间(hyperspace)重要区域的细致刻画,这表明该算法具备深入研究优化函数表面特性的强大能力。
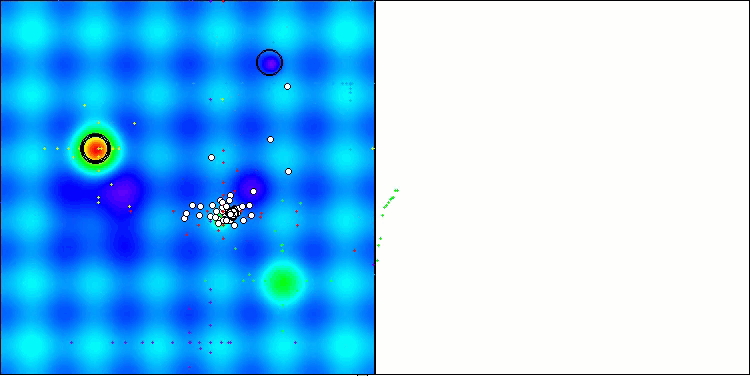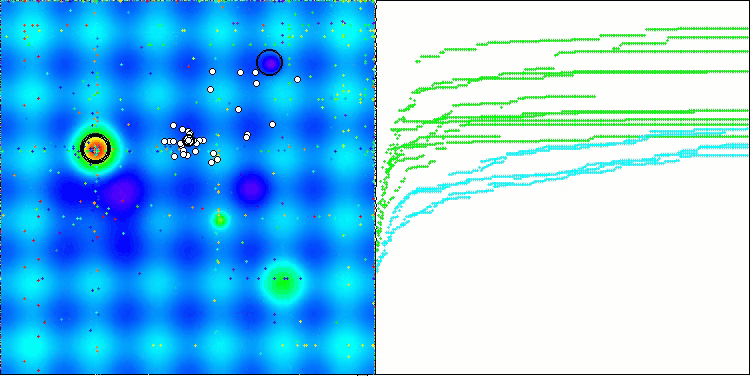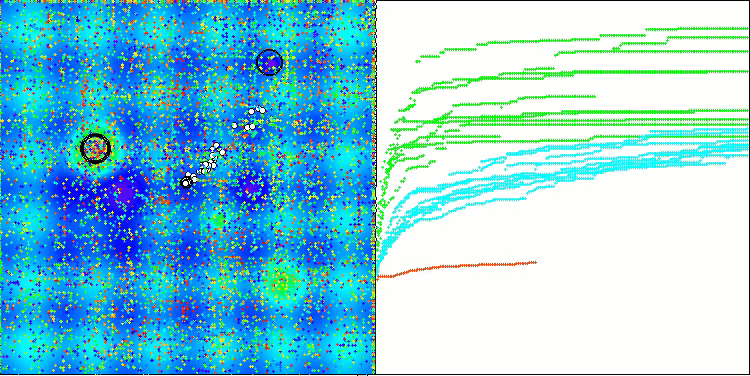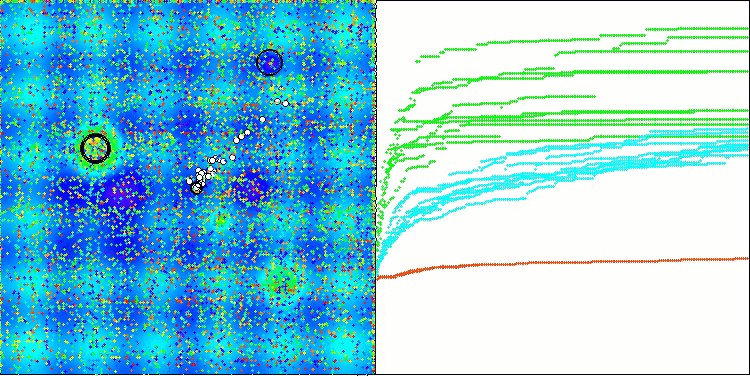
BCOm 在 Hilly 测试函数上的表现
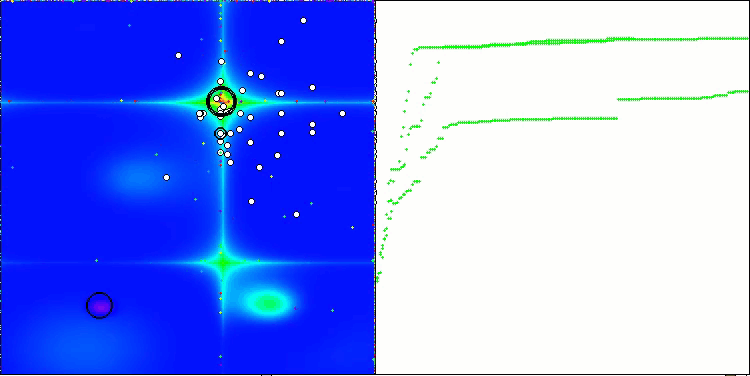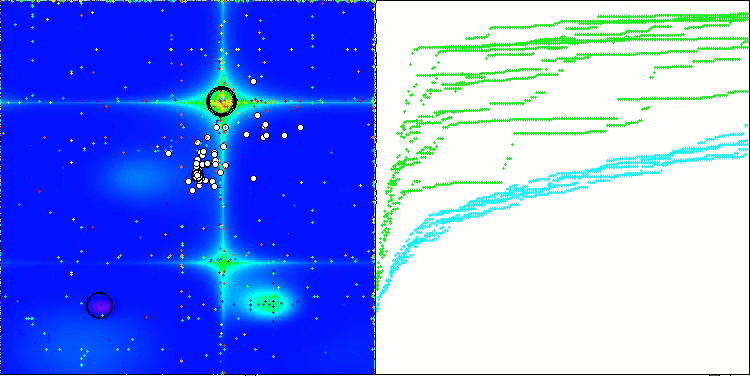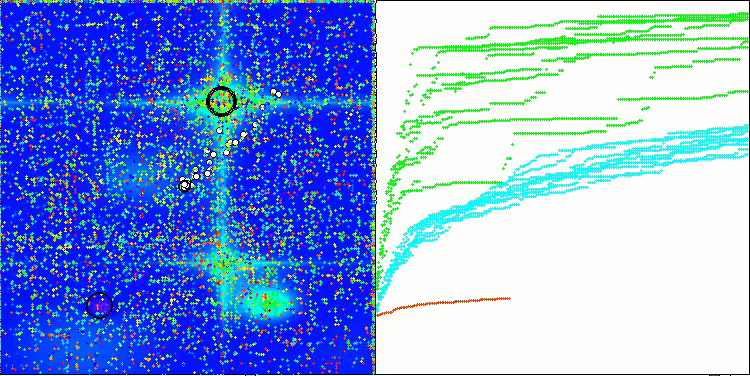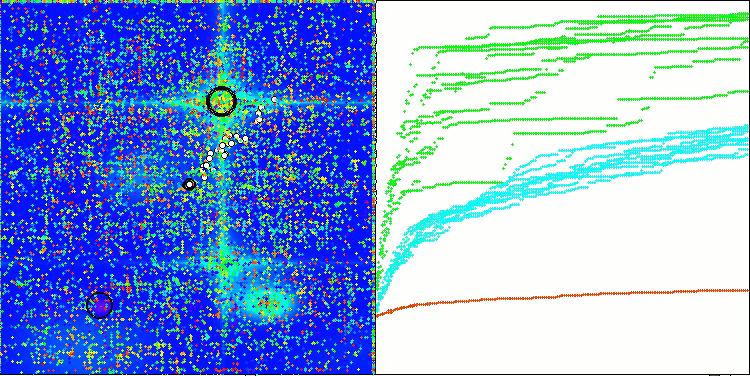
BCOm 在 Forest 测试函数上的表现
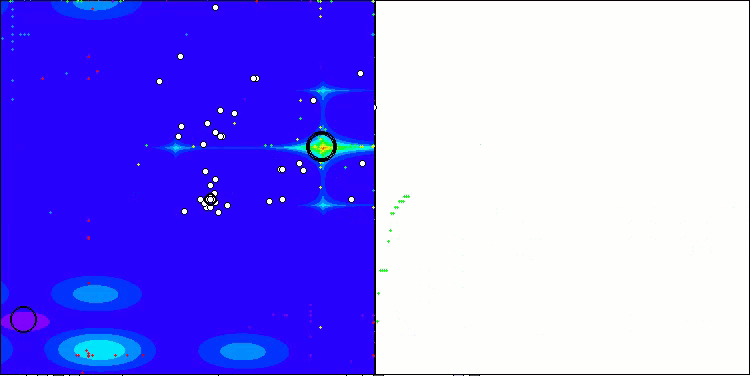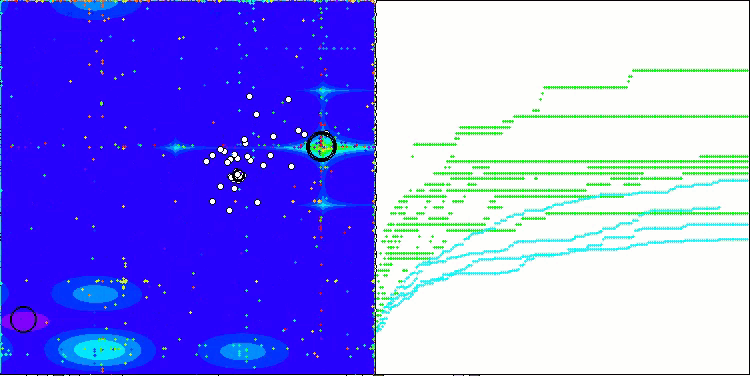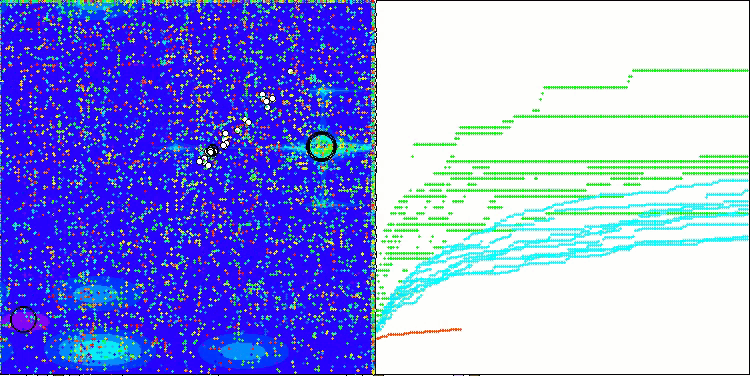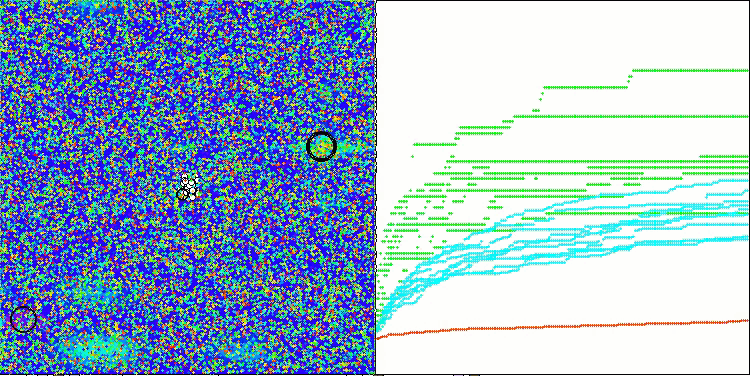
BCOm 在 Megacity 测试函数上的表现
根据测试结果,该算法在优化算法的总体排名中稳定地占据了第17位。
| # | 算法 | 说明 | Hilly | Hilly final | Forest | Forest final | Megacity (离散) | Megacity final | 最终结果 | 最大 % | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | ANS | 交叉邻近搜索 | 0.94948 | 0.84776 | 0.43857 | 2.23581 | 1.00000 | 0.92334 | 0.39988 | 2.32323 | 0.70923 | 0.63477 | 0.23091 | 1.57491 | 6.134 | 68.15 |
| 2 | CLA | 密码锁算法 | 0.95345 | 0.87107 | 0.37590 | 2.20042 | 0.98942 | 0.91709 | 0.31642 | 2.22294 | 0.79692 | 0.69385 | 0.19303 | 1.68380 | 6.107 | 67.86 |
| 3 | AMOm | 动物迁徙优化M | 0.90358 | 0.84317 | 0.46284 | 2.20959 | 0.99001 | 0.92436 | 0.46598 | 2.38034 | 0.56769 | 0.59132 | 0.23773 | 1.39675 | 5.987 | 66.52 |
| 4 | (P+O)ES | (P+O) 进化策略 | 0.92256 | 0.88101 | 0.40021 | 2.20379 | 0.97750 | 0.87490 | 0.31945 | 2.17185 | 0.67385 | 0.62985 | 0.18634 | 1.49003 | 5.866 | 65.17 |
| 5 | CTA | 彗星尾算法 | 0.95346 | 0.86319 | 0.27770 | 2.09435 | 0.99794 | 0.85740 | 0.33949 | 2.19484 | 0.88769 | 0.56431 | 0.10512 | 1.55712 | 5.846 | 64.96 |
| 6 | SDSm | 随机扩散搜索 M | 0.93066 | 0.85445 | 0.39476 | 2.17988 | 0.99983 | 0.89244 | 0.19619 | 2.08846 | 0.72333 | 0.61100 | 0.10670 | 1.44103 | 5.709 | 63.44 |
| 7 | ESG | 社会群体的演化 | 0.99906 | 0.79654 | 0.35056 | 2.14616 | 1.00000 | 0.82863 | 0.13102 | 1.95965 | 0.82333 | 0.55300 | 0.04725 | 1.42358 | 5.529 | 61.44 |
| 8 | SIA | 模拟各向同性退火 | 0.95784 | 0.84264 | 0.41465 | 2.21513 | 0.98239 | 0.79586 | 0.20507 | 1.98332 | 0.68667 | 0.49300 | 0.09053 | 1.27020 | 5.469 | 60.76 |
| 9 | ACS | 人工协同搜索 | 0.75547 | 0.74744 | 0.30407 | 1.80698 | 1.00000 | 0.88861 | 0.22413 | 2.11274 | 0.69077 | 0.48185 | 0.13322 | 1.30583 | 5.226 | 58.06 |
| 10 | ASO | 无政府社会优化 | 0.84872 | 0.74646 | 0.31465 | 1.90983 | 0.96148 | 0.79150 | 0.23803 | 1.99101 | 0.57077 | 0.54062 | 0.16614 | 1.27752 | 5.178 | 57.54 |
| 11 | TSEA | 龟壳演化算法 | 0.96798 | 0.64480 | 0.29672 | 1.90949 | 0.99449 | 0.61981 | 0.22708 | 1.84139 | 0.69077 | 0.42646 | 0.13598 | 1.25322 | 5.004 | 55.60 |
| 12 | DE | 差分进化 | 0.95044 | 0.61674 | 0.30308 | 1.87026 | 0.95317 | 0.78896 | 0.16652 | 1.90865 | 0.78667 | 0.36033 | 0.02953 | 1.17653 | 4.955 | 55.06 |
| 13 | CRO | 化学反应优化 | 0.94629 | 0.66112 | 0.29853 | 1.90593 | 0.87906 | 0.58422 | 0.21146 | 1.67473 | 0.75846 | 0.42646 | 0.12686 | 1.31178 | 4.892 | 54.36 |
| 14 | BSA | 鸟群算法 | 0.89306 | 0.64900 | 0.26250 | 1.80455 | 0.92420 | 0.71121 | 0.24939 | 1.88479 | 0.69385 | 0.32615 | 0.10012 | 1.12012 | 4.809 | 53.44 |
| 15 | HS | 和声搜索算法 | 0.86509 | 0.68782 | 0.32527 | 1.87818 | 0.99999 | 0.68002 | 0.09590 | 1.77592 | 0.62000 | 0.42267 | 0.05458 | 1.09725 | 4.751 | 52.79 |
| 16 | SSG | 树苗播种和生长算法 | 0.77839 | 0.64925 | 0.39543 | 1.82308 | 0.85973 | 0.62467 | 0.17429 | 1.65869 | 0.64667 | 0.44133 | 0.10598 | 1.19398 | 4.676 | 51.95 |
| 17 | BCOm | 细菌趋化优化算法M | 0.75953 | 0.62268 | 0.31483 | 1.69704 | 0.89378 | 0.61339 | 0.22542 | 1.73259 | 0.65385 | 0.42092 | 0.14435 | 1.21912 | 4.649 | 51.65 |
| 18 | (PO)ES | (PO) 进化策略 | 0.79025 | 0.62647 | 0.42935 | 1.84606 | 0.87616 | 0.60943 | 0.19591 | 1.68151 | 0.59000 | 0.37933 | 0.11322 | 1.08255 | 4.610 | 51.22 |
| 19 | TSm | 禁忌搜索优化算法 | 0.87795 | 0.61431 | 0.29104 | 1.78330 | 0.92885 | 0.51844 | 0.19054 | 1.63783 | 0.61077 | 0.38215 | 0.12157 | 1.11449 | 4.536 | 50.40 |
| 20 | BSO | 头脑风暴优化 | 0.93736 | 0.57616 | 0.29688 | 1.81041 | 0.93131 | 0.55866 | 0.23537 | 1.72534 | 0.55231 | 0.29077 | 0.11914 | 0.96222 | 4.498 | 49.98 |
| 21 | WOAm | 鲸鱼优化算法M | 0.84521 | 0.56298 | 0.26263 | 1.67081 | 0.93100 | 0.52278 | 0.16365 | 1.61743 | 0.66308 | 0.41138 | 0.11357 | 1.18803 | 4.476 | 49.74 |
| 22 | AEFA | 人工电场算法 | 0.87700 | 0.61753 | 0.25235 | 1.74688 | 0.92729 | 0.72698 | 0.18064 | 1.83490 | 0.66615 | 0.11631 | 0.09508 | 0.87754 | 4.459 | 49.55 |
| 23 | ACOm | 蚁群优化 M | 0.88190 | 0.66127 | 0.30377 | 1.84693 | 0.85873 | 0.58680 | 0.15051 | 1.59604 | 0.59667 | 0.37333 | 0.02472 | 0.99472 | 4.438 | 49.31 |
| 24 | BFO-GA | 细菌觅食优化 - ga | 0.89150 | 0.55111 | 0.31529 | 1.75790 | 0.96982 | 0.39612 | 0.06305 | 1.42899 | 0.72667 | 0.27500 | 0.03525 | 1.03692 | 4.224 | 46.93 |
| 25 | ABHA | 人工蜂巢算法 | 0.84131 | 0.54227 | 0.26304 | 1.64663 | 0.87858 | 0.47779 | 0.17181 | 1.52818 | 0.50923 | 0.33877 | 0.10397 | 0.95197 | 4.127 | 45.85 |
| 26 | ASBO | 适应性社会行为优化 | 0.76331 | 0.49253 | 0.32619 | 1.58202 | 0.79546 | 0.40035 | 0.26097 | 1.45677 | 0.26462 | 0.17169 | 0.18200 | 0.61831 | 3.657 | 40.63 |
| 27 | MEC | 思维进化计算 | 0.69533 | 0.53376 | 0.32661 | 1.55569 | 0.72464 | 0.33036 | 0.07198 | 1.12698 | 0.52500 | 0.22000 | 0.04198 | 0.78698 | 3.470 | 38.55 |
| 28 | IWO | 入侵性杂草优化 | 0.72679 | 0.52256 | 0.33123 | 1.58058 | 0.70756 | 0.33955 | 0.07484 | 1.12196 | 0.42333 | 0.23067 | 0.04617 | 0.70017 | 3.403 | 37.81 |
| 29 | Micro-AIS | 微型人工免疫系统 | 0.79547 | 0.51922 | 0.30861 | 1.62330 | 0.72956 | 0.36879 | 0.09398 | 1.19233 | 0.37667 | 0.15867 | 0.02802 | 0.56335 | 3.379 | 37.54 |
| 30 | COAm | 布谷鸟优化算法 M | 0.75820 | 0.48652 | 0.31369 | 1.55841 | 0.74054 | 0.28051 | 0.05599 | 1.07704 | 0.50500 | 0.17467 | 0.03380 | 0.71347 | 3.349 | 37.21 |
| 31 | SDOm | 螺旋动力学优化 M | 0.74601 | 0.44623 | 0.29687 | 1.48912 | 0.70204 | 0.34678 | 0.10944 | 1.15826 | 0.42833 | 0.16767 | 0.03663 | 0.63263 | 3.280 | 36.44 |
| 32 | NMm | Nelder-Mead 方法 M | 0.73807 | 0.50598 | 0.31342 | 1.55747 | 0.63674 | 0.28302 | 0.08221 | 1.00197 | 0.44667 | 0.18667 | 0.04028 | 0.67362 | 3.233 | 35.92 |
| 33 | FAm | 萤火虫算法 M | 0.58634 | 0.47228 | 0.32276 | 1.38138 | 0.68467 | 0.37439 | 0.10908 | 1.16814 | 0.28667 | 0.16467 | 0.04722 | 0.49855 | 3.048 | 33.87 |
| 34 | GSA | 重力搜索算法 | 0.64757 | 0.49197 | 0.30062 | 1.44016 | 0.53962 | 0.36353 | 0.09945 | 1.00260 | 0.32667 | 0.12200 | 0.01917 | 0.46783 | 2.911 | 32.34 |
| 35 | BFO | 细菌觅食优化 | 0.61171 | 0.43270 | 0.31318 | 1.35759 | 0.54410 | 0.21511 | 0.05676 | 0.81597 | 0.42167 | 0.13800 | 0.03195 | 0.59162 | 2.765 | 30.72 |
| 36 | ABC | 人工蜂群 | 0.63377 | 0.42402 | 0.30892 | 1.36671 | 0.55103 | 0.21874 | 0.05623 | 0.82600 | 0.34000 | 0.14200 | 0.03102 | 0.51302 | 2.706 | 30.06 |
| 37 | BA | 蝙蝠算法 | 0.59761 | 0.45911 | 0.35242 | 1.40915 | 0.40321 | 0.19313 | 0.07175 | 0.66810 | 0.21000 | 0.10100 | 0.03517 | 0.34617 | 2.423 | 26.93 |
| 38 | AAA | 藻类自适应算法 | 0.50007 | 0.32040 | 0.25525 | 1.07572 | 0.37021 | 0.22284 | 0.16785 | 0.76089 | 0.27846 | 0.14800 | 0.09755 | 0.52402 | 2.361 | 26.23 |
| 39 | SA | 模拟退火 | 0.55787 | 0.42177 | 0.31549 | 1.29513 | 0.34998 | 0.15259 | 0.05023 | 0.55280 | 0.31167 | 0.10033 | 0.02883 | 0.44083 | 2.289 | 25.43 |
| 40 | IWDm | 智能水滴 M | 0.54501 | 0.37897 | 0.30124 | 1.22522 | 0.46104 | 0.14704 | 0.04369 | 0.65177 | 0.25833 | 0.09700 | 0.02308 | 0.37842 | 2.255 | 25.06 |
| 41 | PSO | 粒子群优化 | 0.59726 | 0.36923 | 0.29928 | 1.26577 | 0.37237 | 0.16324 | 0.07010 | 0.60572 | 0.25667 | 0.08000 | 0.02157 | 0.35823 | 2.230 | 24.77 |
| 42 | Boids | boids算法 | 0.43340 | 0.30581 | 0.25425 | 0.99346 | 0.35718 | 0.20160 | 0.15708 | 0.71586 | 0.27846 | 0.14277 | 0.09834 | 0.51957 | 2.229 | 24.77 |
| 43 | MA | 猴群算法 | 0.59107 | 0.42681 | 0.31816 | 1.33604 | 0.31138 | 0.14069 | 0.06612 | 0.51819 | 0.22833 | 0.08567 | 0.02790 | 0.34190 | 2.196 | 24.40 |
| 44 | SFL | 混合蛙跳算法 | 0.53925 | 0.35816 | 0.29809 | 1.19551 | 0.37141 | 0.11427 | 0.04051 | 0.52618 | 0.27167 | 0.08667 | 0.02402 | 0.38235 | 2.104 | 23.38 |
| 45 | FSS | 鱼群搜索 | 0.55669 | 0.39992 | 0.31172 | 1.26833 | 0.31009 | 0.11889 | 0.04569 | 0.47467 | 0.21167 | 0.07633 | 0.02488 | 0.31288 | 2.056 | 22.84 |
总结
我们已经探讨了 BCO 算法的原始版本和改进版本。从开源资源重建的原始版本结果表明,它充斥着重计算量的三角函数运算,并且搜索性能较弱,还存在一些数学上的“错误”。因此,有必要重新思考整个算法,并对搜索策略进行深入分析,从而创建一个新的改进版本。算法逻辑的更改使得每个优化步骤的计算更加简单,这对代码执行速度产生了积极影响。新算法在探索新的解空间和利用已找到的良好解之间取得了不同的平衡。
尽管新方法与细菌的自然行为有些相悖,但它在实际应用中被证明是非常高效的。算法运行的可视化结果显示了其深入探索超空间重要区域的能力,这也表明了其改进的研究能力。因此,与原始版本相比,新版本的算法被证明更强大且更高效。
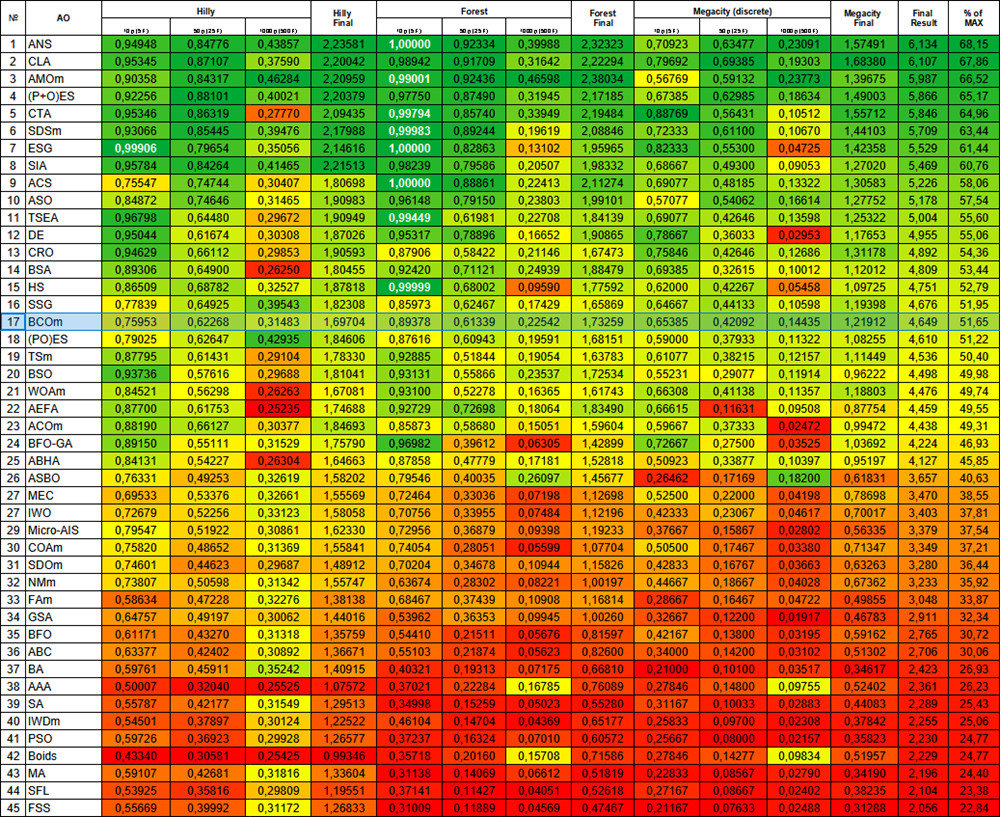
图例 1. 根据相关测试结果对算法进行颜色分级,结果大于或等于 0.99 的部分 以白色突出显示。
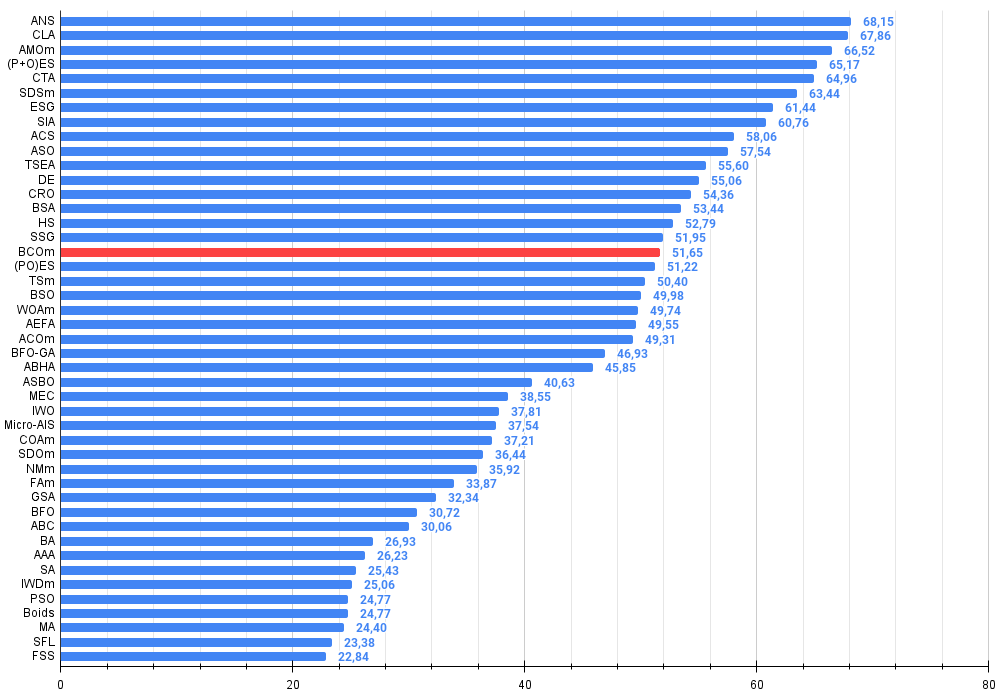
图例 2. 算法测试结果的直方图(标尺从 0 到 100,越多越好,
其中100是理论上的最大可能结果,附件提供了一个计算评级表格的脚本)
BCOm的优缺点:
优点:
- 速度快。
- 自适应。
- 良好的扩展性。
- 只有一个外部参数。
缺点:
- 在低维函数上结果分布较为分散。
本文附带了一个文档,其中包含了算法代码的当前版本。文章作者不对典型算法描述的绝对准确性负责。对其中进行了多处修改,从而提升搜索能力。文章中表述的结论和论断是基于实验的结果。
本文由MetaQuotes Ltd译自俄文
原文地址: https://www.mql5.com/ru/articles/15711
 使用MQL5和Python构建自优化EA(第三部分):破解Boom 1000算法
使用MQL5和Python构建自优化EA(第三部分):破解Boom 1000算法
 从基础到中级:操作符
从基础到中级:操作符
 交易中的神经网络:将全局信息注入独立通道(InjectTST)
交易中的神经网络:将全局信息注入独立通道(InjectTST)
 在 MQL5 中重新构想经典策略(第二部分):富时 100 指数(FTSE100)与英国国债(UK Gilts)
在 MQL5 中重新构想经典策略(第二部分):富时 100 指数(FTSE100)与英国国债(UK Gilts)