动物迁徙优化(AMO)算法
概述
动物迁徙:自然的和谐与生存策略。动物通常在越冬地和繁殖地之间迁徙,沿着经过数百年进化形成的固定路线。这些季节性旅程并非随机游荡,而是精心规划的路线,通向最有利于它们生存和繁殖的区域。根据季节,动物们为了寻找食物、庇护所和适合繁殖的条件而移动,这些行为受到它们群体的自然需求和本能的引导。每一次这样的迁徙不仅是对生存的斗争,也是与自然和谐相处的体现,其中每一个个体都在整个生态系统中扮演着独特的角色。
例如,驯鹿会迁徙很长的距离以寻找更好的栖息地,而鸟类(如鹤和雁)则会在越冬地和繁殖地之间进行长途飞行,它们使用代代相传的特定路线。这些迁徙不仅确保了各个物种的生存,还支持了整个生态系统,促进了植物的授粉和种子的传播。
从自然中汲取灵感。 AMO(动物迁徙优化)算法于2013年由研究者李先涛提出。该算法的主要思想是模拟动物在自然中寻找生存和繁殖最优条件的季节性迁徙过程。算法的灵感来源于对鸟类、鱼类和哺乳动物等迁徙动物行为的观察。这些动物在越冬地和繁殖地之间进行季节性移动,遵循自然在迁徙过程中发展出的特定互动规则。
AMO算法模拟了动物长距离移动的三个主要组成部分:避免与邻近个体发生碰撞、与群体(群体)保持相同方向移动以及保持彼此之间的适当距离。这些原则不仅有助于避免冲突,还维持了集体行为,这对于在野外生存至关重要。
AMO算法中的优化阶段。该算法在一个迭代中包含两个关键的优化阶段:
- 迁徙:在这个阶段,个体的位置会根据其邻居进行更新。
- 种群更新:在这个阶段,个体部分地被新的个体替换,替换的概率取决于个体在群体中的位置。
模拟迁徙动物的集体行为可以成为解决复杂优化问题的有效方法。该算法尝试对搜索空间的平衡探索和对已找到的最优解决方案的利用,模仿自然过程。
2. 算法实现
在动物迁徙的算法模型中,基本概念是在每个动物周围创建同心圆的“区域”。在排斥区域,动物倾向于远离其邻居以避免碰撞。根据作者的观点,动物迁徙算法分为两个主要过程:
1. 动物迁徙。使用拓扑环来描述个体的有限邻域。为了方便起见,每个维度的邻域长度都被设置为5。邻域拓扑保持静止,并根据个体在群体中的索引确定。如果动物的索引是“j”,那么它的邻居的索引为 [j - 2, j - 1, j, j + 1, j + 2]。如果动物的索引是“1”,其邻居的索引将是 [N - 1, N, 1, 2, 3],依此类推。一旦形成邻域拓扑,随机选择一个邻居,并更新个体的位置以反映该邻居的位置。这是原始算法作者给出的描述。在这种情况下,邻居的数量限制可以作为算法的参数输入,以便通过实验找到最佳的邻居数量,从而确保算法的最高效率。
2. 种群更新在种群更新过程中,算法模拟一些动物离开群体,而另一些动物加入群体。个体可以以概率“p”被新动物替换,该概率基于适应度函数的质量确定。我们根据适应度函数值对种群进行降序排序,这意味着具有最佳适应度的个体被替换的概率为1/N,而适应度最差的个体被替换的概率为1。
根据作者的版本,动物迁徙和种群更新可以用算法描述如下。
动物迁徙:
1. 对于每只动物: a. 确定动物的拓扑邻域(5个最近的邻居)。b. 从邻居列表中随机选择一个邻居。
c. 使用以下公式更新动物朝向选定邻居的方向上的位置:
x_j_new = x_j_old + r * (x_neighbor - x_j_old),其中:
- x_j_new — 第j只动物的新位置,
- x_j_old — 当前位置,
- x_neighbor — 选定邻居的位置,
- r — 从正态分布中抽取的随机数。
d. 使用目标函数评估动物的新位置。
种群更新:
1. 按目标函数值对动物进行降序排序。2. 对于每只动物: a. 计算用新随机动物替换原动物的概率:p = 1.0 - (1.0 / (x + 1)),其中x是排序列表中第i只动物的排名(索引)。
b. 如果随机数小于p,则替换该动物(将其坐标更改为从种群中随机选择的两只动物坐标的平均值)。 c. 否则,保持动物不变。3. 使用目标函数评估新种群。
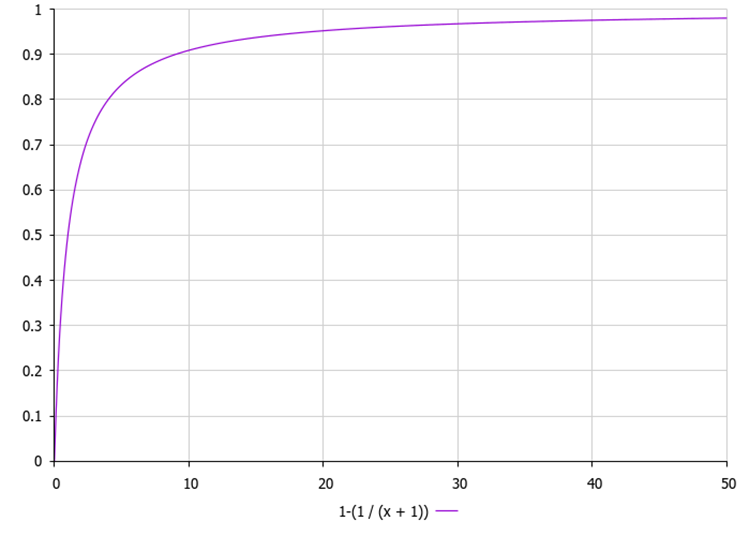
图例1. 根据个体在群体中的位置改变其概率,其中“x”是个体在群体中的索引。
让我们为AMO算法编写伪代码。
1. 初始化动物群体的随机位置。
2. 主循环:
a. 对于每只动物:
定义拓扑邻域。随机选择一个邻居。
朝向该邻居的方向更新动物的位置。
评估新位置。
b. 根据目标函数值对群体进行排序。
c. 以概率方式用新的个体替换表现较差的个体。
d. 评估更新后的群体。
e. 更新最优解。
3. 从步骤2开始重复,直到满足停止条件。
现在我们已经熟悉了该算法,可以继续编写代码了。让我们更仔细地看一下C_AO_AMO类的代码:
1. C_AO_AMO类继承自C_AO基类,这使得我们可以使用其功能并对其进行扩展。
2. 构造函数指定了算法的基本特征,例如名称、描述以及文章链接。算法参数也被初始化,包括种群大小、偏差和邻居数量。
3. popSize、deviation、neighborsNumberOnSide — 这些变量决定了种群大小、标准差以及在迁徙过程中考虑的一侧邻居数量。
4. SetParams — 该方法负责根据存储在params数组中的值更新算法参数。
5. Init — 初始化方法,接受最小值和最大值范围数组、步长以及迭代次数。
6. Moving () — 负责在搜索空间中移动智能体的方法,Revision () — 检查并更新种群中智能体状态的方法。
7. S_AO_Agent population []— 该数组存储当前种群中的智能体(动物),S_AO_Agent pTemp []— 在对种群进行排序时使用的临时数组。
8. GetNeighborsIndex — 用于获取特定智能体邻居索引的私有方法。
C_AO_AMO类实现了基于动物迁徙的优化算法,提供了设置和执行算法所需的必要方法和参数。它继承了其基类的功能,这使得我们可以根据任务需求扩展和修改其行为。
//—————————————————————————————————————————————————————————————————————————————— class C_AO_AMOm : public C_AO { public: //-------------------------------------------------------------------- ~C_AO_AMOm () { } C_AO_AMOm () { ao_name = "AMOm"; ao_desc = "Animal Migration Optimization M"; ao_link = "https://www.mql5.com/en/articles/15543"; popSize = 50; // Population size deviation = 8; neighborsNumberOnSide = 10; ArrayResize (params, 3); params [0].name = "popSize"; params [0].val = popSize; params [1].name = "deviation"; params [1].val = deviation; params [2].name = "neighborsNumberOnSide"; params [2].val = neighborsNumberOnSide; } void SetParams () { popSize = (int)params [0].val; deviation = params [1].val; neighborsNumberOnSide = (int)params [2].val; } bool Init (const double &rangeMinP [], const double &rangeMaxP [], const double &rangeStepP [], const int epochsP = 0); void Moving (); void Revision (); //---------------------------------------------------------------------------- double deviation; int neighborsNumberOnSide; S_AO_Agent population []; // Animal population S_AO_Agent pTemp []; // Temporary animal population private: //------------------------------------------------------------------- int GetNeighborsIndex (int i); }; //——————————————————————————————————————————————————————————————————————————————
接下来,让我们看看C_AO_AMO类中Init方法的代码。该方法的每一部分描述如下:
1. rangeMinP []、rangeMaxP []、 rangeStepP [] — 用于确定优化参数的最小值和最大值范围及其步长的数组。
2. StandardInit方法根据传递的范围执行标准初始化。
3. 根据popSize调整population和pTemp数组的大小。
4. 基于population数组的所有元素,并且使用Init方法通过传递coords坐标数量来初始化每个智能体。
5. 如果所有操作都成功,该方法返回true。
C_AO_AMO类中的Init方法负责根据给定的范围和参数初始化智能体群体。
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_AMO::Init (const double &rangeMinP [], const double &rangeMaxP [], const double &rangeStepP [], const int epochsP = 0) { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (population, popSize); ArrayResize (pTemp, popSize); for (int i = 0; i < popSize; i++) population [i].Init (coords); return true; } //——————————————————————————————————————————————————————————————————————————————
接下来,我们将查看C_AO_AMO类中的 Moving 方法,该方法负责群体中智能体的移动。代码的主要部分如下:
1. 检查revision状态:
- 如果revision等于false,则表示方法是首次调用或在重置后调用。在这种情况下,会将种群初始化。
- 对于种群中的每个个体(从0到popSize)以及每个坐标(从0到coords),在指定范围内(rangeMin和rangeMax)生成随机值。
- 这些值随后通过SeInDiSp函数进行处理,该函数会根据指定的步长(rangeStep)进行调整。
2. 设置revision标识:
- 初始化完成后,将revision设置为true,方法随即终止。
3. 基本迁徙循环:
- 如果revision等于true,方法将切换到主要的迁徙逻辑。
- 对于每个个体,再次对坐标进行迭代。
- 使用GetNeighborsIndex(i)获取当前个体将要比较的邻居的索引。
- 计算邻居坐标与当前个体坐标之间的dist距离。
- 根据该距离,确定新坐标所在的最小值和最大值边界(min和max)。
- 如果计算出的边界超出了可接受范围,将对其进行调整,以考虑rangeMin和 rangeMax。
- 随后,使用正态分布(GaussDistribution函数)计算新的坐标值,这允许考虑标准差(deviation)。
- 与第一种情况一样,新值也通过SeInDiSp进行处理。
Moving方法负责根据智能体的邻居和随机因素更新它们的位置。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_AMO::Moving () { //---------------------------------------------------------------------------- if (!revision) { for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { a [i].c [c] = u.RNDfromCI (rangeMin [c], rangeMax [c]); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } revision = true; return; } //---------------------------------------------------------------------------- int ind1 = 0; double dist = 0.0; double x = 0.0; double min = 0.0; double max = 0.0; for (int i = 0; i < popSize; i++) { for (int c = 0; c < coords; c++) { //------------------------------------------------------------------------ ind1 = GetNeighborsIndex (i); dist = fabs (population [ind1].c [c] - a [i].c [c]); x = a [i].c [c]; min = x - dist; max = x + dist; if (min < rangeMin [c]) min = rangeMin [c]; if (max > rangeMax [c]) max = rangeMax [c]; a [i].c [c] = u.GaussDistribution (x, min, max, deviation); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } } //——————————————————————————————————————————————————————————————————————————————
以下是C_AO_AMO类中的Revision方法代码。让我们逐步查看:
1. 寻找最佳个体:
- 使用变量ind存储具有最佳函数值(f)的个体索引。
- 遍历整个种群(从 0到popSize),如果当前个体的函数值最佳,则更新fB的值。
- 如果找到最佳个体,将其特征(坐标)复制到数组cB中。
- 对于种群中的每个个体(从0到popSize),计算概率prob ,该概率取决于索引i。
- 对于每个坐标(从0到coords),随机与概率prob进行比较。
- 如果随机数小于prob,则随机选择两个个体ind1和ind2。
- 如果两个个体相同,则增加ind2以避免选择同一个个体。
- 当前个体的新坐标值计算为两个随机个体坐标的平均值,通过SeInDiSp函数进行调整,该函数将值限制在给定范围内。
- 完成所有更改后,通过从数组a复制值来更新整个种群。
- 接下来调用Sorting函数。根据函数f对种群排序。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_AMO::Revision () { //---------------------------------------------------------------------------- int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); //---------------------------------------------------------------------------- int ind1 = 0; int ind2 = 0; double dist = 0.0; double x = 0.0; double min = 0.0; double max = 0.0; double prob = 0.0; for (int i = 0; i < popSize; i++) { prob = 1.0 - (1.0 / (i + 1)); for (int c = 0; c < coords; c++) { if (u.RNDprobab() < prob) { ind1 = u.RNDminusOne (popSize); ind2 = u.RNDminusOne (popSize); if (ind1 == ind2) { ind2++; if (ind2 > popSize - 1) ind2 = 0; } a [i].c [c] = (population [ind1].c [c] + population [ind2].c [c]) * 0.5; a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } } //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) population [i] = a [i]; u.Sorting (population, pTemp, popSize); } //——————————————————————————————————————————————————————————————————————————————
最后,我们来看C_AO_AMO类中GetNeighborsIndex方法的代码,该方法负责为指定的索引i获取随机邻居的索引,同时考虑数组边界。
1. 变量初始化:
- Ncount — 由neighborsNumberOnSide 变量确定的邻居数量。
- N — 包括元素本身在内的总邻居数量,定义为 Ncount * 2 + 1。
2. 该方法使用条件语句来检查索引i相对于数组边界的位置。
3. 处理前面Ncount个元素(左侧边界)。如果i索引小于Ncount,意味着其位于数组的开头。在这种情况下,该方法是从0 到N-1生成一个随机邻居索引。
4. 处理后面Ncount个元素(右侧边界):如果i索引大于或等于popSize - Ncount,这意味着其位于数组的末尾。该方法从popSize - N开始生成邻居索引,以考虑边界。
5. 处理所有其他元素:如果i索引位于数组的中间部分,方法会生成一个向左偏移Ncount的邻居索引,并加上一个从0到N-1的随机值。
6. 最后,该方法返回生成的邻居索引。
GetNeighborsIndex方法允许为给定的索引i获取随机邻居的索引,同时考虑数组边界。
//—————————————————————————————————————————————————————————————————————————————— int C_AO_AMO::GetNeighborsIndex (int i) { int Ncount = neighborsNumberOnSide; int N = Ncount * 2 + 1; int neighborIndex; // Select a random neighbor given the array boundaries if (i < Ncount) { // For the first Ncount elements neighborIndex = MathRand () % N; } else { if (i >= popSize - Ncount) { // For the last Ncount elements neighborIndex = (popSize - N) + MathRand () % N; } else { // For all other elements neighborIndex = i - Ncount + MathRand () % N; } } return neighborIndex; } //——————————————————————————————————————————————————————————————————————————————
现在,既然我们已经完成了算法的编写,让我们检查一下它是如何工作的。算法原始版本的结果:
AMO|Animal Migration Optimization|50.0|1.0|2.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.43676335174918435
25 Hilly's; Func runs: 10000; result: 0.28370099295372453
500 Hilly's; Func runs: 10000; result: 0.25169663266864406
=============================
5 Forest's; Func runs: 10000; result: 0.312993148861033
25 Forest's; Func runs: 10000; result: 0.23960515885149344
500 Forest's; Func runs: 10000; result: 0.18938496103401775
=============================
5 Megacity's; Func runs: 10000; result: 0.18461538461538463
25 Megacity's; Func runs: 10000; result: 0.12246153846153851
500 Megacity's; Func runs: 10000; result: 0.10223076923076994
=============================
总分:2.12345 (23.59%)
遗憾的是,原始版本的搜索质量较差。这样的指标将不会被纳入评分表。对结果的分析揭示了该算法与其他同类算法之间存在显著差距,这促使我重新对其进行更深入地思考。
经过仔细检查策略,发现了一个关键缺陷:种群排序并没有促进最优个体的遗传物质积累。只是改变了它们的拓扑排列,而没有影响它们的本质。排序的影响仅限于调整个体在搜索空间中坐标变化的概率,而这一概率与它们的适应度成反比。
值得注意的是,新的坐标完全是在种群中已经存在的基础上形成的,通过对两个随机选择的个体值进行平均。对这些细节的认识促使了扩大种群以在排序之前将后代整合到父群体中的想法。这种方法不仅会保留最佳的遗传组合,还会自然地淘汰适应性较差的个体。毫无疑问,更新种群基因库的问题仍然很重要,但所提出的修改应该会显著提高进化过程的动态性。为了实现这一想法,我们首先通过将父代种群的规模加倍来改变初始化方法。
让我们完整地呈现初始化代码,我们可以看到父代种群的加倍。
//—————————————————————————————————————————————————————————————————————————————— bool C_AO_AMOm::Init (const double &rangeMinP [], const double &rangeMaxP [], const double &rangeStepP [], const int epochsP = 0) { if (!StandardInit (rangeMinP, rangeMaxP, rangeStepP)) return false; //---------------------------------------------------------------------------- ArrayResize (population, popSize * 2); ArrayResize (pTemp, popSize * 2); for (int i = 0; i < popSize * 2; i++) population [i].Init (coords); return true; } //——————————————————————————————————————————————————————————————————————————————
相应地,有必要对Revision方法进行修正。
//---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) population [i + popSize] = a [i]; u.Sorting (population, pTemp, popSize * 2);
在进行相应的调整后,我们将再次测试该算法并对比结果:
AMOm|Animal Migration Optimization M|50.0|1.0|2.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.4759595972704031
25 Hilly's; Func runs: 10000; result: 0.31711543296080447
500 Hilly's; Func runs: 10000; result: 0.2540492181444619
=============================
5 Forest's; Func runs: 10000; result: 0.40387880560608347
25 Forest's; Func runs: 10000; result: 0.27049305409901064
500 Forest's; Func runs: 10000; result: 0.19135802944407254
=============================
5 Megacity's; Func runs: 10000; result: 0.23692307692307696
25 Megacity's; Func runs: 10000; result: 0.14461538461538465
500 Megacity's; Func runs: 10000; result: 0.10109230769230851
=============================
总分:2.39548 (26.62%)
在这种情况下,我们看到总体结果提高了3%,这表明在选择的路径上取得成功的可能性。
接下来,我们将尝试将基于排名的个体概率性变化传递到Moving方法中。允许在从最近邻域获得新坐标后,立即对个体的坐标进行更改。
//---------------------------------------------------------------------------- int ind1 = 0; int ind2 = 0; double dist = 0.0; double x = 0.0; double min = 0.0; double max = 0.0; double prob = 0.0; for (int i = 0; i < popSize; i++) { prob = 1.0 - (1.0 / (i + 1)); for (int c = 0; c < coords; c++) { //------------------------------------------------------------------------ ind1 = GetNeighborsIndex (i); dist = fabs (population [ind1].c [c] - a [i].c [c]); x = a [i].c [c]; min = x - dist; max = x + dist; if (min < rangeMin [c]) min = rangeMin [c]; if (max > rangeMax [c]) max = rangeMax [c]; a [i].c [c] = u.GaussDistribution (x, min, max, deviation); //---------------------------------------------------------------------------- if (u.RNDprobab() < prob) { ind1 = u.RNDminusOne (popSize); ind2 = u.RNDminusOne (popSize); if (ind1 == ind2) { ind2++; if (ind2 > popSize - 1) ind2 = 0; } a [i].c [c] = (population [ind1].c [c] + population [ind2].c [c]) * 0.5; } a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } }
让我们再次检查结果:
AMO|Animal Migration Optimization|50.0|1.0|2.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.7204154413083147
25 Hilly's; Func runs: 10000; result: 0.4480389094268583
500 Hilly's; Func runs: 10000; result: 0.25286213277651365
=============================
5 Forest's; Func runs: 10000; result: 0.7097109421461968
25 Forest's; Func runs: 10000; result: 0.3299544372347476
500 Forest's; Func runs: 10000; result: 0.18667655927410348
=============================
5 Megacity's; Func runs: 10000; result: 0.41076923076923083
25 Megacity's; Func runs: 10000; result: 0.20400000000000001
500 Megacity's; Func runs: 10000; result: 0.09586153846153929
=============================
总分:3.35829 (37.31%)
这次要好得多,值得继续下去。经过对代码的若干次实验,我们得到了Moving方法的最终版本:
//---------------------------------------------------------------------------- int ind1 = 0; int ind2 = 0; double dist = 0.0; double x = 0.0; double min = 0.0; double max = 0.0; double prob = 0.0; for (int i = 0; i < popSize; i++) { prob = 1.0 - (1.0 / (i + 1)); for (int c = 0; c < coords; c++) { //------------------------------------------------------------------------ ind1 = GetNeighborsIndex (i); dist = fabs (population [ind1].c [c] - a [i].c [c]); x = population [ind1].c [c]; min = x - dist; max = x + dist; if (min < rangeMin [c]) min = rangeMin [c]; if (max > rangeMax [c]) max = rangeMax [c]; a [i].c [c] = u.GaussDistribution (x, min, max, deviation); a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); //------------------------------------------------------------------------ if (u.RNDprobab() < prob) { if (u.RNDprobab() <= 0.01) { ind1 = u.RNDminusOne (popSize); ind2 = u.RNDminusOne (popSize); //if (ind1 == ind2) { //ind2++; //if (ind2 > popSize - 1) ind2 = 0; a [i].c [c] = (population [ind1].c [c] + population [ind2].c [c]) * 0.5; a [i].c [c] = u.SeInDiSp (a [i].c [c], rangeMin [c], rangeMax [c], rangeStep [c]); } } //ind1 = u.RNDminusOne (popSize); //a [i].c [c] = population [ind1].c [c]; } } } } //——————————————————————————————————————————————————————————————————————————————
让我们从Moving方法转向C_AO_AMO中负责更新和排序智能体种群的Revision方法的最终版本。
//—————————————————————————————————————————————————————————————————————————————— void C_AO_AMO::Revision () { //---------------------------------------------------------------------------- int ind = -1; for (int i = 0; i < popSize; i++) { if (a [i].f > fB) { fB = a [i].f; ind = i; } } if (ind != -1) ArrayCopy (cB, a [ind].c, 0, 0, WHOLE_ARRAY); //---------------------------------------------------------------------------- for (int i = 0; i < popSize; i++) population [popSize + i] = a [i]; u.Sorting (population, pTemp, popSize * 2); } //——————————————————————————————————————————————————————————————————————————————一旦代码最终形成,我们便再次进行测试。
3. 测试结果
AMO试验台结果:
AMOm|Animal Migration Optimization|50.0|8.0|10.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.9627642143272663
25 Hilly's; Func runs: 10000; result: 0.8703754433240446
500 Hilly's; Func runs: 10000; result: 0.467183248460726
=============================
5 Forest's; Func runs: 10000; result: 0.9681183408862706
25 Forest's; Func runs: 10000; result: 0.9109372988714968
500 Forest's; Func runs: 10000; result: 0.4719026790932256
=============================
5 Megacity's; Func runs: 10000; result: 0.6676923076923076
25 Megacity's; Func runs: 10000; result: 0.5886153846153845
500 Megacity's; Func runs: 10000; result: 0.23546153846153978
=============================
总分: 6.14305 (68.26%)
我们在评分表中看到了高分的结果。然而,代价是在小维度函数上最终值的分布范围较广。让我们进行50次测试,而不是通常的10次。
AMOm|Animal Migration Optimization|50.0|8.0|10.0|
=============================
5 Hilly's; Func runs: 10000; result: 0.903577388020872
25 Hilly's; Func runs: 10000; result: 0.8431723262743616
500 Hilly's; Func runs: 10000; result: 0.46284266807030283
=============================
5 Forest's; Func runs: 10000; result: 0.9900061169785055
25 Forest's; Func runs: 10000; result: 0.9243600311397848
500 Forest's; Func runs: 10000; result: 0.4659761237381695
=============================
5 Megacity's; Func runs: 10000; result: 0.5676923076923077
25 Megacity's; Func runs: 10000; result: 0.5913230769230771
500 Megacity's; Func runs: 10000; result: 0.23773230769230896
=============================
总分:5.98668 (66.52%)
现在结果更加接近真实情况,但效率也略有下降。
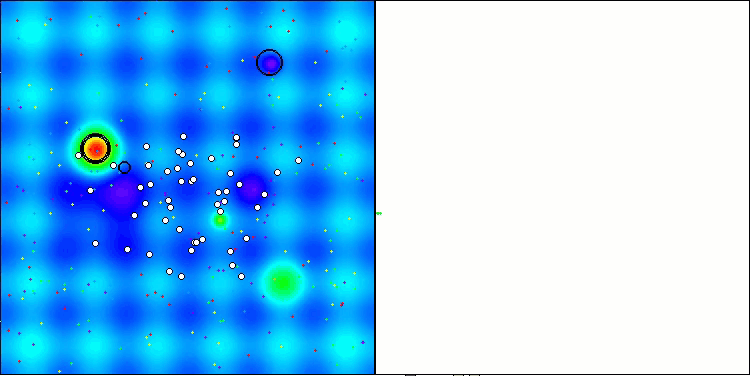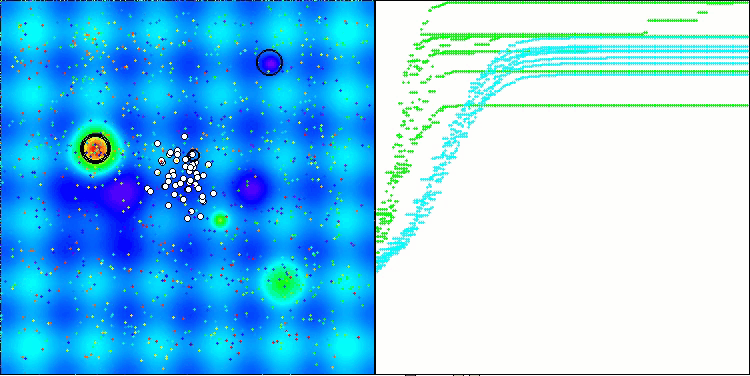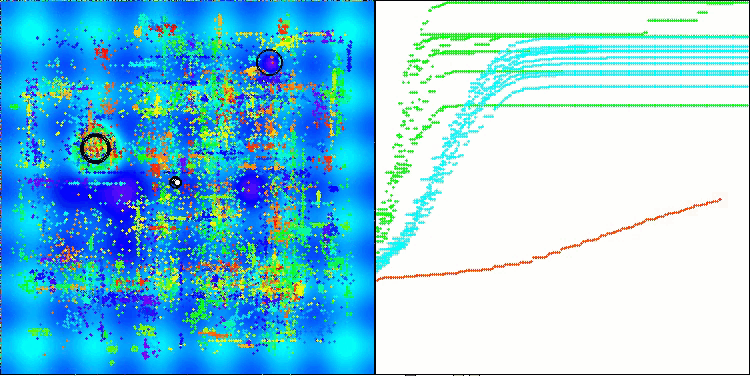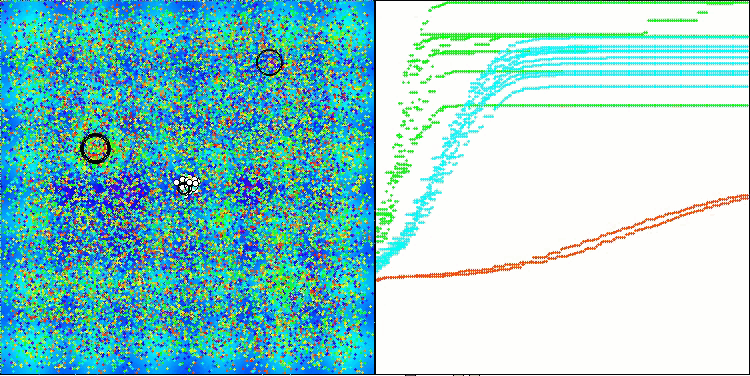
AMOm在 Hilly 测试函数上
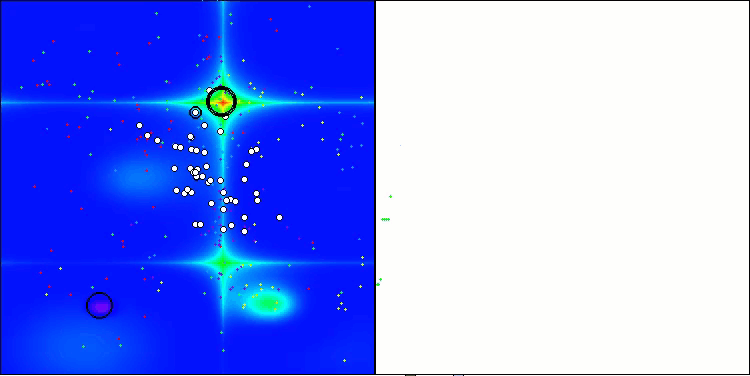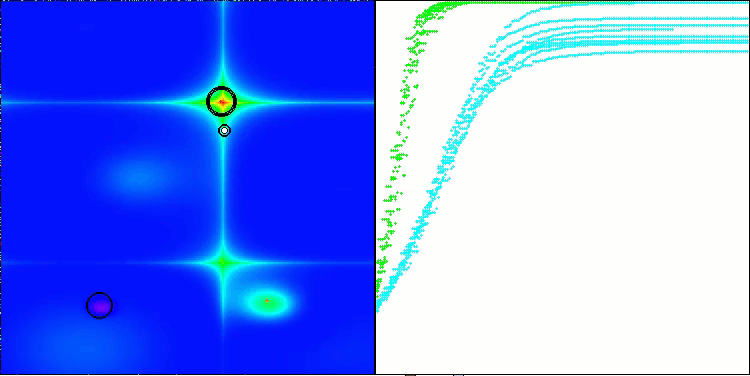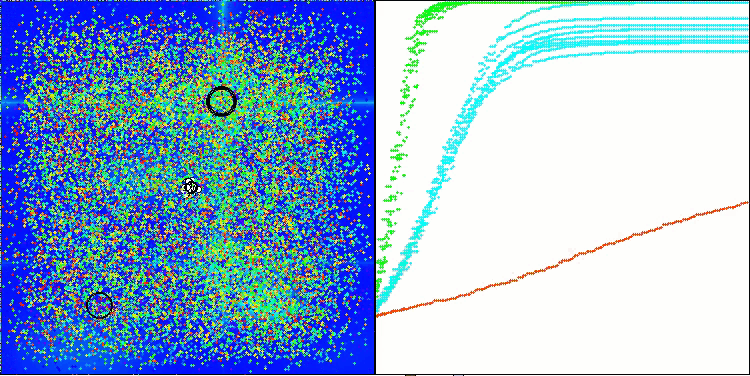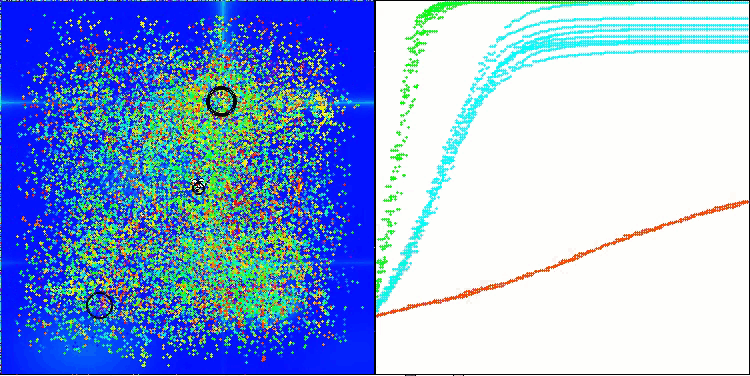
AMOm在 Forest 测试函数上
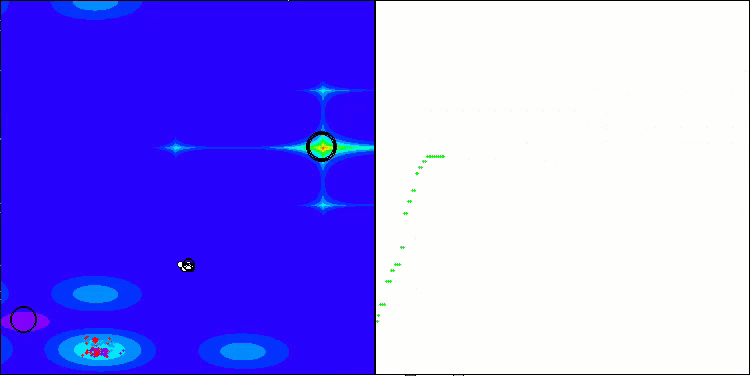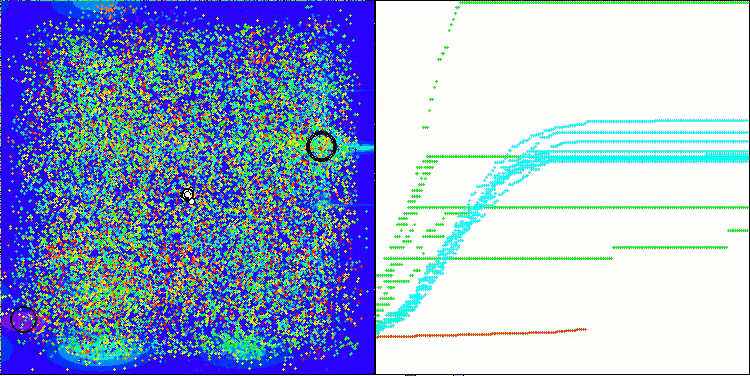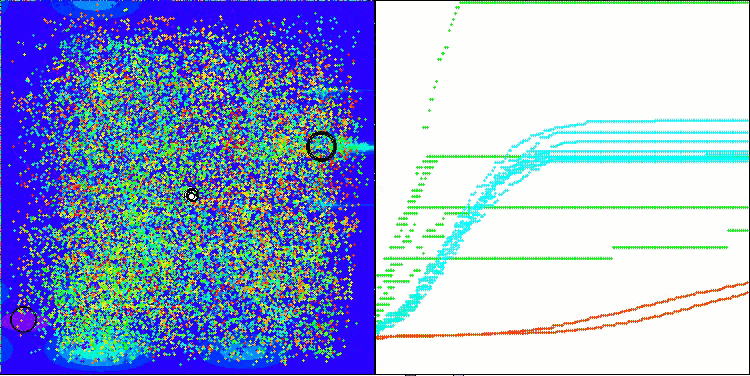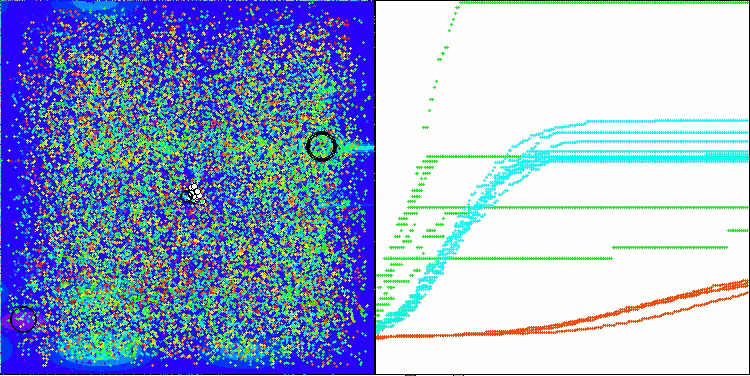
AMOm在 Megacity 测试函数上
经过了一些惊人的改进后,该算法在评分表中稳稳地占据了第三名的位置。
| # | AO | 说明 | Hilly值 | Hilly最终值 | Forest值 | Forest最终值 | Megacity (离散) | Megacity最终值 | 最终结果 | 最大百分比 | ||||||
| 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | 10 p (5 F) | 50 p (25 F) | 1000 p (500 F) | ||||||||
| 1 | ANS | 跨邻域搜索 | 0.94948 | 0.84776 | 0.43857 | 2.23581 | 1.00000 | 0.92334 | 0.39988 | 2.32323 | 0.70923 | 0.63477 | 0.23091 | 1.57491 | 6.134 | 68.15 |
| 2 | CLA | 密码锁算法 | 0.95345 | 0.87107 | 0.37590 | 2.20042 | 0.98942 | 0.91709 | 0.31642 | 2.22294 | 0.79692 | 0.69385 | 0.19303 | 1.68380 | 6.107 | 67.86 |
| 3 | AMOm | 动物迁徙优化M | 0.90358 | 0.84317 | 0.46284 | 2.20959 | 0.99001 | 0.92436 | 0.46598 | 2.38034 | 0.56769 | 0.59132 | 0.23773 | 1.39675 | 5.987 | 66.52 |
| 4 | (P+O)ES | (P+O) 进化策略 | 0.92256 | 0.88101 | 0.40021 | 2.20379 | 0.97750 | 0.87490 | 0.31945 | 2.17185 | 0.67385 | 0.62985 | 0.18634 | 1.49003 | 5.866 | 65.17 |
| 5 | CTA | 彗星尾算法 | 0.95346 | 0.86319 | 0.27770 | 2.09435 | 0.99794 | 0.85740 | 0.33949 | 2.19484 | 0.88769 | 0.56431 | 0.10512 | 1.55712 | 5.846 | 64.96 |
| 6 | SDSm | 随机扩散搜索 M | 0.93066 | 0.85445 | 0.39476 | 2.17988 | 0.99983 | 0.89244 | 0.19619 | 2.08846 | 0.72333 | 0.61100 | 0.10670 | 1.44103 | 5.709 | 63.44 |
| 7 | ESG | 社会群体的进化 | 0.99906 | 0.79654 | 0.35056 | 2.14616 | 1.00000 | 0.82863 | 0.13102 | 1.95965 | 0.82333 | 0.55300 | 0.04725 | 1.42358 | 5.529 | 61.44 |
| 8 | SIA | 模拟各向同性退火 | 0.95784 | 0.84264 | 0.41465 | 2.21513 | 0.98239 | 0.79586 | 0.20507 | 1.98332 | 0.68667 | 0.49300 | 0.09053 | 1.27020 | 5.469 | 60.76 |
| 9 | ACS | 人工协同搜索 | 0.75547 | 0.74744 | 0.30407 | 1.80698 | 1.00000 | 0.88861 | 0.22413 | 2.11274 | 0.69077 | 0.48185 | 0.13322 | 1.30583 | 5.226 | 58.06 |
| 10 | ASO | 无序社会优化 | 0.84872 | 0.74646 | 0.31465 | 1.90983 | 0.96148 | 0.79150 | 0.23803 | 1.99101 | 0.57077 | 0.54062 | 0.16614 | 1.27752 | 5.178 | 57.54 |
| 11 | TSEA | 龟壳演化算法 | 0.96798 | 0.64480 | 0.29672 | 1.90949 | 0.99449 | 0.61981 | 0.22708 | 1.84139 | 0.69077 | 0.42646 | 0.13598 | 1.25322 | 5.004 | 55.60 |
| 12 | DE | 差分进化 | 0.95044 | 0.61674 | 0.30308 | 1.87026 | 0.95317 | 0.78896 | 0.16652 | 1.90865 | 0.78667 | 0.36033 | 0.02953 | 1.17653 | 4.955 | 55.06 |
| 13 | CRO | 化学反应优化 | 0.94629 | 0.66112 | 0.29853 | 1.90593 | 0.87906 | 0.58422 | 0.21146 | 1.67473 | 0.75846 | 0.42646 | 0.12686 | 1.31178 | 4.892 | 54.36 |
| 14 | BSA | 鸟群算法 | 0.89306 | 0.64900 | 0.26250 | 1.80455 | 0.92420 | 0.71121 | 0.24939 | 1.88479 | 0.69385 | 0.32615 | 0.10012 | 1.12012 | 4.809 | 53.44 |
| 15 | HS | 和声搜索 | 0.86509 | 0.68782 | 0.32527 | 1.87818 | 0.99999 | 0.68002 | 0.09590 | 1.77592 | 0.62000 | 0.42267 | 0.05458 | 1.09725 | 4.751 | 52.79 |
| 16 | SSG | 树苗播种和生长 | 0.77839 | 0.64925 | 0.39543 | 1.82308 | 0.85973 | 0.62467 | 0.17429 | 1.65869 | 0.64667 | 0.44133 | 0.10598 | 1.19398 | 4.676 | 51.95 |
| 17 | (PO)ES | (PO) 进化策略 | 0.79025 | 0.62647 | 0.42935 | 1.84606 | 0.87616 | 0.60943 | 0.19591 | 1.68151 | 0.59000 | 0.37933 | 0.11322 | 1.08255 | 4.610 | 51.22 |
| 18 | BSO | 头脑风暴优化 | 0.93736 | 0.57616 | 0.29688 | 1.81041 | 0.93131 | 0.55866 | 0.23537 | 1.72534 | 0.55231 | 0.29077 | 0.11914 | 0.96222 | 4.498 | 49.98 |
| 19 | WOAm | 鲸鱼优化算法M | 0.84521 | 0.56298 | 0.26263 | 1.67081 | 0.93100 | 0.52278 | 0.16365 | 1.61743 | 0.66308 | 0.41138 | 0.11357 | 1.18803 | 4.476 | 49.74 |
| 20 | AEFA | 人工电场算法 | 0.87700 | 0.61753 | 0.25235 | 1.74688 | 0.92729 | 0.72698 | 0.18064 | 1.83490 | 0.66615 | 0.11631 | 0.09508 | 0.87754 | 4.459 | 49.55 |
| 21 | ACOm | 蚁群优化 M | 0.88190 | 0.66127 | 0.30377 | 1.84693 | 0.85873 | 0.58680 | 0.15051 | 1.59604 | 0.59667 | 0.37333 | 0.02472 | 0.99472 | 4.438 | 49.31 |
| 22 | BFO-GA | 细菌觅食优化 - ga | 0.89150 | 0.55111 | 0.31529 | 1.75790 | 0.96982 | 0.39612 | 0.06305 | 1.42899 | 0.72667 | 0.27500 | 0.03525 | 1.03692 | 4.224 | 46.93 |
| 23 | ABHA | 人工蜂巢算法 | 0.84131 | 0.54227 | 0.26304 | 1.64663 | 0.87858 | 0.47779 | 0.17181 | 1.52818 | 0.50923 | 0.33877 | 0.10397 | 0.95197 | 4.127 | 45.85 |
| 24 | ASBO | 适应性社会行为优化 | 0.76331 | 0.49253 | 0.32619 | 1.58202 | 0.79546 | 0.40035 | 0.26097 | 1.45677 | 0.26462 | 0.17169 | 0.18200 | 0.61831 | 3.657 | 40.63 |
| 25 | MEC | 思维进化计算 | 0.69533 | 0.53376 | 0.32661 | 1.55569 | 0.72464 | 0.33036 | 0.07198 | 1.12698 | 0.52500 | 0.22000 | 0.04198 | 0.78698 | 3.470 | 38.55 |
| 26 | IWO | 入侵杂草优化 | 0.72679 | 0.52256 | 0.33123 | 1.58058 | 0.70756 | 0.33955 | 0.07484 | 1.12196 | 0.42333 | 0.23067 | 0.04617 | 0.70017 | 3.403 | 37.81 |
| 27 | Micro-AIS | 微型人工免疫系统 | 0.79547 | 0.51922 | 0.30861 | 1.62330 | 0.72956 | 0.36879 | 0.09398 | 1.19233 | 0.37667 | 0.15867 | 0.02802 | 0.56335 | 3.379 | 37.54 |
| 28 | COAm | 布谷鸟优化算法 M | 0.75820 | 0.48652 | 0.31369 | 1.55841 | 0.74054 | 0.28051 | 0.05599 | 1.07704 | 0.50500 | 0.17467 | 0.03380 | 0.71347 | 3.349 | 37.21 |
| 29 | SDOm | 螺旋动力学优化 M | 0.74601 | 0.44623 | 0.29687 | 1.48912 | 0.70204 | 0.34678 | 0.10944 | 1.15826 | 0.42833 | 0.16767 | 0.03663 | 0.63263 | 3.280 | 36.44 |
| 30 | NMm | Nelder-Mead方法 M | 0.73807 | 0.50598 | 0.31342 | 1.55747 | 0.63674 | 0.28302 | 0.08221 | 1.00197 | 0.44667 | 0.18667 | 0.04028 | 0.67362 | 3.233 | 35.92 |
| 31 | FAm | 萤火虫算法 M | 0.58634 | 0.47228 | 0.32276 | 1.38138 | 0.68467 | 0.37439 | 0.10908 | 1.16814 | 0.28667 | 0.16467 | 0.04722 | 0.49855 | 3.048 | 33.87 |
| 32 | GSA | 引力搜索算法 | 0.64757 | 0.49197 | 0.30062 | 1.44016 | 0.53962 | 0.36353 | 0.09945 | 1.00260 | 0.32667 | 0.12200 | 0.01917 | 0.46783 | 2.911 | 32.34 |
| 33 | BFO | 细菌觅食优化 | 0.61171 | 0.43270 | 0.31318 | 1.35759 | 0.54410 | 0.21511 | 0.05676 | 0.81597 | 0.42167 | 0.13800 | 0.03195 | 0.59162 | 2.765 | 30.72 |
| 34 | ABC | 人工蜂群 | 0.63377 | 0.42402 | 0.30892 | 1.36671 | 0.55103 | 0.21874 | 0.05623 | 0.82600 | 0.34000 | 0.14200 | 0.03102 | 0.51302 | 2.706 | 30.06 |
| 35 | BA | 蝙蝠算法 | 0.59761 | 0.45911 | 0.35242 | 1.40915 | 0.40321 | 0.19313 | 0.07175 | 0.66810 | 0.21000 | 0.10100 | 0.03517 | 0.34617 | 2.423 | 26.93 |
| 36 | SA | 模拟退火 | 0.55787 | 0.42177 | 0.31549 | 1.29513 | 0.34998 | 0.15259 | 0.05023 | 0.55280 | 0.31167 | 0.10033 | 0.02883 | 0.44083 | 2.289 | 25.43 |
| 37 | IWDm | 智能水滴 M | 0.54501 | 0.37897 | 0.30124 | 1.22522 | 0.46104 | 0.14704 | 0.04369 | 0.65177 | 0.25833 | 0.09700 | 0.02308 | 0.37842 | 2.255 | 25.06 |
| 38 | PSO | 粒子群优化 | 0.59726 | 0.36923 | 0.29928 | 1.26577 | 0.37237 | 0.16324 | 0.07010 | 0.60572 | 0.25667 | 0.08000 | 0.02157 | 0.35823 | 2.230 | 24.77 |
| 39 | Boids算法 | 虚拟生物算法 | 0.43340 | 0.30581 | 0.25425 | 0.99346 | 0.35718 | 0.20160 | 0.15708 | 0.71586 | 0.27846 | 0.14277 | 0.09834 | 0.51957 | 2.229 | 24.77 |
| 40 | MA | 猴群算法 | 0.59107 | 0.42681 | 0.31816 | 1.33604 | 0.31138 | 0.14069 | 0.06612 | 0.51819 | 0.22833 | 0.08567 | 0.02790 | 0.34190 | 2.196 | 24.40 |
| 41 | SFL | 混合蛙跳算法 | 0.53925 | 0.35816 | 0.29809 | 1.19551 | 0.37141 | 0.11427 | 0.04051 | 0.52618 | 0.27167 | 0.08667 | 0.02402 | 0.38235 | 2.104 | 23.38 |
| 42 | FSS | 鱼群搜索 | 0.55669 | 0.39992 | 0.31172 | 1.26833 | 0.31009 | 0.11889 | 0.04569 | 0.47467 | 0.21167 | 0.07633 | 0.02488 | 0.31288 | 2.056 | 22.84 |
| 43 | RND | 随机 | 0.52033 | 0.36068 | 0.30133 | 1.18234 | 0.31335 | 0.11787 | 0.04354 | 0.47476 | 0.25333 | 0.07933 | 0.02382 | 0.35648 | 2.014 | 22.37 |
| 44 | GWO | 灰狼优化算法 | 0.59169 | 0.36561 | 0.29595 | 1.25326 | 0.24499 | 0.09047 | 0.03612 | 0.37158 | 0.27667 | 0.08567 | 0.02170 | 0.38403 | 2.009 | 22.32 |
| 45 | CSS | 人工电场算法 | 0.44252 | 0.35454 | 0.35201 | 1.14907 | 0.24140 | 0.11345 | 0.06814 | 0.42299 | 0.18333 | 0.06300 | 0.02322 | 0.26955 | 1.842 | 20.46 |
总结
根据AMOm算法在测试函数上的结果,可以得出以下结论:尽管在小维度函数上值的分布范围较大,但该算法在大维度函数上展现出了出色的可扩展性。对算法原始版本所做的重大改进显著提升了其性能。在这种情况下,通过将父代种群规模加倍(以便与子代个体一起进行排序)并改变算法各阶段的执行顺序,我们获得了更广泛、更多样化的解决方案。该算法成为了使用附加技术进行修改的可行性的一个明确例证,并取得了显著改善。这得益于算法逻辑本身的改进,而这种改进并不总是适用于其他优化算法。
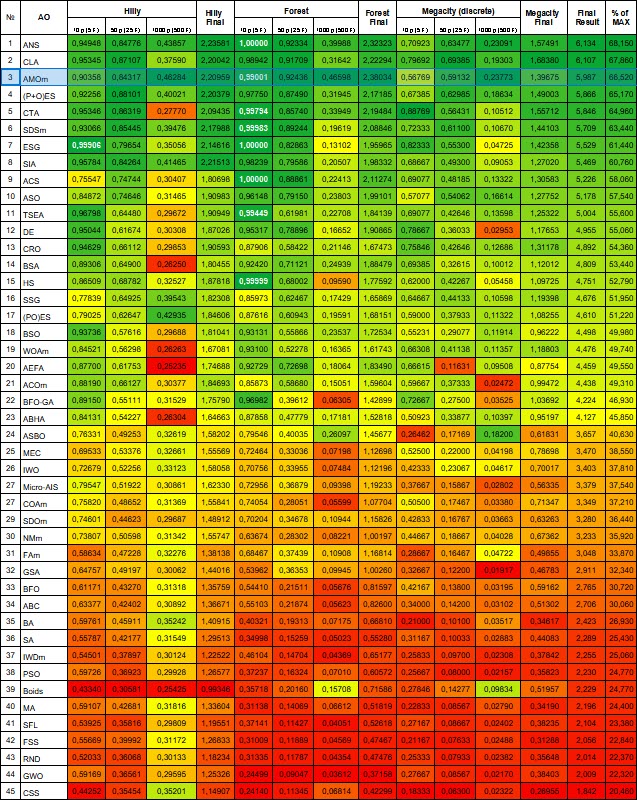
图例2. 根据相关测试,将算法的颜色等级大于或等于0.99的结果以白色突出显示。
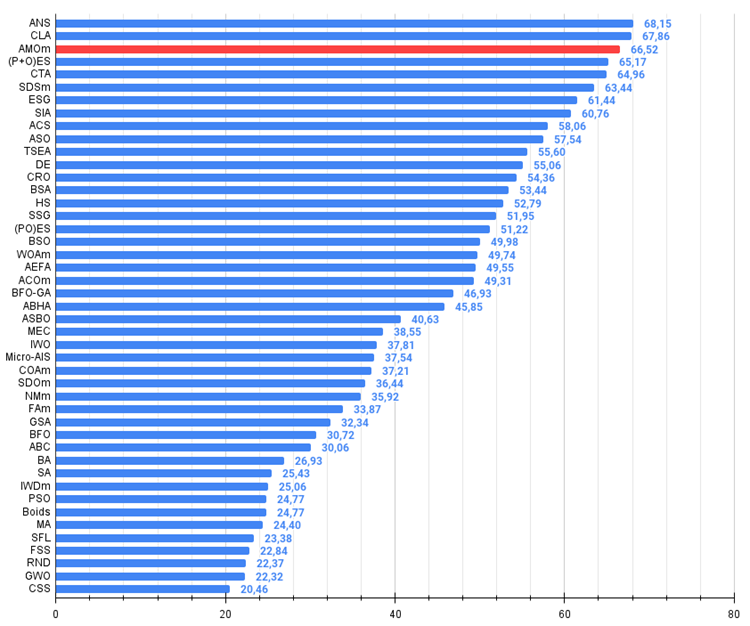
图例 3. 算法测试结果的直方图(标尺从 0 到 100,数字越大结果越好,
其中 100 是理论上的最大可能结果,将计算评级表格的脚本存档)
AMOm的优缺点
优势:
- 出色的收敛性。
- 较高的可扩展性。
- 较少的参数。
- 实现简单。
缺点:
- 低维函数结果分散。
文章附有一个包含当前版本算法代码的归档文件。本文作者对标准算法描述的绝对准确性不承担责任。为提升搜索能力,已经对其中的许多算法进行了修改。文章中表述的结论和论断都是基于实验的结果。
本文由MetaQuotes Ltd译自俄文
原文地址: https://www.mql5.com/ru/articles/15543
 构建K线图趋势约束模型(第8部分):EA的开发(一)
构建K线图趋势约束模型(第8部分):EA的开发(一)
 开发多币种 EA 交易系统(第 16 部分):不同报价历史对测试结果的影响
开发多币种 EA 交易系统(第 16 部分):不同报价历史对测试结果的影响
 神经网络变得简单(第 96 部分):多尺度特征提取(MSFformer)
神经网络变得简单(第 96 部分):多尺度特征提取(MSFformer)