
Uso práctico de las redes neuronales de Kohonen en el trading algorítmico (Parte I) Instrumental
El tema de las redes neuronales de Kohonen ya fue abordado en algunos artículos del sitio web mql5.com, a saber: Utilizar mapas con funciones de auto-organización (mapas Kohonen) en MetaTrader 5 y Hablando de nuevo sobre los mapas de Kohonen. Los lectores de estos artículos conocieron los principios generales de la construcción de redes neuronales de este tipo y el análisis visual de los indicadores económicos usando los mapas.
No obstante, en términos prácticos, la aplicación de la red de Kohonen precisamente para el trading algorítmico fue limitado con un solo enfoque, es decir, con el análisis visual de los mapas topológicos formados para los resultados de la optimización del Asesor Experto (EA). En este caso, la opinión subjetiva -o más bien, la percepción- de una persona y su capacidad de sacar conclusiones de la imagen resulta ser un factor determinante, relegando a segundo plano las capacidades de la red de representar los datos en relación con las categorías prácticas.
En otras palabras, las capacidades de los algoritmos de redes neuronales no fueron utilizados al máximo, o sea, sin la extracción automática de conocimientos y soporte de la toma de decisiones con recomendaciones específicas. En este artículo, vamos a considerar el problema de la definición de los conjuntos óptimos de los parámetros de los robots en una forma más formalizada. Además, aplicaremos la red de Kohonen para la previsión de las series económicas. Pero antes de empezar con estas tareas aplicadas, hace falta revisar los códigos fuente existentes, corregir algunos errores y hacer las mejoras.
A los que no están familiarizados con los términos como «red», «capa», «neurona» (nodo), «», «peso», «velocidad de aprendizaje», «rango de aprendizaje», y otras nociones relacionadas con las redes de Kohonen, les recomiendo insistentemente leer primero los artículos arriba mencionados. A continuación, tendremos que enfrascarnos en este tema, así que, la repetición de las nociones básicas prolongaría significativamente esta publicación.
Corrigiendo errores
Vamos a invocar las clases CSOM y CSOMNode publicadas en el primer artículo, tomando en cuenta las adiciones en el segundo artículo. Los fragmentos principales en ellas son prácticamente idénticas y heredan problemas comunes.
En primer lugar, hay que notar que, por algunas razones, las neuronas en estas clases se indexan (se identifican y se definen a través de los parámetros de los constructores) por coordenadas de píxel. Eso no es muy lógico y complica los cálculos y la depuración en algunos puntos. En particular, con este enfoque, los ajustes de presentación afectan los cálculos. Sólo imagínese: hay dos instancias de las redes absolutamente idénticas, con tamaños iguales de la cuadrícula y aprenden usando el mismo conjunto de datos, tienen las configuraciones iguales e inicialización del generador de números aleatorios. Sin embargo, los resultados obtenidos son diferentes sólo porque las imágenes de una red son más grandes que de la otra. Es un error.
Vamos a indexar las neuronas por números: cada neurona del array m_node (clase CSOM) va a tener las coordenadas «x» y «y» que corresponden a los números de la columna y la línea en la capa de salida de la red de Kohonen. Cada neurona va a inicializarse a través del método CSOMNode::InitNode(x, y) en vez de CSOMNode::InitNode(x1, y1, x2, y2). Cuando pasaremos a la visualización, las coordenadas de la neurona permanecerán inalteradas al alterar los tamaños del mapa en píxeles.
En los códigos fuente heredados, la normalización de los datos de entrada no se usa. Sin embargo, ella es muy importante cuando diferentes componentes (características) de los vectores de entrada tienen un rango de valores diferente. Es así tanto para los resultados de la optimización de los EAs, como durante el agrupamiento de los valores de diferentes indicadores. En cuanto a los resultados de la optimización, podemos ver que los valores con un beneficio neto de decenas de millares están juntos con los valores pequeños del coeficiente de Sharp o con valores de un dígito del factor de recuperación.
No se puede enseñar a la red de Kohonen usando los datos de la escala tan diferente, porque la red va a considerar prácticamente sólo los componentes grandes e ignorar los pequeños. Usted puede ver eso en la imagen de abajo obtenida a través del programa que será considerado paso a paso en este artículo y adjuntado al final. En el programa existe la posibilidad de generar los vectores de entrada aleatorios en los cuales tres componentes están determinados dentro de los rangos [0, 1000], [0, 1] y [-1, +1]. El parámetro de entrada especial UseNormalization permite activar/desactivar la normalización.
Vamos a ver la estructura de la red de Kohonen en tres planos correspondientes a tres dimensiones de los vectores. Primero, veremos el resultado del aprendizaje de la red sin normalización.

Resultado del aprendizaje de la red de Kohonen sin normalización de los datos de entrada
Ahora, veremos los mismo con normalización.
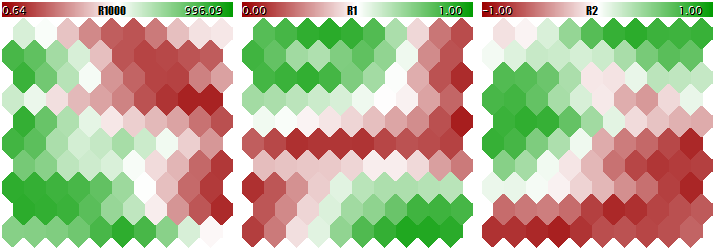
Resultado del aprendizaje de la red de Kohonen con normalización de los datos de entrada
El grado de adaptación de los pesos de neuronas es proporcional al gradiente de color. Obviamente, a falta de la normalización, la red aprende la división topológica (clasificación) solamente en el primer plano, mientras que el segundo y el tercer componente llevan un pequeño ruido. Es decir, las capacidades analíticas han sido implementadas solamente a un tercio. En caso de la normalización activada, la estructura espacial es visible en todos los tres planos.
Se conocen muchos métodos de la normalización, pero el más popular, tal vez, sea la sustracción del valor medio de toda la muestra de cada componente, y la división por la desviación estándar (sigma, raíz cuadrada de la dispersión). Eso lleve el valor medio de datos transformados a cero, y la desviación estándar a uno.
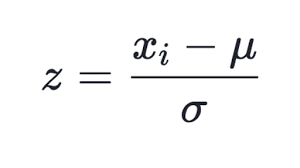 (1)
(1)
Esta técnica se usa en la clase actualizada CSOM, en el método Normalize. Esta claro que primero hace falta calcular la media y la sigma para cada componente del conjunto de datos de entrada, lo que se hace en el método InitNormalization (véase más abajo).
Las fórmulas canónicas del cálculo de la media y de la desviación estándar suponen un algoritmo de dos pasos: en primer lugar, se busca la media, y después, se calcula la sigma usando la media.
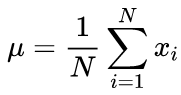 (2)
(2)
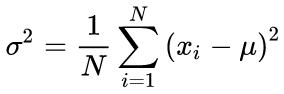 (3)
(3)
En nuestro código fuente, se usa el algoritmo de un paso basado en la siguiente fórmula:
 (4)
(4)
Obviamente, la normalización en la entrada requiere una operación inversa (denormalización) en la salida, es decir, al transformar los valores de salida de la red en el rango de valores reales. De eso se encarga el método CSOM::Denormalize.
Puesto que los valores normalizados caen simétricamente alrededor de cero, vamos a cambiar el principio de la inicialización de los pesos de neuronas antes del aprendizaje: en vez del rango [0, 1], ahora será el rango [-1, +1] (véase el método CSOMNode::InitNode). Eso aumentará la eficacia del aprendizaje de la red.
Otro aspecto a ser corregido es el cálculo de las iteraciones del aprendizaje. En las clases fuente, la iteración se entiende como la presentación de cada vector de entrada individual a la red. Por tanto, el número de iteraciones se corrige a base de y de acuerdo con el tamaño de la muestra de aprendizaje. Recuerde que el principio del aprendizaje y de la generalización de la información por la red de Kohonen supone que cada muestra se presenta a la red muchas veces. Por ejemplo, si la muestra incluye 100 entradas, entonces para repasar cada una de ellas 100 veces (como media), habrá que establecer el número de iteraciones igual a 10 000. Sin embargo, si la muestra es de 10 000 entradas, el número de iteraciones tendrá que ser 100 000 El método más conveniente y convencional para establecer el número de, así llamadas, las «épocas del aprendizaje», es decir, los ciclos dentro de cada uno de los cuales todas las instancias se pasan aletoriamente a la entrada de la red. Este número será definido en el parámetro EpochNumber. Debido a su introducción, la duración del aprendizaje se separa parametricamente del tamaño del conjunto de datos.
Es más aun importante, ya que el conjunto general de datos puede ser dividido en dos componentes, en caso de necesidad: la muestra de aprendizaje y la llamada muestra de validación. La última se aplica para monitorear la calidad del aprendizaje de la red. La cosa es que la adaptación de la red a los datos de entrada durante el aprendizaje tiene el «reverso de la medalla»: la red empieza a adaptarse a las particularidades específicas de las instancias, y así, pierde la capacidad de generalizar y funcionar adecuadamente en datos nunca vistos antes (diferentes de los que han sido usados para el aprendizaje). Y eso que la idea del aprendizaje, normalmente, consiste en utilizar las particularidades detectadas a través de la red en el futuro.
En el programa considerado, el parámetro ValidationSetPercent se encarga de la activación de la validación. Por defecto, es igual a 0, y todos los datos se usan para el aprendizaje. Si especificamos, digamos, el valor 10, entonces sólo 90% de las instancias se usan para el aprendizaje. Para los 10% restantes se calcula el error medio cuadrático normalizado relativo (Normalized Mean Squared Error) en cada iteración (época), y el proceso de aprendizaje se detiene en el momento cuando el error empieza a crecer.
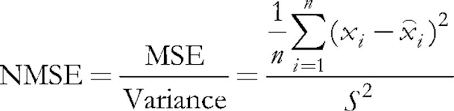 (5)
(5)
La normalización consiste en la división del error medio cuadrático por la dispersión de los mismos datos, lo que resulta que el índice siempre está por debajo de 1. Al considerar cada vector por separado, este error medio cuadrático, en realidad, representa un error de cuantización, porque se basa en la diferencia entre sus componentes y los pesos de las sinapses correspondientes de las neuronas, dando la mejor aproximación de este vector entre todas las neuronas. Recordemos que en las redes de Kohonen esta neurona ganadora se llama BMU (best matching unit) o BMN (best matching node). El método GetBestMatchingNode se encarga de su búsqueda en la clase CSOM.
Al activar la validación, el número de las iteraciones va a superar los valores establecidos en el parámetro EpochNumber. Debido a las particularidades de la arquitectura de la red de Kohonen, la validación puede realizarse sólo después de que la red pase la fase de autoorganización en las épocas EpochNumber. Después de que se termine esta fase, la velocidad de aprendizaje se disminuye de forma que se empieza el ajuste fino de los pesos y se empieza la fase de la convergencia. Es aquí donde se usa la «interrupción precoz» del aprendizaje a través del conjunto de validación.
La validación se usa o no, dependiendo de la especificación del problema. Además de eso, el conjunto de validación puede ser usado para ajustar el tamaño de la red. En este artículo, no vamos a entrar en este asunto. Usaremos una conocida regla empírica que relaciona el tamaño de la red con el número de datos de aprendizaje:
N ~ 5 * sqrt(M) (6)
aquí, N es el número de neuronas en la red, M es el número de los vectores de entrada. Para una red de Kohenen con la capa de salida cuadrada, obtenemos el siguiente tamaño:
S = sqrt(5 * sqrt(M)) (7)
aquí, S es el número de neuronas por la vertical y por la horizontal. Vamos a introducir este valor en los parámetros CellsX y CellsY.
El último problema que tenemos que corregir en los códigos fuente originales concierne al procesamiento de la cuadrícula hexagonal. Como se sabe, los mapas de Kohonen normalmente se construyen usando la colocación rectangular o hexagonal de las células (neuronas), siendo que ambos modos están realizados inicialmente en los códigos fuente. Sin embargo, la cuadrícula hexagonal se muestra en forma de las células, pero se calcula igualmente como la rectangular. Para llegar a la raíz del error, vamos a analizar la siguiente ilustración.
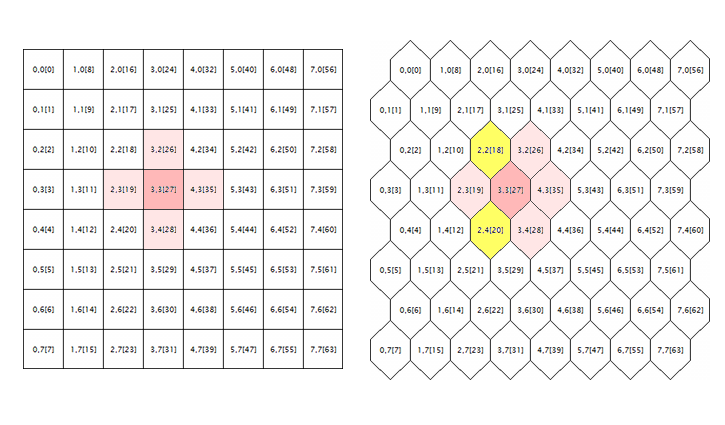
Geometría de los alrededores de la neurona en la cuadrícula rectangular y hexagonal
Aquí se muestra el entorno lógico de una neurona aleatoria (en este caso, con coordenadas 3;3) para las cuadrículas de ambas geometrías. El radio del entorno es igual a 1. En la cuadrícula cuadrada, la neurona tiene 4 vecinos; y en la cuadrícula hexagonal, son 6. La implementación de la apariencia celular se consigue a través del desplazamiento de cada segunda línea de las células a la mitad de la célula al lado. Sin embargo, sus coordenadas internas no se alteran, y desde el punto de vista del algoritmo, el entorno de la neurona en la cuadrícula hexagonal aparece como antes (está marcada en color rosa).
Aparentemente, no es correcto y tiene que ser corregido mediante la inclusión de las neuronas marcadas en amarillo.
Formalmente, el algoritmo calcula el entorno no sólo usando los vecinos adyacentes, sino como una función radial decreciente convexa que depende de la distancia entre las coordenadas de las células. En otras palabras, la vecindad no es una propiedad binaria de la neurona (sea un vecino o no), sino un valor continuo que se calcula a través de la fórmula de Gaussian:
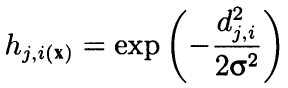 (8)
(8)
Aquí, dji es la distancia entre las neuronas j y i (se tiene en cuenta la numeración continua, en vez de las coordenadas X y Y); la sigma es el ancho eficaz del entorno o el rango de aprendizaje que se reduce gradualmente durante el aprendizaje. Al principio del aprendizaje, el entorno cubre con una «campana» un espacio más grande que las neuronas adyacentes.
Puesto que esta fórmula depende de la distancia, ella también altera el entorno si las coordenadas no han sido corregidas de manera correspondiente. Por tanto, las siguientes líneas del código fuente original del método CSOM::Train:
for(int i = 0; i < total_nodes; i++) { double DistToNodeSqr = (m_som_nodes[winningnode].X() - m_som_nodes[i].X()) * (m_som_nodes[winningnode].X() - m_som_nodes[i].X()) + (m_som_nodes[winningnode].Y() - m_som_nodes[i].Y()) * (m_som_nodes[winningnode].Y() - m_som_nodes[i].Y());
han sido completadas:
bool odd = ((winningnode % m_ycells) % 2) == 1; for(int i = 0; i < total_nodes; i++) { bool odd_i = ((i % m_ycells) % 2) == 1; double shiftx = 0; if(m_hexCells && odd != odd_i) { if(odd && !odd_i) { shiftx = +0.5; } else // vice versa (!odd && odd_i) { shiftx = -0.5; } } double DistToNodeSqr = (m_node[winningnode].GetX() - (m_node[i].GetX() + shiftx)) * (m_node[winningnode].GetX() - (m_node[i].GetX() + shiftx)) + (m_node[winningnode].GetY() - m_node[i].GetY()) * (m_node[winningnode].GetY() - m_node[i].GetY());
La dirección de la corrección shiftx depende de la relación de las propiedades par-impar de las líneas donde se encuentran dos neuronas entre las cuales se calcula la distancia. Si las neuronas las neuronas se encuentran en las líneas igualmente niveladas, entonces no hay corrección. Si la neurona ganadora se encuentra en una línea impar, entonces las líneas pares respecto a ella aparentan desplazadas a la mitad de la célula a la derecha, por eso shiftx es igual a +0,5. Si la neurona ganadora se encuentra en una línea par, entonces las líneas impares respecto a ella aparentan desplazadas a la mitad de la célula a la izquierda, por eso shiftx es igual a -0,5.
Ahora, es especialmente importante prestar atención a las siguientes líneas originales:
if(DistToNodeSqr < WS) { double influence = MathExp(-DistToNodeSqr / (2 * WS)); m_node[i].AdjustWeights(data, learning_rate, influence); }
Este operador condicional garantiza prácticamente una cierta aceleración de los cálculos a través del rechazo de las neuronas fuera del entorno de una sigma. No obstante, desde el punto de vista de la calidad del aprendizaje, la fórmula de Gaussianes una forma ideal y esta intervención no es razonable. Si realizamos la omisión de las neuronas demasiado lejanas, entonces por tres sigmas, y no por una. Y eso es aún más crítico después de que hayamos corregido el cálculo de la cuadrícula hexagonal, porque la distancia entre las neuronas adyacentes ubicadas en las líneas vecinas es igual a sqrt(1*1 + 0.5*0.5) = 1.118, es decir, más de uno. En los códigos adjuntos, este operador condicional está comentado. Si surge la necesidad de acelerar los cálculos, utilice la siguiente opción:
if(DistToNodeSqr < 9 * WS)
¡ATENCIÓN! Debido al detalle especificado de la diferencia de las distancias entre las neuronas adyacentes dependiendo de su línea (las líneas únicas tienen la distancia 1, las líneas adyacentes, 1,118), la implementación actual no es ideal y necesita la siguiente corrección para alcanzar la anisotropía completa.
Visualización
A pesar de que las redes de Kohonen se asocian principalmente con un mapa gráfico visible, su topología y los algoritmos de aprendizaje pueden funcionar perfectamente sin la interfaz de usuarios. Particularmente, las tareas de predecir o compactar la información no requieren ningún análisis visual obligatorio, mientras que la clasificación de las imágenes puede mostrar el resultado en forma de un número (número de la clase o la probabilidad del evento). Por tanto, la funcionalidad de las redes de Kohonen fue dividida entre dos clases. En la clase CSOM, queda sólo la parte del cálculo, la carga y el guardado de los datos, la carga y el guardado de las redes. Adicionalmente, fue creada la clase derivada CSOMDisplay que contiene la gráfica. Según mi parecer, es una jerarquía más simple y más lógica de la propuesta en el artículo 2. A continuación, para las tareas de la selección de los parámetros óptimos del EA vamos a usar CSOMDisplay, y para la previsión, usaremos CSOM.
Nótese que el indicio tipo cuadrícula (rectangular o hexagonal) pertenece a la clase base, puesto que influye en el cálculo de las distancias. Juntamente con el número de los nodos por la vertical y horizontal, así como la dimensionalidad del espacio de entrada de los datos, el tipo de la cuadrícula forma parte de la arquitectura de la red y se guarda en el archivo. Cuando la red se carga desde el archivo, todos estos parámetros se leen precisamente desde ahí, y no desde las configuraciones del programa. Otras configuraciones que influyen en la representación gráfica, tales como, el tamaño de los mapas en píxeles, visualización de los límites entre las celdas, visualización de las leyendas, no se guardan en el archivo de la red y pueden alterarse repetidamente y aleatoriamente para una red después de su aprendizaje.
Cabe destacar que ninguna de las clases actualizadas proporciona la interfaz gráfica de usuario con los controles, es que todas las configuraciones se establecen a través de los parámetros de entrada de los programas MQL. Al mismo tiempo, la clase CSOMDisplay realiza algunos recursos útiles.
Vamos a recordar que en los ejemplos anteriores del trabajo con las redes de Kohonen, había un parámetro de entrada MaxPictures, y él ha sido dejado en la nueva implementación. Se pasa como maxpict al método CSOMDisplay::Init y establece el número de los mapas (planos) de la red que se muestran en el gráfico en una línea. operando con este parámetro junto con el tamaño unificado de las imágenes ImageW y ImageH, podemos encontrar una opción cuando todos los mapas caben en la pantalla. No obstante, cuando hay muchos mapas (por ejemplo, cuando necesitamos analizar muchas configuraciones del EA), tenemos que reducir su tamaño, y eso no es muy conveniente. En estos casos, usamos MaxPictures para activar el nuevo modo, estableciendo el parámetro a 0.
En este modo, las visualizaciones de los mapas no se generan en el gráfico como objetos OBJ_BITMAP_LABEL con la vinculación a las coordenadas de los píxeles, sino en forma de los objetos OBJ_BITMAP con vinculación a la escala de tiempo. El tamaño de estos mapas puede ser aumentado hasta la altura total del gráfico, y Usted puede ojearlos usando la barra de desplazamiento horizontal a través del ratón, la rueda o el teclado. El número de los mapas ya no se limita con el tamaño de la pantalla, pero tenemos que asegurarnos de la presencia del número suficiente de las barras.
El aumento de los tamaños de los mapas permite estudiarlos más detalladamente, especialmente porque la clase CSOMDisplay muestra opcionalmente varios datos dentro de las celdas, como el valor del peso de sinapsis del plano correspondiente, número de las ocurrencias de los vectores del conjunto de aprendizaje, el valor medio y la dispersión de los valores del indicio correspondiente de todos los vectores que entran en la celda. Esta información no se muestra por defecto, pero siempre está disponible en las ayudas emergentes que aparecen si mantenemos el cursor del ratón sobre alguna celda. Además, en las ayudas se muestra el nombre del plano actual y las coordenadas de la neurona.
Aparte de eso, si hacemos doble clic en cualquier neurona, se resalta con el color inverso en el mapa actual, así como en el resto de los mapas. Eso permite comparar visualmente la actividad de las neuronas por todos los recursos simultáneamente.
Finalmente, cabe mencionar que la gráfica entera ha sido pasada a la clase estándar CCanvas. Eso libera el código de las dependencias externas, pero tiene un efecto secundario: las coordenadas Y ahora se calculan de arriba abajo y no de abajo arriba como antes. Como resultado, la leyenda de los mapas con el nombre de los componentes y los rangos de sus valores se muestra por encima de los mapas y no debajo, pero este cambio no parece ser crítico.
Mejoras
Antes de proceder a las tareas aplicadas, hay que mejorar las clases de la red neuronal. En adición a los mapas estándar que representan los pesos de sinapsis en los espacios 2D de características específicas, vamos a preparar el cálculo y visualización de algunos mapas de servicio que representan un estándar de-facto para las redes de Kohonen. Hablando con adelantado, digamos que vamos a necesitar muchos de ellos en la fase de los experimentos aplicados.
Vamos a definir los índices de las dimensiones adicionales (en total, serán 5).
#define EXTRA_DIMENSIONS 5 #define DIM_HITCOUNT (m_dimension + 0) #define DIM_UMATRIX (m_dimension + 1) #define DIM_NODEMSE (m_dimension + 2) // quantization errors per node: average variance (square of standard deviation) #define DIM_CLUSTERS (m_dimension + 3) #define DIM_OUTPUT (m_dimension + 4)
U-Matrix
Primeramente, vamos a calcular la matriz unificada de las distancias U-matrix para evaluar la topología generada en el proceso del aprendizaje dentro de la red. Para cada neurona en la red, esta matriz contiene la distancia media entre esta neurona y sus vecinos inmediatos. Puesto que la red de Kohonen muestra un espacio multidimensional de los recursos en el espacio bidimensional del mapa, en este espacio bidimensional surgen los pliegues. En otras palabras, a pesar de la propiedad de la red de Kohonen de mantener el orden inherente al espacio original, eso es imposible igualmente en el plano 2D entero, y en algunas ocasiones la proximidad geográfica de las neuronas se hace engañosa. Para detectar estas áreas, se usa U-matrix. Ahi, las zonas donde existe una diferencia grande entre los pesos de la neurona y los pesos de sus vecinos tienen la apariencia de los «picos», las zonas donde las neuronas son muy parecidas parecen a los «valles».
Para calcular la distancia entre la neurona y el vector de características, existe el método CSOMNode::CalculateDistance. Vamos a crear un método de contraparte que va a obtener el puntero a otra neurona en vez del vector (array double).
double CSOMNode::CalculateDistance(const CSOMNode *other) const { double vector[]; other.GetCodeVector(vector); return CalculateDistance(vector); }
Aquí, el método GetCodeVector obtiene el array con los pesos de otra neurona, y lo envía inmediatamente para el cálculo de la distancia de manera común.
Para obtener la distancia de la neurona unificada, hay que calcular la distancia con todas las neuronas vecinas y promediarla. Puesto que el repaso de las neuronas vecinas es una tarea común para varias operaciones en la cuadrícula de la red, creamos la clase base para el repaso, y luego, implementamos los algoritmos determinados en sus descendientes, incluyendo la suma de las distancias.
#define NBH_SQUARE_SIZE 4 #define NBH_HEXAGONAL_SIZE 6 template<typename T> class Neighbourhood { protected: int neighbours[]; int nbhsize; bool hex; int m_ycells; public: Neighbourhood(const bool _hex, const int ysize) { hex = _hex; m_ycells = ysize; if(hex) { nbhsize = NBH_HEXAGONAL_SIZE; ArrayResize(neighbours, NBH_HEXAGONAL_SIZE); neighbours[0] = -1; // up (visually) neighbours[1] = +1; // down (visually) neighbours[2] = -m_ycells; // left neighbours[3] = +m_ycells; // right /* template, applied dynamically in the loop below // odd row neighbours[4] = -m_ycells - 1; // left-up neighbours[5] = -m_ycells + 1; // left-down // even row neighbours[4] = +m_ycells - 1; // right-up neighbours[5] = +m_ycells + 1; // right-down */ } else { nbhsize = NBH_SQUARE_SIZE; ArrayResize(neighbours, NBH_SQUARE_SIZE); neighbours[0] = -1; // up (visually) neighbours[1] = +1; // down (visually) neighbours[2] = -m_ycells; // left neighbours[3] = +m_ycells; // right } } ~Neighbourhood() { ArrayResize(neighbours, 0); } T loop(const int ind, const CSOMNode &p_node[]) { int nodes = ArraySize(p_node); int j = ind % m_ycells; if(hex) { int oddy = ((j % 2) == 1) ? -1 : +1; neighbours[4] = oddy * m_ycells - 1; neighbours[5] = oddy * m_ycells + 1; } reset(); for(int k = 0; k < nbhsize; k++) { if(ind + neighbours[k] >= 0 && ind + neighbours[k] < nodes) { // skip wrapping edges if(j == 0) // upper row { if(k == 0 || k == 4) continue; } else if(j == m_ycells - 1) // bottom row { if(k == 1 || k == 5) continue; } iterate(p_node[ind], p_node[ind + neighbours[k]]); } } return getResult(); } virtual void reset() = 0; virtual void iterate(const CSOMNode &node1, const CSOMNode &node2) = 0; virtual T getResult() const = 0; };
Dependiendo del tipo de la cuadrícula pasada al constructor, el número de los vecinos nbhsize será considerado igual a 4 y 6. Los incrementos de los números de las neuronas vecinas respecto a la neurona actual se guarda en el array neighbours. Por ejemplo, en la cuadrícula cuadrada, el vecino de arriba se obtiene restando 1, y el vecino de abajo, sumando 1 al número de la neurona. Los vecinos de la izquierda y de la derecha tienen los números que se diferencian por la altura de la columna de la cuadrícula, por eso, este valor se pasa al constructor como ysize.
El repaso de los vecinos se realiza por el método loop. La clase Neighbourhood no incluye ningún array de neuronas, y por eso, se pasa al método loop como un parámetro.
Este método repasa cíclicamente el array neighbours y verifica adicionalmente que el número del vecino no salga fuera de la cuadrícula, teniendo en cuenta el incremento. Para todos los números válidos, se invoca el método abstracto iterate, a donde se pasan las referencias a la neurona actual y una de las neuronas del entorno.
Antes del ciclo se invoca el método abstracto reset, y después del ciclo, el método abstracto getResult. El conjunto de tres métodos abstractos permite preparar y ejecutar la iteración de los vecinos en las clases descendientes, así como generar el resultado. El concepto de la construcción del método «loop» corresponde al patrón de la proyección de la POO (método de plantilla). Aquí, se debe distinguir el término «plantilla» en el nombre del patrón, de la construcción de lenguaje de las plantillas que también se usa en la clase Neighbourhood, porque es una plantilla (es decir, es parametrizado por un determinado tipo variable T). En particular, el propio método loop y el método getResult devuelven el valor tipo T.
Vamos a escribir la clase para el cálculo U-matrix a base de la clase Neighbourhood.
class UMatrixNeighbourhood: public Neighbourhood<double> { private: int n; double d; public: UMatrixNeighbourhood(const bool _hex, const int ysize): Neighbourhood(_hex, ysize) { } virtual void reset() override { n = 0; d = 0.0; } virtual void iterate(const CSOMNode &node1, const CSOMNode &node2) override { d += node1.CalculateDistance(&node2); n++; } virtual double getResult() const override { return d / n; } };
El tipo de trabajo es double. Gracias a la clase base, los cálculos de la distancia son bastante transparentes.
Vamos a calcular las distancias de todo el mapa en el método CSOM::CalculateDistances.
void CSOM::CalculateDistances() { UMatrixNeighbourhood umnh(m_hexCells, m_ycells); for(int i = 0; i < m_xcells * m_ycells; i++) { double d = umnh.loop(i, m_node); if(d > m_max[DIM_UMATRIX]) { m_max[DIM_UMATRIX] = d; } m_node[i].SetDistance(d); } }
El valor de la distancia unificada se guardan en el objeto de la neurona. Posteriormente, al mostrar todos los planos, podremos definir los valores de las distancias de manera estándar usando una paleta de colores, incluyendo la dimensión adicional DIM_UMATRIX en el cálculo. Para escalar la paleta correctamente, guardamos en este método el valor máximo de la distancia dentro del elemento correspondiente del array m_max (todos los principios de la visualización permanecen inalterados desde las implementaciones anteriores).
Número de ocurrencias y error de cuantización
La siguiente dimensión adicional va a recopilar las estadísticas respecto al número de ocurrencias de los vectores de aprendizaje en las determinadas neuronas. En otras palabras, es la densidad de la población de las neuronas con datos aplicados. Cuanto más alta sea para una neurona específica, más justificados serán sus coeficientes de ponderación. En la red, pueden aparecer las neuronas con una cobertura de datos pequeña o incluso nula. Si son muchas, eso puede indicar en los problemas en la selección del tamaño de la red o la distorsión de la topología en la proyección 2D del espacio multidimensional. Para calcular las ocurrencias de las instancias en una determinada neurona, se usa el método:
void CSOMNode::RegisterPatternHit(const double &vector[]) { m_hitCount++; double e = 0; for(int i = 0; i < m_dimension; i++) { m_sum[i] += vector[i]; m_sumP2[i] += vector[i] * vector[i]; e += (m_weights[i] - vector[i]) * (m_weights[i] - vector[i]); } m_mse += e / m_dimension; }
El cálculo se realiza en la primera línea m_hitCount++, donde se aumenta el contador interno. El resto del código realiza otro trabajo útil del que hablaremos más abajo.
La llamada al método RegisterPatternHit va a realizarse después de la finalización del aprendizaje, desde la clase CSOM, donde crearemos un método especial del procesamiento estadístico de cada vector.
double CSOM::AddPatternStats(const double &data[]) { static double vector[]; ArrayCopy(vector, data); int ind = GetBestMatchingIndex(vector); m_node[ind].RegisterPatternHit(vector); double code[]; m_node[ind].GetCodeVector(code); Denormalize(code); double mse = 0; for(int i = 0; i < m_dimension; i++) { mse += (data[i] - code[i]) * (data[i] - code[i]); } mse /= m_dimension; return mse; }
Como una digresión, cabe mencionar que el método GetBestMatchingIndex (utilizado aquí), como algunos otros del grupo de los métodos GetBestMatchingXYZ, normaliza los datos de entrada dentro de sí mismo, y por esa razón hay que pasarle una copia del vector (en caso contrario, sería abierta una posibilidad de la modificación difusa de los datos de origen en el código de la llamada).
Aparte del registro de una ocurrencia, este método calcula el error de cuantización para la neurona actual y para el vector pasado. Para eso, a la neurona vencedora se le solicita así llamado el vector de código (code vector), es decir, el array de los pesos de sinapsis, y se calcula la suma de los cuadrados de las diferencias de los componentes entre los pesos y el vector de entrada.
AddPatternStats se invoca desde otro método CSOM::CalculateStats, que simplemente organiza el ciclo por todos los datos de entrada.
double CSOM::CalculateStats(const bool complete = true) { double data[]; ArrayResize(data, m_dimension); double trainedMSE = 0.0; for(int i = complete ? 0 : m_validationOffset; i < m_nSet; i++) { ArrayCopy(data, m_set, 0, m_dimension * i, m_dimension); trainedMSE += AddPatternStats(data, complete); } double nmse = trainedMSE / m_dataMSE; if(complete) Print("Overall NMSE=", nmse); return nmse; }
Este método suma todos los errores de cuantización y los compara con la dispersión de los datos de entrada en m_dataMSE (eso es exactamente el cálculo NMSE del que hemos hablado antes en el contexto de la validación y la detención del aprendizaje). En este método, se menciona la variable m_validationOffset, que se establece al crear el objeto CSOM a base de que si se usa o no la división del conjunto de datos de entrada por el subconjunto de aprendizaje y de validación.
Usted puede fácilmente darse cuenta de que el método CalculateStats se invoca en cada época dentro del método Train (si la fase de la convergencia ya ha sido iniciada), y puede juzgar por el valor devuelto si el error general de la red ha empezado a crecer, es decir, si es el momento de parar.
La dispersión m_dataMSE se calcula de antemano a través del método:
void CSOM::CalculateDataMSE() { double data[]; m_dataMSE = 0.0; for(int i = m_validationOffset; i < m_nSet; i++) { ArrayCopy(data, m_set, 0, m_dimension * i, m_dimension); double mse = 0; for(int k = 0; k < m_dimension; k++) { mse += (data[k] - m_mean[k]) * (data[k] - m_mean[k]); } mse /= m_dimension; m_dataMSE += mse; } }
El valor medio de m_mean para cada componente se obtiene en la fase de la normalización de datos.
void CSOM::InitNormalization(const bool normalization = true) { ArrayResize(m_max, m_dimension + EXTRA_DIMENSIONS); ArrayResize(m_min, m_dimension + EXTRA_DIMENSIONS); ArrayInitialize(m_max, 0); ArrayInitialize(m_min, 0); ArrayResize(m_mean, m_dimension); ArrayResize(m_sigma, m_dimension); for(int j = 0; j < m_dimension; j++) { double maxv = -DBL_MAX; double minv = +DBL_MAX; if(normalization) { m_mean[j] = 0; m_sigma[j] = 0; } for(int i = 0; i < m_nSet; i++) { double v = m_set[m_dimension * i + j]; if(v > maxv) maxv = v; if(v < minv) minv = v; if(normalization) { m_mean[j] += v; m_sigma[j] += v * v; } } m_max[j] = maxv; m_min[j] = minv; if(normalization && m_nSet > 0) { m_mean[j] /= m_nSet; m_sigma[j] = MathSqrt(m_sigma[j] / m_nSet - m_mean[j] * m_mean[j]); } else { m_mean[j] = 0; m_sigma[j] = 1; } } }
Volviendo a los planos adicionales, hay que notar que después de los cálculos realizados en CSOMNode::RegisterPatternHit, cada neurona es capaz de devolver la estadística correspondiente a través de los métodos:
int CSOMNode::GetHitsCount() const { return m_hitCount; } double CSOMNode::GetHitsMean(const int plane) const { if(m_hitCount == 0) return 0; return m_sum[plane] / m_hitCount; } double CSOMNode::GetHitsDeviation(const int plane) const { if(m_hitCount == 0) return 0; double z = m_sumP2[plane] / m_hitCount - m_sum[plane] / m_hitCount * m_sum[plane] / m_hitCount; if(z < 0) return 0; return MathSqrt(z); } double CSOMNode::GetMSE() const { if(m_hitCount == 0) return 0; return m_mse / m_hitCount; }
De esta manera, obtenemos los datos para el llenado de dos planos, con el número de las visualizaciones de los vectores de entrada por las neuronas y con el error de cuantización.
Respuesta de la red
El próximo plano adicional es el mapa de la salida o la respuesta de la red a una determinada instancia. No hay que olvidar que después del envío de la señal a la red, juntamente con la neurona vencedora, se activan todas las demás neuronas (en mayor o en menor grado). La posibilidad de comparar la amplitud de las áreas activas de la respuesta puede ayudar a determinar la estabilidad de la solución propuesta por la red.
El cálculo de la respuesta de la red es bastante simple. Escribimos el siguiente método en la clase CSOMNode:
double CSOMNode::CalculateOutput(const double &vector[]) { m_output = CalculateDistance(vector); return m_output; }
Vamos a llamarlo para cada neurona en la clase de la red.
void CSOM::CalculateOutput(const double &vector[], const bool normalize = false) { double temp[]; ArrayCopy(temp, vector); if(normalize) Normalize(temp); m_min[DIM_OUTPUT] = DBL_MAX; m_max[DIM_OUTPUT] = -DBL_MAX; for(int i = 0; i < ArraySize(m_node); i++) { double x = m_node[i].CalculateOutput(temp); if(x < m_min[DIM_OUTPUT]) m_min[DIM_OUTPUT] = x; if(x > m_max[DIM_OUTPUT]) m_max[DIM_OUTPUT] = x; } }
Si el vector de prueba no figura en el programa, la respuesta se calcula por defecto, es decir, para el vector cero.
Agrupamiento (Clusterización)
Finalmente, el último de los planos considerados, pero probablemente sea el más importante, será el mapa de los clusters. Ordenar los datos de entrada en el mapa bidimensional es sólo la mitad del asunto. El propósito real del análisis es detectar las particularidades y clasificarlas en las clases fáciles de entender en términos de la aplicación. Cuando la dimensionalidad del espacio de las características es relativamente pequeña, podemos fácilmente distinguir las áreas con las características necesarias por las manchas de color en determinados planos, siendo que esas manchas normalmente están aisladas. Pero con la expansión de la estructura de los datos de entrada, la imagen se hace más complicada, y en vez del análisis cruzado de decenas de mapas con diferentes índices, es mucho más conveniente tener un solo mapa dividido en áreas que reivindican la atención.
El resultado del agrupamiento consistirá en marcar el mapa en áreas con características semejantes e identificar los centros de los clusters. Entonces, podemos considerarlos como las muestras más representativas (en términos estadísticos) de las clases correspondientes. Aquí, nos acercamos poco a poco a la tarea de la selección de los parámetros óptimos del EA. Pero primero, tenemos que implementar el agrupamiento.
K-Means
Existen muchos métodos de clasterización. La opción más óptima para MQL5 es usar la versión de ALGLIB que forma parte de la biblioteca estándar. Basta con incluir el archivo de cabecera:
#include <Math/Alglib/dataanalysis.mqh>
y escribir aproximadamente el siguiente método:
void CSOM::Clusterize(const int clusterNumber) { int count = m_xcells * m_ycells; CMatrixDouble xy(count, m_dimension); int info; CMatrixDouble clusters; int membership[]; double weights[]; for(int i = 0; i < count; i++) { m_node[i].GetCodeVector(weights); xy[i] = weights; } CKMeans::KMeansGenerate(xy, count, m_dimension, clusterNumber, KMEANS_RETRY_NUMBER, info, clusters, membership); Print("KMeans result: ", info); if(info == 1) // ok { for(int i = 0; i < m_xcells * m_ycells; i++) { m_node[i].SetCluster(membership[i]); } ArrayResize(m_clusters, clusterNumber * m_dimension); for(int j = 0; j < clusterNumber; j++) { for(int i = 0; i < m_dimension; i++) { m_clusters[j * m_dimension + i] = clusters[i][j]; } } } }
Realiza el agrupamiento a través del algoritmo K-Means. Lamentablemente, según sepa, es el único algoritmo del agrupamiento en la versión ALGLIB en MQL5, aunque la última versión de la biblioteca original ofrece otras, como el agrupamiento jerárquico tipo aglomerativo.
He dicho «lamentablemente» porque el algoritmo K-Means es el más «directo» en cierta medida: su esencia se reduce a la búsqueda de los centros del número establecido de los esferoides (en el espacio de las características) que cubren los puntos de la muestra de una manera más eficaz (el mínimo de la suma de los cuadrados de las distancias desde los puntos hasta los centros de los clusters). El problema es que, debido a su forma especifica, los esferoides tienen algunas limitaciones específicas en cuanto a la separabilidad de los clusters no lineales. En principio, K-Means es un caso particular del algoritmo Expectation Maximization que opera con los elipsoide de diferentes orientaciones y formas, siendo por eso más preferible. Sin embargo, incluso cuando se usa, queda la probabilidad de perdurar en el mínimo local (puesto que ambos algoritmos utilizan sólo las formas convexas y la ubicación inicial aleatoria de los centros de los clusters). Además de eso, las desventajas pueden incluir el hecho de que es necesario especificar de antemano el número de los clusters.
No obstante, vamos a considerar cómo el agrupamiento está organizado a través de K-Means en ALGLIB. El trabajo principal se ejecuta por el método CKMeans::KMeansGenerate. Le pasamos el array con los datos iniciales en un formato especial basado en objeto (CMatrixDouble xy), el número de vectores (count), la dimensionalidad del espacio de características (m_dimension) y el número deseado de los clusters (clusterNumber), el último se establece en los parámetros del programa MQL. El siguiente parámetro de entrada, KMEANS_RETRY_NUMBER, representa el número de iteraciones que se hacen por el algoritmo con diferentes centros iniciales elegidos aleatoriamente, intentando evitar una solución local. En nuestro caso, es la macro igual a 10. Como resultado del trabajo de la función, obtenemos el código de la ejecución info (diferentes valores indican en el éxito o en el error), array basado en el objeto CMatrixDouble clusters con coordenadas de los clusters, así como el array de la pertenencia de los datos de entrada a los clusters (membership).
Guardamos los centros de los clusters en el array m_clusters para marcarlos en el mapa, y coloreamos cada neurona con el color de acuerdo con la pertenencia al cluster:
m_node[i].SetCluster(membership[i]);
A la hora de trabajar con ALGLIB, tenga en cuenta que ella usa su propio generador de números aleatorios que considera el estado interno del objeto estático especial. Por eso, incluso la inicialización explícita del generador estándar a través de MathSrand no resetea sus estado. Eso es especialmente crítico para los EAs, ya que los objetos globales dentro de ellos no se generan de nuevo al modificar las configuraciones. Como resultado, en caso de ALGLIB, pueden surgir dificultades con la reproductibilidad de los resultados de los cálculos, si CMath::m_state no se pone a cero en OnInit.
Tomando en cuenta las desventajas mencionadas de K-Means, es deseable tener una opción alternativa del agrupamiento. Una solución alternativa es evidente.
Alternativa
Vamos a fijarnos de nuevo en los mapas de Kohonen, y sobre todo en las dimensionalidades adicionales que hemos introducido. U-Matrix representa un interés particular. Este plano muestra las áreas de las neuronas más próximas: es decir, las neuronas próximas no sólo en el sentido topológico del mapa 2D, sino próximas en los términos del espacio de las características. Como recordamos, las neuronas parecidas forman una especie de los «valles» en U-Matrix. Son unos candidatos perfectos para convertirse en los clasters.
Podemos transformar el mapa de las distancias unificadas en los clasters, por ejemplo, de la siguiente manera.
Copiamos la información sobre todas las neuronas al array y lo ordenamos de acuerdo con el valor de la dirección U (CSOMNode::GetDistance()) en orden ascendiente.
Para una neurona dada, vamos a verificar cíclicamente por el array si las neuronas vecinas pertenecen a algún cláster.
- Si no es así, creamos un cláster nuevo y le atribuimos la neurona actual. Obsérvese que los clasters van a crearse a partir del índice cero, lo que corresponde al cláster más «importante» puesto que él corresponde a la distancia U mínima, y luego, en orden decreciente de la importancia. Cada siguiente cláster será menos compacto en el sentido de las distancias U.
- Si entre las neuronas vecinas hay algunas marcadas por un cláster, seleccionamos la mayor de ellas, es decir, con el índice más bajo, y le atribuimos la neurona actual a este cláster.
Todo es muy simple. ¿Pero merece la pena considerar también la densidad de la población de las neuronas? Es que la distancia U tiene un soporte diferente para las neuronas con un número de penetraciones diferente. En otras palabras, si dos neuronas tienen la misma distancia U, la que tiene mostradas más instancias debe tener una ventaja ante la neurona con el número más pequeño.
Entonces, será suficiente alterar la ordenación inicial en el algoritmo descrito en orden de los valores de la fórmula CSOMNode::GetDistance() / sqrt(CSOMNode::GetHitsCount()). He añadido la raíz cuadrada para suavizar su influencia en caso de una población grande, mientras que la población pequeña debe «ser multada» de una manera más estricta.
Pero si usamos dos planos de servicio, ¿puede que tenga sentido analizar también el tercer plano, es decir, con el error de cuantización? Es verdad, cuanto más grande sea el error de cuantización en una determinada neurona, menos confianza hay que tener a la información sobre la distancia U pequeña en ella, y viceversa.
Si recordamos cómo es la función con el error de cuantización:
double CSOMNode::GetMSE() const { if(m_hitCount == 0) return 0; return m_mse / m_hitCount; }
es fácil de notar que en ella se utiliza el contador de ocurrencias m_hitCount (sólo en el denominador). Por eso, podemos reescribir la fórmula anterior para el ordenamiento del array de las neuronas como CSOMNode::GetDistance() * MathSqrt(CSOMNode::.GetMSE()), en ella van a considerarse tres índices los cuales serán añadidos a nuestra implementación de la red de Kohonen.
Casi estamos preparados para presentar un algoritmo alternativo del agrupamiento en su forma final, pero nos queda un pequeño detalle. Dentro del ciclo por el array de las neuronas, tenemos que verificar los alrededores de la neurona actual respecto a la presencia de los clusters vecinos. Antes, hemos implementado la clase de plantilla Neighbourhood para el análisis local. Ahora, vamos a crear su descendiente que se especializa en la búsqueda de los clusters.
class ClusterNeighbourhood: public Neighbourhood<int> { private: int cluster; public: ClusterNeighbourhood(const bool _hex, const int ysize): Neighbourhood(_hex, ysize) { } virtual void reset() override { cluster = -1; } virtual void iterate(const CSOMNode &node1, const CSOMNode &node2) override { int x = node2.GetCluster(); if(x > -1) { if(cluster != -1) cluster = MathMin(cluster, x); else cluster = x; } } virtual int getResult() const override { return cluster; } };
La clase contiene el número del cláster potencial «cluster» (el número es un número interno por eso paratremizamos la plantilla tipo int). Inicialmente, esta variable se inicializa en -1 en el método reset, es decir, no hay cláster. Luego, a la medida de que la clase padre llame desde su método «loop» a nuestra nueva implementación «iterate», obtenemos el número del cláster de cada neurona vecina, lo comparamos con «cluster» y guardamos el valor mínimo. Lo mismo o -1, si ningún cláster ha sido encontrado, se devuelve por el método getResult.
Como una mejora, proponemos monitorear «la altura de los picos» entre las neuronas, (es decir, el valor node1.CalculateDistance(&node2)), y ejecutar el «fluido» del número del clúster desde una neurona a otra sólo si la «altura» es menor que antes. La versión final de la implementación se presenta en el código fuente.
Por fin, podemos implementar el agrupamiento alternativo.
void CSOM::Clusterize() { double array[][2]; int n = m_xcells * m_ycells; ArrayResize(array, n); for(int i = 0; i < n; i++) { if(m_node[i].GetHitsCount() > 0) { array[i][0] = m_node[i].GetDistance() * MathSqrt(m_node[i].GetMSE()); } else { array[i][0] = DBL_MAX; } array[i][1] = i; m_node[i].SetCluster(-1); } ArraySort(array); ClusterNeighbourhood clnh(m_hexCells, m_ycells); int count = 0; // number of clusters ArrayResize(m_clusters, 0); for(int i = 0; i < n; i++) { // skip if already assigned if(m_node[(int)array[i][1]].GetCluster() > -1) continue; // check if current node is adjusent to any existing cluster int r = clnh.loop((int)array[i][1], m_node); if(r > -1) // a neighbour belongs to a cluster already { m_node[(int)array[i][1]].SetCluster(r); } else // we need new cluster { ArrayResize(m_clusters, (count + 1) * m_dimension); double vector[]; m_node[(int)array[i][1]].GetCodeVector(vector); ArrayCopy(m_clusters, vector, count * m_dimension, 0, m_dimension); m_node[(int)array[i][1]].SetCluster(count++); } } }
El algoritmo sigue prácticamente todo el pseudocódigo verbal descrito anteriormente: llenamos el array bidimensional (el valor de la fórmula en la primera dimensión, y el índice de la neurona en la segunda), ordenamos, recorremos cíclicamente todas las neuronas y analizamos los alrededores para cada una de ellas.
Desde luego, la calidad del agrupamiento tiene que ser evaluada en la práctica, y yo supongo la presencia de los problemas topológicos desde el principio. Sin embargo, considerando que la mayoría de los modos clásicos del agrupamiento también tienen sus problemas y ceden ante el modo propuesto en la sencillez, la nueva solución parece bastante atractiva.
Entre las ventajas de esta implementación, yo mencionaría que los clásteres están ordenados por la relevancia (en K-Means mencionado antes, los clasteres son del mismo valor), su forma es aleatoria y no hace falta establecer su número de antemano. Cabe mencionar que la última ventaja tiene su reverso, es decir, el número de los clusters puede ser bastante grande. Al mismo tiempo, el ordenamiento de los clusters por el grado de la semejanza del contenido y por el error mínimo permite considerar en la práctica sólo 5-10 primeros clusters, dejando el resto al margen.
Puesto que no he encontrado el método parecido del agrupamiento en las fuentes abiertas, yo propongo llamarlo el agrupamiento de Korotky (o de forma más larga pero simple, short-path clusterization) a base de U-Matrix y el error de cuantización (QE).
Adelantándome, diré que después de muchas pruebas, ha sido confirmado que los centros de los clusters encontrados por el algoritmo K-Means ofrecen los resultados peores que el agrupamiento alternativo (por lo menos, en la tarea del análisis de los resultados de la optimización). Debido a eso, siempre va a entenderse y aplicarse solamente este método del agrupamiento a continuación.
Simulación
Es la hora de pasar de la teoría a la práctica y comprobar el funcionamiento de la red. Vamos a crear un EA simple y universal con posibilidades de demostrar la funcionalidad principal. Lo llamaremos SOM-Explorer.
Incluiremos los archivos de cabecera con las clases consideradas anteriormente. Definimos los parámetros de entrada.
Grupo — Network Structure and Data Settings
- DataFileName — el nombre del archivo de texto con los datos para el aprendizaje o la simulación; la clase CSOM soporta el formato csv, pero añadiremos un poco más tarde la lectura de los archivos set, porque el análisis de las configuraciones de optimización de otros EAs «están en el juego»; cuando se especifica el archivo con los datos de entrada, su nombre se usa también para el guardado de la red tras su aprendizaje pero con otra extensión (véase más abajo); se puede indicar o no la extensión csv; el nombre puede incluir una carpeta dentro de MQL5/Files;
- NetFileName — el nombre de un archivo binario de su propio formato con la extensión som; la clase CSOM es capaz de guardar y leer las redes en/desde los archivos de este tipo; si necesita modificar la estructura de los datos guardados, cambie el nombre de la versión en la signatura que se escribe al principio del archivo; si NetFileName está vacío, el EA trabaja en modo de aprendizaje, y si la red está especificada, trabaja en el modo de simulación (visualización de los datos de entrada en la red preparada); se puede especificar o no la extensión som; el nombre puede incluir una carpeta dentro de MQL5/Files;
- si ambos parámetros DataFileName y NetFileName están vacíos, el EA va a generar un conjunto de demostración de datos aleatorios 3D y realizar el aprendizaje a su base;
- si el nombre de la red en NetFileName es correcto, se puede indicar el nombre de un archivo inexistente en el parámetro DataFileName, por ejemplo, el símbolo '?', y como resultado, el EA generará una incidencia aleatoria de los datos de prueba para el intervalo de definición que se guarda en el archivo de la red (obsérvese que esta información es necesaria para que la red enseñada normalice correctamente los datos desconocidos en el modo de servicio; el envío de los valores de otro intervalo de definición a la entrada de la red, desde luego, no provocará ningún fallo, pero los resultados serán incorrectos; por ejemplo, es difícil de esperar un funcionamiento correcto de la red, si se le pasa un valor negativo de la reducción (drawdone) o del número de transacciones);
- CellsX — tamaño de la cuadrícula por la horizontal (número de neuronas), por defecto es 10;
- CellsY — tamaño de la cuadrícula por la vertical (número de neuronas), por defecto es 10;
- HexagonalCell — característica del uso de la cuadrícula hexagonal, por defecto es true; ponga false para una cuadrícula rectangular;
- UseNormalization — activar/desactivar la normalización de los datos de entrada, por defecto es true, siendo recomendado no desactivar;
- EpochNumber — número de las épocas del aprendizaje, por defecto es 100;
- ValidationSetPercent — tamaño de la muestra de validación en por cientos del número total de los datos de entradas, por defecto es 0, es decir, la validación está desactivada; en caso de usarlo, los valores recomendados están cerca de 10;
- ClusterNumber — número de los clusters; por defecto, es 1, lo que indica en nuestro agrupamiento adaptable; el valor 0 desactiva el agrupamiento; el valor más de 0 inicia el agrupamiento a través del método K-Means; el agrupamiento se ejecuta inmediatamente después del aprendizaje, los clusters se guardan en el archivo de la red;
Grupo - Visualization
- ImageW — tamaño de cada mapa (plano) en píxeles por la horizontal, por defecto — 500;
- ImageH — tamaño de cada mapa (plano) en píxeles por la vertical, por defecto — 500;
- MaxPictures — número de mapas en una línea, por defecto es 0, lo que significa la visualización de los mapas en una línea continua con posibilidad del desplazamiento (se permiten las imágenes grandes); si MaxPictures es más de 0, el conjunto entero de los planos se muestra en varias líneas, en cada una de las cuales hay MaxPictures de los mapas (es cómodo para observar todos los mapas en una pequeña escala);
- ShowBorders — activar/desactivar el dibujado de los bordes entre las neuronas, por defecto, es false;
- ShowTitles — activar/desactivar la visualización de los textos con las características de las neuronas, por defecto, es true;
- ColorScheme — selección de una de los 4 esquemas de color, por defecto, es Blue_Green_Red (máximo de colores);
- ShowProgress — activar/desactivar la actualización dinámica de la visualización de las imágenes de la red durante el aprendizaje; se realiza cada segundo, por defecto es true;
Grupo - Options
- RandomSeed — número entero para inicializar el generador de números aleatorios, por defecto es 0;
- SaveImages — opción del guardado de las imágenes de la red después de la conclusión; se puede usarla después del aprendizaje y después del inicio de prueba; por defecto es false;
Solamente se trata de las configuraciones básicas. Vamos a añadir otra parte de los parámetros específicos a la medida de que continuemos resolviendo los problemas.
¡Atención! El Asesor Experto altera los ajustes del gráfico actual— abra el gráfico nuevo seleccionado sólo para trabajar con este EA.
El objeto de la clase CSOMDisplay ejecutará todo el trabajo en el EA.
CSOMDisplay KohonenMap;
Durante la inicialización, no olvidemos altivar el procesamiento de eventos del desplazamiento del ratón; la clase los usa para visualizar las ayudas emergentes y el scrolling.
void OnInit() { ChartSetInteger(0, CHART_EVENT_MOUSE_MOVE, true); EventSetMillisecondTimer(1); } void OnChartEvent(const int id, const long &lparam, const double &dparam, const string &sparam) { KohonenMap.OnChartEvent(id, lparam, dparam, sparam); }
Los algoritmos de redes neuronales (aprendizaje o simulación) se inician en el EA una vez: por el temporizador, y el temporizador luego se desactiva.
void OnTimer() { EventKillTimer(); MathSrand(RandomSeed); bool hasOneTestPattern = false; if(NetFileName != "") { if(!KohonenMap.Load(NetFileName)) return; KohonenMap.DisplayInit(ImageW, ImageH, MaxPictures, ColorScheme, ShowBorders, ShowTitles); Comment("Map ", NetFileName, " is loaded; size: ", KohonenMap.GetWidth(), "*", KohonenMap.GetHeight(), "; features: ", KohonenMap.GetFeatureCount());
Si se indica el archivo preparado con la red, lo cargamos y preparamos la pantalla de acuerdo con ajustes visuales.
if(DataFileName != "") { if(!KohonenMap.LoadPatterns(DataFileName)) { Print("Data loading error, file: ", DataFileName); // generate a random test vector int n = KohonenMap.GetFeatureCount(); double min, max; double v[]; ArrayResize(v, n); for(int i = 0; i < n; i++) { KohonenMap.GetFeatureBounds(i, min, max); v[i] = (max - min) * rand() / 32767 + min; } KohonenMap.AddPattern(v, "RANDOM"); Print("Random Input:"); ArrayPrint(v); double y[]; CSOMNode *node = KohonenMap.GetBestMatchingFeatures(v, y); Print("Matched Node Output (", node.GetX(), ",", node.GetY(), "); Hits:", node.GetHitsCount(), "; Error:", node.GetMSE(),"; Cluster N", node.GetCluster(), ":"); ArrayPrint(y); KohonenMap.CalculateOutput(v, true); hasOneTestPattern = true; } }
Si se indica el archivo con datos de texto, tratamos de cargarlo. Si no conseguimos hacerlo, mostramos el mensaje en el registro y generamos una muestra aleatoria de datos de texto «v». El número de características (dimensionalidad de vectores) y sus intervalos permitidos se determinan a través de los métodos GetFeatureCount y GetFeatureBounds. Luego, usando la llamada a AddPattern, la instancia se añade al conjunto de servicio de datos con el nombre "RANDOM".
Este método conviene para formar las muestras de aprendizaje desde las fuentes de los datos con formatos no soportados, por ejemplo, desde las bases de datos, así como para el llenado directamente desde los indicadores. En principio, en este caso, la adición de la instancia en el conjunto de servicio se requiere solamente para su visualización posterior en el mapa (se muestra más abajo), y para definir la neurona de la red más conveniente basta con llamar a GetBestMatchingFeatures. Este método, entre varios métodos GetBestMatchingXYZ disponibles, permite que el array «y» obtenga los valores correspondientes de las instancias de la neurona vencedora. Finalmente, usando CalculateOutput, mostramos la reacción de la red a la instancia de texto en un plano adicional.
Pues, continuamos con el código del EA.
} else // a net file is not provided, so training is assumed { if(DataFileName == "") { // generate 3-d demo vectors with unscaled values {[0,+1000], [0,+1], [-1,+1]} // feed them to the net to compare results with and without normalization // NB. titles should be valid filenames for BMP string titles[] = {"R1000", "R1", "R2"}; KohonenMap.AssignFeatureTitles(titles); double x[3]; for(int i = 0; i < 1000; i++) { x[0] = 1000.0 * rand() / 32767; x[1] = 1.0 * rand() / 32767; x[2] = -2.0 * rand() / 32767 + 1.0; KohonenMap.AddPattern(x, StringFormat("%f %f %f", x[0], x[1], x[2])); } }
Si la red enseñada no está definida, suponemos el modo de aprendizaje. Comprobamos si hay parámetros de entrada. Si no es así, generamos un conjunto aleatorio de los vectores tridimensionales en el que el primer componente se encuentra en el rango [0,+1000], el segundo está dentro de [0,+1], y el tercer está dentro de [-1,+1]. El nombre de los componentes se pasan a la red a través de AssignFeatureTitles, y los datos se pasan a través de AddPattern.
else // a data file is provided { if(!KohonenMap.LoadPatterns(DataFileName)) { Print("Data loading error, file: ", DataFileName); return; } }
Si los datos de entrada vienen desde un archivo, cargamos este archivo. Si surge un error, terminamos el trabajo, porque no hay ninguna red ni los datos.
Luego, ejecutamos el aprendizaje y el agrupamiento.
KohonenMap.Init(CellsX, CellsY, ImageW, ImageH, MaxPictures, ColorScheme, HexagonalCell, ShowBorders, ShowTitles); if(ValidationSetPercent > 0 && ValidationSetPercent < 50) { KohonenMap.SetValidationSection((int)(KohonenMap.GetDataCount() * (1.0 - ValidationSetPercent / 100.0))); } KohonenMap.Train(EpochNumber, UseNormalization, ShowProgress); if(ClusterNumber > 1) { KohonenMap.Clusterize(ClusterNumber); } else { KohonenMap.Clusterize(); } }
Si el análisis de una determinada instancia de texto no ha sido establecida (particularmente, inmediatamente después del aprendizaje), por defecto, formamos la respuesta de la red para el vector con ceros.
if(!hasOneTestPattern) { double vector[]; ArrayResize(vector, KohonenMap.GetFeatureCount()); ArrayInitialize(vector, 0); KohonenMap.CalculateOutput(vector); }
A continuación, dibujamos todos los mapas en los búferes internos de los recursos gráficos (primero, la capa de color de atrás):
KohonenMap.Render(); // draw maps into internal BMP buffers
luego, las leyendas:
if(hasOneTestPattern) KohonenMap.ShowAllPatterns(); else KohonenMap.ShowAllNodes(); // draw labels in cells in BMP buffers
Marcamos los clusters:
if(ClusterNumber != 0) { KohonenMap.ShowClusters(); // mark clusters }
Mostramos los búferes en el gráfico y guardamos opcionalmente las imágenes en el archivo:
KohonenMap.ShowBMP(SaveImages); // display files as bitmap images on chart, optionally save into files
Los archivos se colocan en una carpeta separada con el mismo nombre que el archivo de la red (si está especificado) o el archivo con datos (si está especificado). Si el archivo con datos no ha sido especificado y la red se enseñaba en los datos generados aleatorios, el nombre para el archivo som y de la carpeta con imágenes se forma usando el prefijo SOM y la fecha y la hora actual.
Finalmente, guardamos la red enseñada en el archivo. Si el nombre del archivo ya ha sido especificado en NetFileName, entonces el EA trabajaba en el modo de simulación, y no es necesario guardar la red de nuevo.
if(NetFileName == "") { KohonenMap.Save(KohonenMap.GetID()); } }
Vamos a intentar iniciar el EA con la generación de los datos de texto aleatorios. Con todas las configuraciones predefinidas, excepto los tamaños reducidos de las imágenes, para que todos los planos entren en la «captura de la pantalla» - ImageW = 230, ImageH = 230, MaxPictures = 3, - obtenemos lo siguiente:
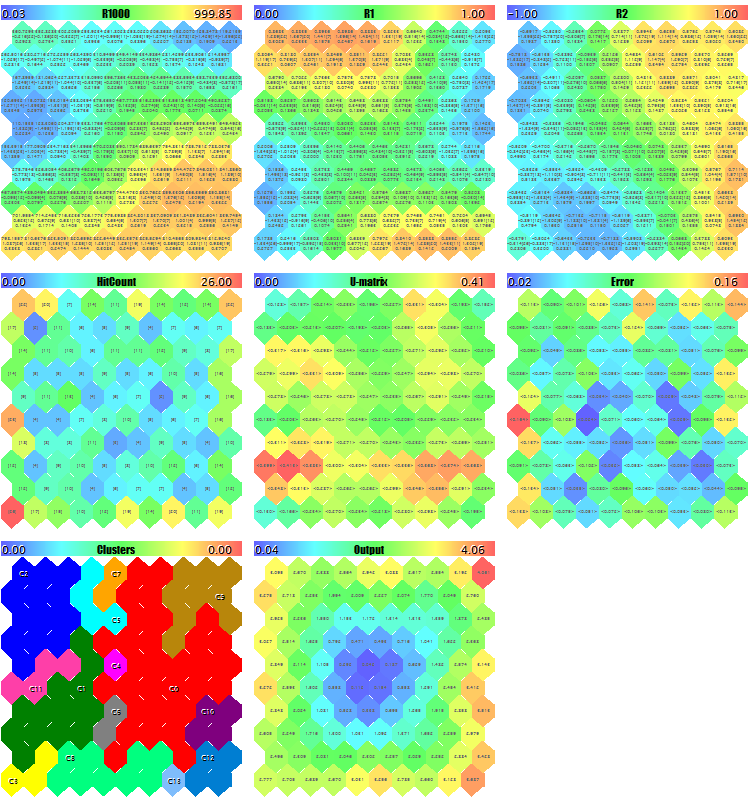
Ejemplo de los mapas de Kohonen para los vectores 3D aleatorios
Aquí, en cada neurona se muestra la información de servicio (se puede ver los detalles apuntando con el cursor) y los clusters encontrados están marcados.
En el registro, se muestra aproximadamente la siguiente información (la información por los clusters está limitada con cinco, se puede cambiar en el código):
Pass 0 from 1000 0% Pass 78 from 1000 7% Pass 157 from 1000 15% Pass 232 from 1000 23% Pass 310 from 1000 31% Pass 389 from 1000 38% Pass 468 from 1000 46% Pass 550 from 1000 55% Pass 631 from 1000 63% Pass 710 from 1000 71% Pass 790 from 1000 79% Pass 870 from 1000 87% Pass 951 from 1000 95% Overall NMSE=0.09420336270396877 Training completed at pass 1000, NMSE=0.09420336270396877 Clusters [14]: "R1000" "R1" "R2" N0 754.83131 0.36778 0.25369 N1 341.39665 0.41402 -0.26702 N2 360.72925 0.86826 -0.69173 N3 798.15569 0.17846 -0.37911 N4 470.30648 0.52326 0.06442 Map file SOM-20181205-134437.som saved
Si ahora especificamos el nombre del archivo creado SOM-20181205-134437.som con la red en el parámetro NetFileName, y en el parámetro DataFileName indicamos '?', obtenemos el resultado de la ejecución de prueba para una muestra aleatoria que no sea del conjunto de aprendizaje. Para ver mejor los mapas, establecemos los tamaños más grandes y MaxPictures en 0.

Los mapas de Kohonen para dos primeros componentes de los vectores 3D aleatorios
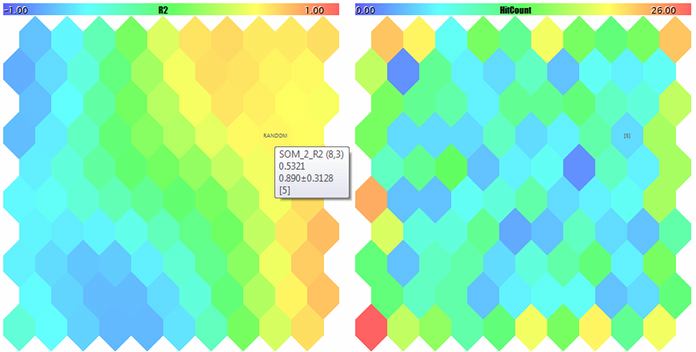
Mapa de Kohonen para el tercer componente de los vectores 3D aleatorios y el contador de ocurrencias
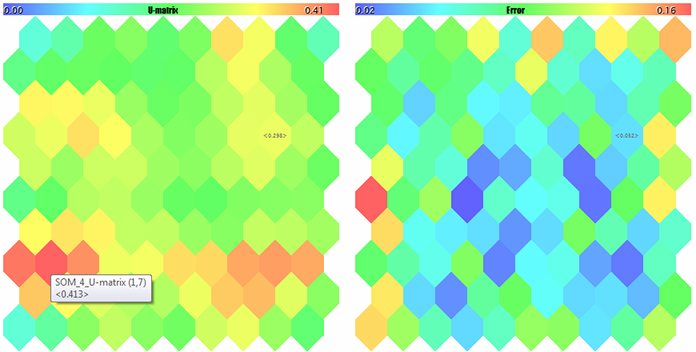
U-Matrix y error de cuantización
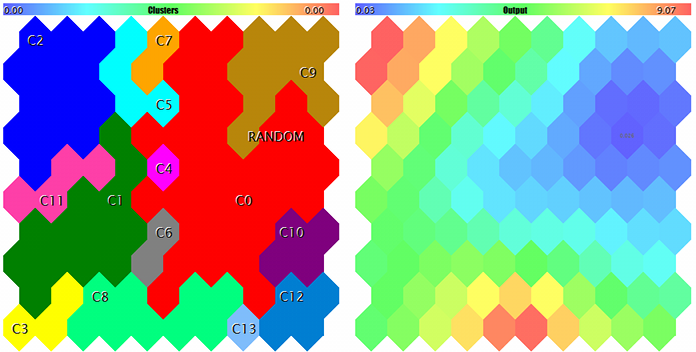
Clusters y respuesta de la red de Kohonen a la muestra de prueba
La muestra está marcada con RANDOM. Las ayudas sobre las neuronas aparecen al apuntar con el ratón. En el registro, se muestra aproximadamente lo siguiente:
FileOpen error ?.csv : 5004 Data loading error, file: ? Random Input: 457.17510 0.29727 0.57621 Matched Node Output (8,3); Hits:5; Error:0.05246704285146882; Cluster N0: 497.20453 0.28675 0.53213
Pues bien, las herramientas para trabajar con la red de Kohonen están listas. Podemos proceder a las tareas aplicadas. Lo haremos en el segundo artículo.
Conclusión
Las implementaciones de las redes de Kohonen están disponibles para los usuarios de MetaTrader ya algunos años. Hemos corregido algunos errores, las hemos completado con unas herramientas útiles y hemos testeado su funcionamiento a través de un EA especial de demostración. Los códigos fuente permiten aplicar las clases para propias tareas. Los ejemplos de eso serán considerados a continuación.
Traducción del ruso hecha por MetaQuotes Ltd.
Artículo original: https://www.mql5.com/ru/articles/5472
Advertencia: todos los derechos de estos materiales pertenecen a MetaQuotes Ltd. Queda totalmente prohibido el copiado total o parcial.
Este artículo ha sido escrito por un usuario del sitio web y refleja su punto de vista personal. MetaQuotes Ltd. no se responsabiliza de la exactitud de la información ofrecida, ni de las posibles consecuencias del uso de las soluciones, estrategias o recomendaciones descritas.
 Utilidad para la selección y navegación en MQL5 y MQL4: añadiendo las pestañas de "recordatorios" y guardando objetos gráficos
Utilidad para la selección y navegación en MQL5 y MQL4: añadiendo las pestañas de "recordatorios" y guardando objetos gráficos
 Aplicando el método de Montecarlo al aprendizaje por refuerzo
Aplicando el método de Montecarlo al aprendizaje por refuerzo
 Analizando resultados comerciales con la ayuda de informes HTML
Analizando resultados comerciales con la ayuda de informes HTML
 Optimización separada de una estrategia en condiciones de tendencia y flat
Optimización separada de una estrategia en condiciones de tendencia y flat
- Aplicaciones de trading gratuitas
- 8 000+ señales para copiar
- Noticias económicas para analizar los mercados financieros
Usted acepta la política del sitio web y las condiciones de uso
Digamos que hay tres parámetros de entrada que se han optimizado. Kohonen ha realizado la agrupación y la visualización. ¿Cuál es la conveniencia de encontrar los parámetros óptimos?
No entiendo en absoluto lo de la previsión. Si es posible, también en pocas palabras la idea.
Normalmente la optimización da una gran variedad de opciones y no da estimaciones de su estabilidad. El segundo artículo intenta resolver estos problemas en parte visualmente, en parte algorítmicamente con la ayuda de los mapas de Kohonen. El artículo ha sido enviado para su verificación.
Introducimos algunos parámetros de entrada adicionales en el Asesor Experto. Y evaluamos su dependencia de otros.
Kohonen nos permite hacer esto visualmente - en forma de mapa. Si estamos hablando de un solo dígito, entonces esto es para otros métodos.
Me interesa más la parte práctica de usar clustering en forex, aparte de hacer tablas de correlación y demás. Además no entiendo el tema de la estabilidad de los mapas sobre nuevos datos, cuales son las formas de estimar la capacidad de generalización y cuanto se reentrenan
Al menos en teoría para entender como se puede utilizar esto de forma efectiva. Lo único que se me ocurre es dividir la serie temporal en varios "estados"
Quizá la parte 2 ofrezca algunas respuestas. Si se preserva la ley de distribución de los datos, debería haber estabilidad. Para controlar la generalizabilidad, se sugiere elegir el tamaño del mapa y/o la duración del entrenamiento mediante un muestreo de validación.
Algunos ejemplos de casos de uso se encuentran en la segunda parte.
Quizá la segunda parte ofrezca algunas respuestas. Si se mantiene la ley de distribución de los datos, debería haber estabilidad. Para controlar la generalizabilidad, se sugiere elegir el tamaño del mapa y/o la duración del entrenamiento mediante un muestreo de validación.
Algunos ejemplos de casos de uso se encuentran en la segunda parte.
sí, lo siento, no había visto que ya existe un método de detención mediante muestreo de validación. Entonces esperemos ejemplos, interesante :)