
遺伝的アルゴリズム - とても簡単です!
はじめに
遺伝的アルゴリズム (GA) は、ヒューリスティックアルゴリズム (EA)を指し、多くのケースにおいて問題に対する適切な解法を提示してくれます。ただ、その意思決定の正しさは数学的に証明されておらず、分析的なソリューションの発見が難しい、もしくは不可能な問題に対して頻繁に用いられます。
このクラス(NP)における問題の古典的な一つの例は、「旅行するセールスマンの問題(travelling salesman problem)」です。(有名な組み合わせ最適化問題の一つです)困難な点は、少なくとも一回特定の都市を通り、最初の地点である都市に戻る最も効果的なルートを探索するという点です。しかし、形式化に陥っているタスクに使用することは避けられません。
EAは、かなりの時間を要する、全てのオプションを探索するという形の代わりに、高く計算的な複雑性を持ち合わせた問題の解決に幅広く用いられます。ウイルス対策ソフト、コンピュータゲーム、エンジニアリング、などその他のエリアにおいて、パターン認識など人工知能の領域にて使用されます。
これは、MetaQuotes Software Corpにて紹介されています。MetaTrader4/5のソフトウェア製品にてGAを使用しましょう。ストラテジーテスターについて、また内蔵のストラテジー最適化を使用することでどれだけの時間や労力を省くことができるかについてはすでにご存知だと思います。直接計数などにより、GAを用いて最適化を行うことができます。さらに、MetaTrader5テスターでは、ユーザーの最適化基準を設定することができます。おそらく、読者の皆さんは、 直接計数と比較した際の、EAから提供されている GA とその利点についての記事に興味がおありだと思います。
1. 歴史
1年以上前に、ニューラルネットワークの練習のために、最適化アルゴリズムが必要でした。様々なアルゴリズムを調べた後、GAを選択するに至りました。すでにできあがっている実装方法を検索した結果、一つは、公的なアクセスを許しており、もう一方は、最適化できる変数の数など、機能的制限があり、「ほんの少しの調整」が可能でした。
全種類のニュートラルネットワークを練習するだけではなく、一般的ないかなる最適化の問題を解決する上で柔軟な手段が必要でした。外国の「遺伝的生成」の長期間の学習の後でも、いかに作動するのか未だ理解することができませんでした。この理由は、洗練されたコードの記述方式か、プログラミング経験の欠如、もしくは両方が起因していました。
主に困難だった点は、遺伝子をバイナリコードに書き換え、それらを扱うという部分から生じています。将来における拡張性や容易な修復に焦点を当てつつ、遺伝的アルゴリズムを1から作成することに決定しました。
また、バイナリ形式の変換を扱いたくなく、直接、遺伝子を扱うことに決定しています。つまり、実数にて、遺伝子の染色体を表現するということを意味します。これが、実数による染色体の表現を行う遺伝的アルゴリズムのコードが生まれた経緯です。その後、何も新しいことを発見できていないことに気づき、類似した遺伝的アルゴリズム(real-coded GAと呼ばれています)が15年以上も前から存在していることを知りました。
実践的な問題における使用の個人的経験に基づいており、読者の皆さまが理解するためのGAの機能原則や実行へのアプローチに関するビジョンを記しています。
2. GAの詳細
GAは自然の原則に基づく規則を備えています。これらは、遺伝性と変異性の原則です。遺伝性は、有機体が修正や進化により培った特徴を子孫に受け継ぐ能力です。この能力により、全ての生物は種の特性を末代に受け継いでいます。
生物の遺伝子の変異性は、遺伝的多様性を保証し、(天候の変化、食料の増減、天敵の出現など)将来の状況に最も適する特徴が何かを前もって知ることができないので、変異結果はランダムになっています。この変異性は、新しい特徴を持った生物を生み、生息地の変化に対応し生き残る子孫を残していくことができます。
生物学では、突然変異により生まれる変異性は、突然変異と呼ばれ、交配により遺伝の組み合わせによる変異は、組み合わせ変異と呼ばれます。これら両方のタイプがGAにて実装されています。さらに、DNAのヌクレチオイドの変異の、自然的(自発的)、人工的(誘発的)な変異の仕組みを模した突然変異生成の実行があります。
アルゴリズムの規範に則った情報の伝達の最小の構成単位は遺伝子です。遺伝子は、特定の習慣や特徴の生成を操作する遺伝性の構造的、機能的単位です。機能の一つの変数を遺伝子と呼ぶこととします。遺伝子は、実数にて表現されています。上記の機能を持つ遺伝子の変数は 染色体の特徴です。
縦の一列が染色体を表していることを確認してください。f(x) = x ^ 2の関数における染色体はこのようになります。
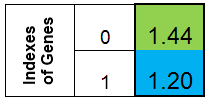
図1 f (x) = x ^ 2の関数の染色体
f(x)関数のインデックスが0の値である時点で、個々の適応が行われます。(この機能をフィットネスファンクションFF、関数の値を VFFと呼びます。)一次元の配列に染色体を保持するのに便利です。これは二重染色体[] 配列です。
同じ進化の時代の種は、 同じ種類に組み込まれます。さらに、その個体群は、任意で二つの同等の集団に分けられます。親と、子孫の二つのグループです。全種類の中から選択された親の集団の交配の結果、その種の子孫集団を取り替える、その種の半分ほどに匹敵する量の新しい子孫集団が生まれます。
f(x)=x^2の最小関数の検索における、その個体の全体の量はこのような形になります。
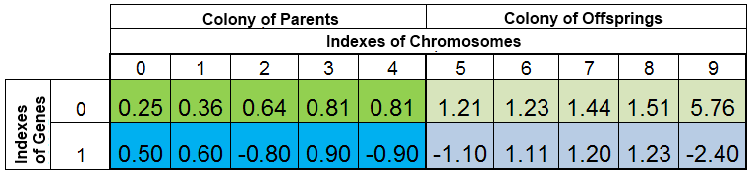
図 2個体の全種
VFFにより、その種は分類されます。ここで、染色体の0番目のインデックスは、最小のVFFを持つ試験体が占めます。その新しい子孫集団は、親集団が何もされず残る一方、子孫集団の種を完全に取り替えます。しかしながら、親集団は必ずしも完全に残るわけではありません。複製された種が破壊され、新しい市子孫の種が親集団の空きを埋め合わせます。そして残りの種は、子孫集団の場所に置かれます。
言い換えれば、種の量は、一定ではなく、自然現象と同じく、時代ごとに変化しています。例えば、繁殖前の種と繁殖後の種はこのようになります。
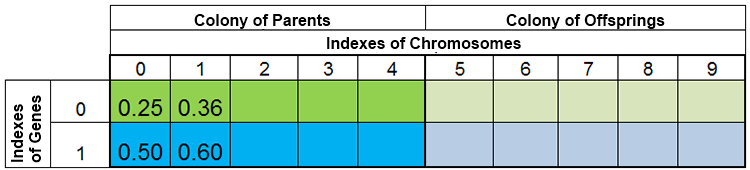
図 3. 繁殖前の種
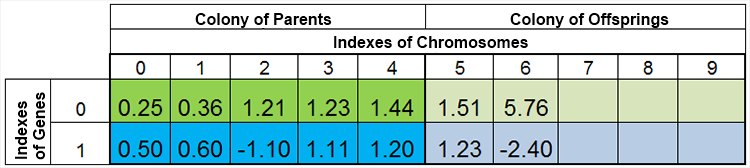
図4繁殖後の種
複製の破壊と個体間での交配繁殖の禁止と同様に、紹介されている子孫集団による種の半分の置き換えの仕組みは、「ボトルネック効果」(一つの種の絶滅へとつながる様々な理由による大幅な種の遺伝子プールの減少を意味する生物学の用語であり、GAはある特定の極値における「詰まり」、ある染色体の終了に直面するということを指す)を避けるというただ一つのゴールを目指しています。
3. UGAlib機能の紹介
GAアルゴリズム:
- プロトタイプの種の作成ある特定の範囲においてランダムに遺伝子が生成されます。
- 個々の適合性の決定、FFの計算
- 染色体の複製の除去後の生殖準備
- (最適なVFFを持つ)染色体の隔離と保護
- UGAの管理(選択、交配、変異)それぞれの交配、変異において、新しい親の種が選択されます。次の時代への種の準備
- 基準染色体を持つ遺伝子の最適な子孫の遺伝子の比較もしその子孫の染色体が、基準の染色体よりもよければ、基準の染色体と取り替えてください。
一定の時期に出現する基準の染色体よりも良い染色体がなくなるまでパラグラフ5から続けてください。
3.1. グローバル変数グローバル変数
グローバルレベルでの以下の変数の宣言:
//----------------------Global variables----------------------------- double Chromosome[]; //A set of optimized arguments of the function - genes //(for example: the weight of the neuron network, etc.)- of the chromosome int ChromosomeCount =0; //Maximum possible amount of chromosomes in a colony int TotalOfChromosomesInHistory=0;//Total number of chromosomes in the history int ChrCountInHistory =0; //Number of unique chromosomes in the base chromosome int GeneCount =0; //Number of genes in the chromosome double RangeMinimum =0.0;//Minimum search range double RangeMaximum =0.0;//Maximum search range double Precision =0.0;//Search step int OptimizeMethod =0; //1-minimum, any other - maximum int FFNormalizeDigits =0; //Number of symbols after the comma in the fitness value int GeneNormalizeDigits =0; //Number of symbols after the comma in the genes value double Population [][1000]; //Population double Colony [][500]; //Offspring colony int PopulChromosCount =0; //The current number of chromosomes in a population int Epoch =0; //Number of epochs without progress int AmountStartsFF=0; //Number of launches of the fitness function //————————————————————————————————————————————————————————————————————————
3.2. UGAGAの主要機能
実は、プログラムから呼ばれるGAのメインの機能は上述されており、それについて詳しくは述べません。
アルゴリズムが完成しだい、ログに以下の情報を記録します。
- 全体でいくつの時代(世代)があったか
- 全ての欠陥数
- 特有の染色体数
- FFの起動回数
- 歴史上の全染色体数
- 歴史上の全染色体数と、複製の割合
- 最良の結果
「特有の染色体数」と「FFの起動回数」は同じ数であるが、異なる計算方法である。コントールの使用
//———————————————————————————————————————————————————————————————————————— //Basic function UGA void UGA ( double ReplicationPortion, // Proportion of replication. double NMutationPortion, // Proportion of natural mutations. double ArtificialMutation, // Proportion of artificial mutations. double GenoMergingPortion, // Proportion of borrowed genes. double CrossingOverPortion, // Proportion of cross -over. //--- double ReplicationOffset, // Coefficient of displacement of interval borders double NMutationProbability // Probability of mutation of each gene in% ) { //generator reset takes place only once MathSrand((int)TimeLocal()); //-----------------------Variables------------------------------------- int chromos=0, gene =0;//indexes of chromosomes and genes int resetCounterFF =0;//counter of resets of "Epochs without progress" int currentEpoch =1;//number of the current epoch int SumOfCurrentEpoch=0;//sum of "Epochs without progress" int MinOfCurrentEpoch=Epoch;//minimum of "epochs without progress" int MaxOfCurrentEpoch=0;//maximum of "Epochs without progress" int epochGlob =0;//total number of epochs // Colony [number of traits(genes)][number of individuals in a colony] ArrayResize(Population,GeneCount+1); ArrayInitialize(Population,0.0); // Colony of offspring [number of traits(genes)][number of individuals in a colony] ArrayResize(Colony,GeneCount+1); ArrayInitialize(Colony,0.0); // Chromosome bank // [number of traits (genes)][number of chromosomes in the bank] double historyHromosomes[][100000]; ArrayResize(historyHromosomes,GeneCount+1); ArrayInitialize(historyHromosomes,0.0); //---------------------------------------------------------------------- //--------------Verification of the correctness of incoming parameters---------------- //...the number of chromosomes mus be less than 2 if(ChromosomeCount<=1) ChromosomeCount=2; if(ChromosomeCount>500) ChromosomeCount=500; //---------------------------------------------------------------------- //====================================================================== // 1) Create a proto- population —————1) ProtopopulationBuilding(); //====================================================================== // 2) Determine the fitness of each individual —————2) //For the 1st colony for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=0;chromos<ChromosomeCount;chromos++) Population[0][chromos]=Colony[0][chromos]; //For the 2nd colony for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos-ChromosomeCount]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) Population[0][chromos]=Colony[0][chromos-ChromosomeCount]; //====================================================================== // 3) Prepare the population for reproduction ————3) RemovalDuplicates(); //====================================================================== // 4) Extract the reference chromosome —————4) for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; //====================================================================== ServiceFunction(); //The main cycle The main cycle of the genetic algorithm from 5 to 6 while(currentEpoch<=Epoch) { //==================================================================== // 5) Operators of UGA —————5) CycleOfOperators ( historyHromosomes, //--- ReplicationPortion, //Proportion of replication. NMutationPortion, //Proportion of natural mutation. ArtificialMutation, //Proportion of artificial mutation. GenoMergingPortion, //Proportion of borrowed genes. CrossingOverPortion,//Proportion of cross- over. //--- ReplicationOffset, //Coefficient of displacement of interval borders NMutationProbability//Probability of mutation of each gene in % ); //==================================================================== // 6) Compare the genes of the best offspring with the genes of the reference chromosome. // If the chromosome of the best offspring is better that the reference chromosome, // replace the reference. —————6) //If the optimization mode is - minimization if(OptimizeMethod==1) { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]<Chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Rest the counter of "epochs without progress" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //If the optimization mode is - minimization else { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]>Chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Reset the counter of "epochs without progress" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //==================================================================== //Another epoch went by.... epochGlob++; } Print("Epochs went by=",epochGlob," Number of resets=",resetCounterFF); Print("MinOfCurrentEpoch",MinOfCurrentEpoch, " AverageOfCurrentEpoch",NormalizeDouble((double)SumOfCurrentEpoch/(double)resetCounterFF,2), " MaxOfCurrentEpoch",MaxOfCurrentEpoch); Print(ChrCountInHistory," - Unique chromosome"); Print(AmountStartsFF," - Total number of launches of FF"); Print(TotalOfChromosomesInHistory," - Total number of chromosomes in the history"); Print(NormalizeDouble(100.0-((double)ChrCountInHistory*100.0/(double)TotalOfChromosomesInHistory),2),"% of duplicates"); Print(Chromosome[0]," - best result"); } //————————————————————————————————————————————————————————————————————————
3.3. プロト種の生成プロトタイプの種の作成
多くの最適化の問題において、関数の引数がどの行にあるのかを前もって知る方法はないため、最善の選択肢は、特定の領域におけるランダムな生成です。
//———————————————————————————————————————————————————————————————————————— //Creating a proto- population void ProtopopulationBuilding() { PopulChromosCount=ChromosomeCount*2; //Fill up the population with chromosomes with random //...genes in the range between RangeMinimum...RangeMaximum for(int chromos=0;chromos<PopulChromosCount;chromos++) { //beginning with the 1st indexes (the 0 index is reserved for VFF) for(int gene=1;gene<=GeneCount;gene++) Population[gene][chromos]= NormalizeDouble(SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); TotalOfChromosomesInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.4. GetFitness適合性の取得
順番に個々の染色体における最適な関数を実行する
//------------------------------------------------ ------------------------ // Getting the fitness for each individual. void GetFitness ( double &historyHromosomes[][100000] ) { for(int chromos=0;chromos<ChromosomeCount;chromos++) CheckHistoryChromosomes(chromos,historyHromosomes); } //————————————————————————————————————————————————————————————————————————
3.5. CheckHistoryChromosomes. 染色体ベースにおける染色体の検証
個々の染色体は、FFが計算される染色体ベース中にて検証されます。確証されれば、VFFがベースから取得され、そうでないならば、FFが呼び出されます。従って、繰り返しのFFの計算が除外されます。
//———————————————————————————————————————————————————————————————————————— //Verification of chromosome through the chromosome base. void CheckHistoryChromosomes ( int chromos, double &historyHromosomes[][100000] ) { //-----------------------Variables------------------------------------- int Ch1=0; //Index of the chromosome from the base int Ge =0; //Index of the gene int cnt=0; //Counter of unique genes. If at least one gene is different //- the chromosome is acknowledged unique //---------------------------------------------------------------------- //If at least one chromosome is stored in the base if(ChrCountInHistory>0) { //Enumerate the chromosomes in the base to find an identical one for(Ch1=0;Ch1<ChrCountInHistory && cnt<GeneCount;Ch1++) { cnt=0; //Compare the genes, while the gene index is less than the number of genes and while there are identical genes for(Ge=1;Ge<=GeneCount;Ge++) { if(Colony[Ge][chromos]!=historyHromosomes[Ge][Ch1]) break; cnt++; } } //If there are enough identical genes then we can use a ready- made solution from the base if(cnt==GeneCount) Colony[0][chromos]=historyHromosomes[0][Ch1-1]; //If there is no such chromosome, then we calculate the FF for it... else { FitnessFunction(chromos); //.. and if there is space in the base, then save it if(ChrCountInHistory<100000) { for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } } //If the base is empty, calculate the FF for it and save it in the base else { FitnessFunction(chromos); for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.6. CycleOfOperators. UGAの管理サイクル
ここでは、人工生成物の全時期における運命が決定されており、それは新しい世代が生まれ、死んでいくということです。これは以下のように発生します:二種類の親が交配のため選択されるか、一つの種が突然変異を起こすかです。GAのそれぞれの管理において、適切なパラメーターが決定されます。結果、一つの子孫を得ることになります。これが、子孫の集団が完全に埋め合わされるまで何度も繰り返されます。それから、子孫グループは、生息地に返され、個々がその能力を証明でき、我々はVFFを計算します。
「火、水、銅パイプ」によりテストされた後、子孫集団は種の一部に組み込まれます。人工進化の次の段階は、将来の世代と新たな種における「血」の枯渇を防ぐためのクローンの残酷な殺害です。
//———————————————————————————————————————————————————————————————————————— //Cycle of operators of UGA void CycleOfOperators ( double &historyHromosomes[][100000], //--- double ReplicationPortion, //Proportion of replications. double NMutationPortion, //Proportion of natural mutations. double ArtificialMutation, //Proportion of artificial mutations. double GenoMergingPortion, //Portion of borrowed genes. double CrossingOverPortion,//Proportion of cross-over. //--- double ReplicationOffset, //Coefficient of displacement of interval borders double NMutationProbability//Probability of mutation of each gene in % ) { //-----------------------Variables------------------------------------- double child[]; ArrayResize (child,GeneCount+1); ArrayInitialize(child,0.0); int gene=0,chromos=0, border=0; int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,6); ArrayInitialize(fit,0.0); //Counter of planting spots in a new population. int T=0; //---------------------------------------------------------------------- //Set a portion of operators of UGA double portion[6]; portion[0]=ReplicationPortion; //Proportion of replications. portion[1]=NMutationPortion; //Proportion of natural mutations. portion[2]=ArtificialMutation; //Proportion of artificial mutations. portion[3]=GenoMergingPortion; //Proportion of borrowed genes. portion[4]=CrossingOverPortion;//Proportion of cross- overs. portion[5]=0.0; //------------------------Cycle of operators of UGA--------- //Fill up the new colony with offspring while(T<ChromosomeCount) { //============================ for(i=0;i<6;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(portion[i]-portion[5]); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[4][1]); for(u=0;u<5;u++) { if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; } //============================ switch(u) { //--------------------- case 0: //------------------------Replication-------------------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { Replication(child,ReplicationOffset); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 1: //---------------------Natural mutation------------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { NaturalMutation(child,NMutationProbability); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 2: //----------------------Artificial mutation----------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { ArtificialMutation(child,ReplicationOffset); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast-forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 3: //-------------The creation of an individual with borrowed genes----------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { GenoMerging(child); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One space is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- default: //---------------------------Crossing-Over--------------------------- //If there is place in the new colony, create a new individual if(T<ChromosomeCount) { CrossingOver(child); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- } }//End of the cycle operators of UGA-- //Determine the fitness of each individual in the colony of offspring GetFitness(historyHromosomes); //Settle the offspring into the main population if(PopulChromosCount>=ChromosomeCount) { border=ChromosomeCount; PopulChromosCount=ChromosomeCount*2; } else { border=PopulChromosCount; PopulChromosCount+=ChromosomeCount; } for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=0;gene<=GeneCount;gene++) Population[gene][chromos+border]=Colony[gene][chromos]; //Prepare the population for the next reproduction RemovalDuplicates(); }//the end of the function //————————————————————————————————————————————————————————————————————————
3.7. Replication:複製
この操作は自然現象に最も近くあり、本質的には異なるが、生物学ではDNA複製と呼ばれます。しかし、同等のものを発見できなかったので、このタイトルにしておくこととしました。
複製は、最も重要な遺伝子操作の一つであり、親の染色体の特性を移しながら新しい遺伝子の生成を行います。アルゴリズムの収束の保証を行う主なオペレーターGAは、その他のオペレーターの使用なしで機能します。しかし、この場合、FFの起動数はより大きくなります。
複製の操作の規則を考慮してください。二つの親の染色体を使用します。新しい子孫の遺伝子は内部からのランダムの数です。
[C1-((C2-C1)*ReplicationOffset),C2+((C2-C1)*ReplicationOffset)]
C1 とC2 の親の遺伝子のReplicationOffset - [C1, C2]のインターバルの境界の移動率
例えば、父方の個体(青)と母方の個体(ピンク)からの子供(緑)が生成されます:
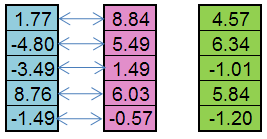
図5Replication オペレーターの動作の規則
子孫の遺伝子の可能性は以下のように示されます:
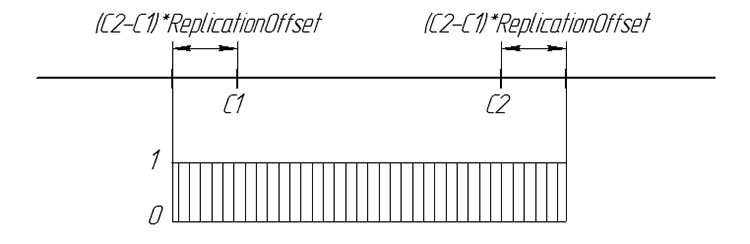
図6数字の行における子孫の遺伝子の様相の可能性
その他の子孫の遺伝子は、同様に生成されます。
//------------------------------------------------ ------------------------ // Replication void Replication ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- SelectTwoParents(address_mama,address_papa); //-------------------Cycle of gene enumeration-------------------------------- for(int i=1;i<=GeneCount;i++) { //----figure out where the father and mother came from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search had not gone over the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if we С1>C2, swi if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of the created gene Minimum = C1-((C2-C1)*ReplicationOffset); Maximum = C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search has not gone over the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- temp=RNDfromCI(Minimum,Maximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } //————————————————————————————————————————————————————————————————————————
3.8. NaturalMutation:自然突然変異
生細胞の中で生じるプロセスの中で突然変異は起こります。そして、自然選択の材料として機能します。普通の生息状態にて、細胞の生成の10^10の頻度で、一生のうちに自然に発生するものです。
私たち、興味を持った研究者たちは、必ずしも自然の秩序に従う必要はなく、遺伝子の次の突然変異を待ちました。パーセントで表示され、染色体中の遺伝子の突然変異の確率を決定するNMutationProbabilityパラメーターが手助けとなります。
NaturalMutation オペレーターにおいて、突然変異は [RangeMinimum, RangeMaximum].のランダムな遺伝子の生成から成り立ちます。NMutationProbabilityが100%というのは、染色体中のすべての遺伝子の100%の突然変異を意味し、 NMutationProbabilityが0%というのは、全く突然変異がないことを指します。後者は、実際的な問題において使用する上で不適任です。
//------------------------------------------------ ------------------------ // The natural mutation. void NaturalMutation ( double &child[], double NMutationProbability ) { //-----------------------Variables------------------------------------- int address=0; double prob=0.0; //---------------------------------------------------------------------- if(NMutationProbability<0.0) prob=0.0; if(NMutationProbability>100.0) prob=100.0; //-----------------Parent selection------------------------ SelectOneParent(address); //--------------------------------------- for(int i=1;i<=GeneCount;i++) if(RNDfromCI(0.0,100.0)<prob) child[i]=NormalizeDouble( SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits ); } //————————————————————————————————————————————————————————————————————————
3.9. ArtificailMutation:人工的突然変異
そのオペレーターの主な仕事は、「新鮮な」血の生成です。二つの親を使用し、子孫の遺伝子は親の遺伝子により置換されていない位置の業から選択します。GAがローカルの極値に止まることを防いでください。多くは、他のオペレーターと比較した際、収束を加速させるか、遅くし、FFの起動数を増やします。
Replicationにおけるのと同様、二つの親の染色体を使用します。しかし、ArtificalMutationオペレーターの仕事は、子孫に親の特性を移すことではなく、子孫を親の特性から異ならせることです。従って、全く逆の方法であり、同じインターバールの境界線の変化率を使用するが、遺伝子はReplicationにより除去される予定であったインターバル内に生成されます。子孫の新しい遺伝子は、インターバル [RangeMinimum, C1-(C2-C1) * ReplicationOffset]と[C2 + (C2-C1) * ReplicationOffset, RangeMaximum]からのランダムの数字である。
図表では、子孫の遺伝子の確率ReplicationOffset 0.25は以下のように表示されます:
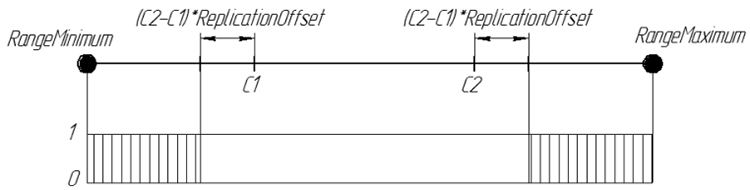
図7子孫の遺伝子の確率 ReplicationOffset、0.25は [RangeMinimum; RangeMaximum]のインターバル間の実線上である。
//———————————————————————————————————————————————————————————————————————— //Artificial mutation. void ArtificialMutation ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0,p=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //-------------------Cycle of genes enumeration------------------------------ for(int i=1;i<=GeneCount;i++) { //----determine where the mother and father are from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search doesn't go beyond the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if С1>C2, we change their places if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of creating the new gene Minimum=C1-((C2-C1)*ReplicationOffset); Maximum=C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search doesn't go beyond the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- p=MathRand(); if(p<16383.5) { temp=RNDfromCI(RangeMinimum,Minimum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } else { temp=RNDfromCI(Maximum,RangeMaximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } } //————————————————————————————————————————————————————————————————————————
3.10. GenoMerging 遺伝子の借用
特定のGAオペレーターは、同等の自然物を持ちません。実際、この素晴らしい仕組みがどのように有機体の中で機能しているのか想像するのは容易くありません。しかし、多くの親から子へ移動する遺伝子(親の数は、遺伝子の数と同じです)の特徴的な性質を持っています。そのオペレーターは新しい遺伝子を生成させない、組み合わせ検索の仕組みです。
以下のように動作します:最初の子孫遺伝子にとって、親は選択され、最初の遺伝子は親から取得されています。そして二つ目の遺伝子は、二つ目の親から選択され、その親から受け継ぎます。これは、遺伝子の数が0以上なら適用が望ましいです。さもなくば、オペレーターは染色体の複製を生成するので、使用すべきではありません。
//———————————————————————————————————————————————————————————————————————— //Borrowing genes. void GenoMerging ( double &child[] ) { //-----------------------Variables------------------------------------- int address=0; //---------------------------------------------------------------------- for(int i=1;i<=GeneCount;i++) { //-----------------Selecting parents------------------------ SelectOneParent(address); //-------------------------------------------------------- child[i]=Population[i][address]; } } //————————————————————————————————————————————————————————————————————————
3.11. CrossingOver. 乗り換え
乗り換え (交配としても生物学では知られている) は、染色体の交換を行う現象です。GenoMergingのように、これは組み合わせ探索の仕組みです。
二つの親の染色体が選択されます。両方がランダムな場所にて「切られ」ます子孫の染色体は、親の染色体の部分から成り立ちます。
図で表したほうがこの仕組みを紹介しやすいと思います。
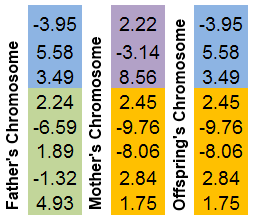
図8染色体の交換の仕組み
これは、遺伝子の数が0以上なら適用が望ましいです。さもなくば、オペレーターは染色体の複製を生成するので、使用すべきではありません。
//———————————————————————————————————————————————————————————————————————— //Crossing-over. void CrossingOver ( double &child[] ) { //-----------------------Variables------------------------------------- int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //Determine the breakage point int address_of_gene=(int)MathFloor((GeneCount-1)*(MathRand()/32767.5)); for(int i=1;i<=GeneCount;i++) { //----copy the mother's genes-------- if(i<=address_of_gene+1) child[i]=Population[i][address_mama]; //----copy the father's genes-------- else child[i]=Population[i][address_papa]; } } //————————————————————————————————————————————————————————————————————————
3.12. SelectTwoParents. 二種類の親の選択
遺伝子プールの枯渇を防ぐため、同じ種による交雑育種の禁止があります。10つの試みが異なる親を発見するためになされ、ペアを見つけるのに失敗すれば、同種での交雑育種を許します。基本的に、同じ種類のコピーを得ることになります。
一方、個体のクローン化の確率は減少し、他方、いくつかのステップ数で異なるペアを見つけることは実質的に不可能に近いので検索の循環性は、防がれます。
Replication、ArtificialMutation、CrossingOverのオペレーターにて使用されます。
//———————————————————————————————————————————————————————————————————————— //Selection of two parents. void SelectTwoParents ( int &address_mama, int &address_papa ) { //-----------------------Variables------------------------------------- int cnt=1; address_mama=0;//address of the mother individual in a population address_papa=0;//address of the father individual in a population //---------------------------------------------------------------------- //----------------------------Selection of parents-------------------------- //Ten attempts to chose different parents. while(cnt<=10) { //For the mother individual address_mama=NaturalSelection(); //For the father individual address_papa=NaturalSelection(); if(address_mama!=address_papa) break; } //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.13. SelectOneParent. 一つの親の選択
一つの親が選択されるだけなので、ここではすべてが簡単です。
NaturalMutationやGenoMergingなどのオペレーターにて使用されます。
//———————————————————————————————————————————————————————————————————————— //Selection of one parent. void SelectOneParent ( int &address//address of the parent individual in the population ) { //-----------------------Variables------------------------------------- address=0; //---------------------------------------------------------------------- //----------------------------Selecting a parent-------------------------- address=NaturalSelection(); //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.14. NaturalSelection. 自然選択
自然選択 - 生存可能性や、環境の状況に適応し、適した遺伝的特性を継承する望ましい個体の生殖につながるプロセスです。
そのオペレーターは、伝統的なオペレーター「Roulette」と類似しています。<分節 1528> (ルーレット回転盤セレクション - ルーレットの「起動」により個体を選択します。ルーレットの回転盤は、種の一つのメンバーにつき一つのセクションが設けられています。セクターのサイズは、適合性の値に比例しています)しかし、重大な違いがあります。最大・最小の値に関連した個々の位置を考慮します。さらに、最悪の遺伝子を持つ個体も子孫を残す可能性があります。公平ではないでしょうか?公平というわけではありません。しかし、自然界の事実においては、すべての個体が子孫を残す機会が与えられているのです。
例えば、最大化の問題において、以下のVFFを持つ:(256, 128, 64, 32, 16, 8, 4, 2, 0, -1、より大きい値がより良い適合性を持ちます。)10の個体を取ります。隣り合う個体間の「距離」は、二つ前の個体の間よりも2倍大きいということを理解するため、この例えを用いました。しかし、円グラフでは、個々の子孫を残す個体の確率は以下のように示されています。
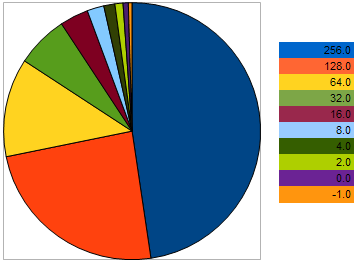
図9親の個体の選択における確率グラフ
最悪の遺伝子へのアプローチにより、彼らの可能性はより悪くなります。逆に、より良い種へ個体が近づけば近づくほど、生殖の可能性は高くなります。

図10親の個体の選択における確率グラフ
//———————————————————————————————————————————————————————————————————————— //Natural selection. int NaturalSelection() { //-----------------------Variables------------------------------------- int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,PopulChromosCount); ArrayInitialize(fit,0.0); double delta=(Population[0][0]-Population[0][PopulChromosCount-1])*0.01-Population[0][PopulChromosCount-1]; //---------------------------------------------------------------------- for(i=0;i<PopulChromosCount;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(Population[0][i]+delta); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[PopulChromosCount-1][1]); for(u=0;u<PopulChromosCount;u++) if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; return(u); } //————————————————————————————————————————————————————————————————————————
3.15. RemovalDuplicates. 複製の除去
その機能は種の複製された染色体を除去し、残った独自の染色体(現世代の種において独自)はVFFや、最適化の種類、増減によって分類されます。
//———————————————————————————————————————————————————————————————————————— //Removing duplicates sorted by VFF void RemovalDuplicates() { //-----------------------Variables------------------------------------- int chromosomeUnique[1000];//Array stores the unique trait //of each chromosome: 0-duplicate, 1-unique ArrayInitialize(chromosomeUnique,1); //Assume that there are no duplicates double PopulationTemp[][1000]; ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Ge =0; //Index of the gene int Ch =0; //Index of the chromosome int Ch2=0; //Index of the second chromosome int cnt=0; //Counter //---------------------------------------------------------------------- //----------------------Remove duplicates---------------------------1 //Chose the first from the pair for comparison... for(Ch=0;Ch<PopulChromosCount;Ch++) { //If it's not a duplicate... if(chromosomeUnique[Ch]!=0) { //Chose the second from the pair... for(Ch2=0;Ch2<PopulChromosCount;Ch2++) { if(Ch!=Ch2 && chromosomeUnique[Ch2]!=0) { //Zeroize the counter of identical genes cnt=0; //Compare the genes. while there are identical genes present for(Ge=1;Ge<=GeneCount;Ge++) { if(Population[Ge][Ch]!=Population[Ge][Ch2]) break; else cnt++; } //If there are the same amount of identical genes as total genes //..the chromosome is considered a duplicate if(cnt==GeneCount) chromosomeUnique[Ch2]=0; } } } } //The counter calculates the number of unique chromosomes cnt=0; //Copy the unique chromosomes into a temporary array for(Ch=0;Ch<PopulChromosCount;Ch++) { //If the chromosome is unique, copy it, if not, go to the next if(chromosomeUnique[Ch]==1) { for(Ge=0;Ge<=GeneCount;Ge++) PopulationTemp[Ge][cnt]=Population[Ge][Ch]; cnt++; } } //Assigning the variable "All chromosomes" the value of counter of unique chromosomes PopulChromosCount=cnt; //Return unique chromosomes back to the array for temporary storage //..of combined populations for(Ch=0;Ch<PopulChromosCount;Ch++) for(Ge=0;Ge<=GeneCount;Ge++) Population[Ge][Ch]=PopulationTemp[Ge][Ch]; //=================================================================1 //----------------Ranking the population---------------------------2 PopulationRanking(); //=================================================================2 } //————————————————————————————————————————————————————————————————————————
3.16. PopulationRanking. 種のランク付け
VFFによって分類されます。そのメソッドは、「 bubbly そのアルゴリズムは、分類された配列への繰り返しの通過により成り立ちます通るごとに、要素は順番にペアで比較され、もしペアの順序が間違っていれば、要素の交換がなされます。配列の通過は、交換がもはや必要ではなくなる、つまり、配列のソートが完了するまで繰り返されます。
アルゴリズムを順次通過していく際、水の中の泡のように目立つ要素は望ましい位置に「ポップアップ」する。しかし、配列の内容ではなく、配列のインデックスのみがソートされるという違いがある。このメソッドはより速く、一つの配列を別のところへコピーするスピードとは少し異なります。そして、分類される配列が大きければ、違いは小さくなっていきます。
//———————————————————————————————————————————————————————————————————————— //Population ranking. void PopulationRanking() { //-----------------------Variables------------------------------------- int cnt=1, i = 0, u = 0; double PopulationTemp[][1000]; //Temporary population ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Indexes[]; //Indexes of chromosomes ArrayResize (Indexes,PopulChromosCount); ArrayInitialize(Indexes,0); int t0=0; double ValueOnIndexes[]; //VFF of corresponding //..chromosome indexes ArrayResize (ValueOnIndexes,PopulChromosCount); ArrayInitialize(ValueOnIndexes,0.0); double t1=0.0; //---------------------------------------------------------------------- //Fill in the indexes in the temporary array temp2 and //...copy the first line from the sorted array for(i=0;i<PopulChromosCount;i++) { Indexes[i] = i; ValueOnIndexes[i] = Population[0][i]; } if(OptimizeMethod==1) { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]>ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } else { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]<ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } //Create a sorted-out array based on the obtained indexes for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) PopulationTemp[i][u]=Population[i][Indexes[u]]; //Copy the sorted-out array back for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) Population[i][u]=PopulationTemp[i][u]; } //————————————————————————————————————————————————————————————————————————
3.17. RNDfromCustomInterval. 特定のインターバル間におけるランダムな数字の生成
使いやすい機能です。UGAにても使いやすいです。
//———————————————————————————————————————————————————————————————————————— //Generator of random numbers from the selected interval. double RNDfromCI(double RangeMinimum,double RangeMaximum) { return(RangeMinimum+((RangeMaximum-RangeMinimum)*MathRand()/32767.5));} //————————————————————————————————————————————————————————————————————————
3.18. SelectInDiscreteSpace. 分離した区間での選択
探索区間を減らすために使用されます。パラメーターstepが0.0で、検索は連続的な区間で実行されます。(15番目に重要なシンボルにMQLの言語的制限がなされます。)GAアルゴリズムをより精度の高い正確性で使用したい場合、追加のより大きい数字を扱うライブラリを記述する必要があります。
RoundMode 1での関数の動作は、以下の図に示されています:
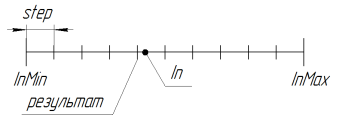
図11 RoundMode = 1のSelectDiscreteSpace関数の動作
//———————————————————————————————————————————————————————————————————————— //Selection in discrete space. //Modes: //1-closest below //2-closest above //any closest double SelectInDiscreteSpace ( double In, double InMin, double InMax, double step, int RoundMode ) { if(step==0.0) return(In); // secure the correctness of borders if(InMax < InMin) { double temp = InMax; InMax = InMin; InMin = temp; } // during a breach - return the breached border if(In < InMin) return(InMin); if(In > InMax) return(InMax); if(InMax == InMin || step <= 0.0) return(InMin); // bring to the specified scale step = (InMax - InMin) / MathCeil ((InMax - InMin) / step); switch(RoundMode) { case 1: return(InMin + step * MathFloor ((In - InMin) / step)); case 2: return(InMin + step * MathCeil ((In - InMin) / step)); default: return(InMin + step * MathRound ((In - InMin) / step)); } } //————————————————————————————————————————————————————————————————————————
3.19. FitnessFunction. 適合性関数
GAの一部ではありません。その関数は、VFFが計算される種の染色体インデックスを取得します。VFFは、移動された染色体の0インデックスにて記述されます。この関数のコードは、それぞれのタスクに固有です。
3.20. ServiceFunction. サービス関数
GAの一部ではありません。この関数のコードはそれぞれの特定のタスクに特有です。数世代上のコントロールを行う上で使用されます。例えば、現世代の最も良いVFFを表示するためです。
4. UGAの動作例
すべての最適化の問題はEAにより解決され、二種類に分けられます。
- 遺伝子型は表現型から成り立ちます。染色体の遺伝子の値は、直接最適化関数の引数より割り当てられます。例1.
- 遺伝子型は、表現型に合致していません。染色体遺伝子の意味の解釈は、最適化関数の計算に必要とされます。例2.
4.1. 例1
知られている答えの問題を、アルゴリズムが動くことを確認するため、考察してみてください、そして、その問題の解決へ進みましょう。その問題の解法は多くのトレーダーにとって興味深いものです。
問題: 最小・最大の関数「Skin」を見つける
[-5, 5] セグメント上
答え: fmin (3.07021,3.315935) = -4.3182, fmax (-3.315699; -3.072485) = 14.0606.
図12. [-5, 5]セグメント上「Skin」のグラフ
問題を解くため、以下のスクリプトを記述します。
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" #include "Skin.mqh"//testing function //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Gene pool parameter----"; input int ChromosomeCount_P = 50; //Number of chromosomes in a colony input int GeneCount_P = 2; //Number of genes input int FFNormalizeDigits_P = 4; //Number of fitness symbols input int GeneNormalizeDigits_P = 4; //Number of genes input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Operator parameters----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing-over. //--- input double ReplicationOffset_P = 0.5; //Coefficient of interval borders displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = -5.0; //Minimum range search input double RangeMaximum_P = 5.0; //Maximum range search input double Precision_P = 0.0001;//The required accuracy input int OptimizeMethod_P = 1; //Optim.:1-Min,other-Max //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double ERROR=0.0;//Average error in gen //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparing global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum range search RangeMaximum =RangeMaximum_P; //Maximum range search Precision =Precision_P; //Search step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of symbols in fitness GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//Proportion of crossing-over. //--- ReplicationOffset_P, //Coefficient of interval border replacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," mc - Time of implementation"); //---------------------------------- return(0); } //————————————————————————————————————————————————————————————————————————
ここにその問題を解決するスクリプトコードがあります。稼働させ、Comment()関数が提示する情報を取得していください。
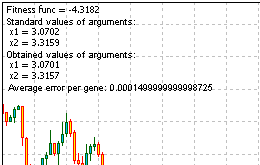
図13. その問題の解決の結果
結果を見ると、アルゴリズムが作動していることがわかります。
4.2. 例2.
ZZインジケーターは、トレーディングシステムの理想の入力を表示すると広く信じられています。そのインジケーターは、「波動説」信者や、「数字」の大きさを決定するために使用する人たちの中でとても人気です。
問題 :ZZ頂点から異なる履歴データのトレーディングシステムの他のエントリーポイントがあるかないか?
その実験に関して、M1 100バーのGBJPYペアを選択します。(5の数字の引用句)80ポイントの広がりを受け入れてください始めるために、最良のZZパラメーターを決定する必要があります。このためには、ExtDepthパラメーターの最良の値を発見する上で簡単な列挙を行います。以下の簡単なスクリプトを使用します。
#property script_show_inputs //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input int History=100; input double Spred =80.0; input int Depth =5; //For "one-time" use input bool loop =true;//Use enumeration or not //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- void OnStart() { //-----------------------Variables------------------------------------- double ZigzagBuffer [];//For storing the buffer of the ZZ indicator double PeaksOfZigzag[];//for storing the values of the ZZ extremum int Zigzag_handle; //Indicator marker ArraySetAsSeries(ZigzagBuffer,true); ArrayResize(PeaksOfZigzag,History); int depth=3; double PipsSum=0.0; int PeaksCount=0; bool flag=true; //---------------------------------------------------------------------- if(loop==true) { while(depth<200 && flag==true) { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",depth); //--- reset the code error ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<100;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Could not copy the indicator buffer. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- PipsSum=0.0; PeaksCount=0; for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; //----------------------------------------------------------- if(PeaksCount<=2) flag=false; else { Print(depth," ",PeaksCount," ",PipsSum); depth+=1; } //----------------------------------------------------------- } } else { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",Depth); //--- reser the error code ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<History;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Was not able to copy the buffer indicator. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; } Print(Depth," ",PeaksCount," ",PipsSum); //----------------------------------------------------------- } } //————————————————————————————————————————————————————————————————————————
そのスクリプトを稼働させると、ExtDepthが2の40077ポイントを取得できます。19のインジケーターの頂点が100のバーにて「フィット」します。ExtDepthの増加に伴い、ZZ頂点の数は低下し、確率も降下します。
さあ、UGAを使用し、ZZの代替となる頂点を見つけることができます。ZZ頂点は、それぞれのバーにつき3つの位置を持ちます。1) High(高い) 2) Low(低い) 3) No vertex(頂点なし)頂点の位置と存在は、それぞれのバーのすべての遺伝子により実現されます。従って、染色体の量は100の遺伝子となります。
私の計算結果によれば(数学者は間違いがあれば正してください)、100のバーにおいて 3 ^ 100か5.15378e47 別の選択肢"zigzags"を構築できます。これは、直接計数を用い行旅されるべき選択肢の正確な数字です。毎秒100000000オプションのスピードでの計算は、1.6e32年必要となります。 !これは宇宙の年齢よりも多いです。ここで、この問題への解法の発見能力について疑い始めました。
しかし、始めましょう。
UGAが実数による染色体の表現を行うので、なんとか頂点の位置を符号化する必要があります。これは、染色体の遺伝子型が表現型と合致しないケースです。遺伝子[0, 5].のインターバル探索を行います。インターバル[0, 1]はHighにおけるZZの頂点と合致し、インターバル[4, 5]はLowにおける頂点と合致します。そしてインターバル[1, 4]は、頂点の不在と合致します。
一つの重要な点を考える必要があります。プロト種は、ランダムに特定のインターバル間の遺伝子とともに生成されるので、最初の種は、とてもひどい結果になり、おそらく、マイナスサインで数百のポイントであろうと思われます。最初の世代でも少なからずチャンスはありますが、いくつかの世代の後、頂点の不在と合致した遺伝子を持つ種が出現します。これは、特製がなく、不可避な拡散の報いであることを指します。
数人のトレーダーによると:「トレードにおいて最良な戦略はトレードを行わないことだ」と言いますこの個体は、人工的進化の頂点になります。この「人工的」進化がトレーダーに生を与えるとためには、つまり、代替ZZの頂点を調節するためには、個々の適合性を割り当て、進化のはしごの底辺に配置させる 「-10000000.0」の値、頂点を失う必要があります。
ここに、代替ZZの頂点を発見するUGAを使用するスクリプトコードがあります。
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Parameters of the gene pool----"; input int ChromosomeCount_P = 100; //Number of chromosomes in the colony input int GeneCount_P = 100; //Number of genes input int FFNormalizeDigits_P = 0; //Number of fitness symbols input int GeneNormalizeDigits_P= 0; //Number of gene symbols input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Parameters of operators----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing - over. input double ReplicationOffset_P = 0.5; //Coefficient of interval border displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = 0.0; //Minimum search range input double RangeMaximum_P = 5.0; //Maximum search range input double Precision_P = 1.0; //Required accuracy input int OptimizeMethod_P = 2; //Optim.:1-Min,other -Max input string Other = "----Other----"; input double Spred = 80.0; input bool Show = true; //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double Hight []; double Low []; datetime Time []; datetime Ti []; double Peaks []; bool show; //———————————————————————————————————————————————————————————————————————— //--------------------------Body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparation of global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum search range RangeMaximum =RangeMaximum_P; //Maximum search range Precision =Precision_P; //Searching step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of fitness symbols GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Preparation of global variables ArraySetAsSeries(Hight,true); CopyHigh (NULL,0,0,GeneCount+1,Hight); ArraySetAsSeries(Low,true); CopyLow (NULL,0,0,GeneCount+1,Low); ArraySetAsSeries(Time,true); CopyTime (NULL,0,0,GeneCount+1,Time); ArrayResize (Ti,GeneCount+1);ArrayInitialize(Ti,0); ArrayResize(Peaks,GeneCount+1);ArrayInitialize(Peaks,0.0); show=Show; //---------------------------------------------------------------------- //local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Очистим экран ObjectsDeleteAll(0,-1,-1); ChartRedraw(0); //launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of replication of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//proportion of crossing- over. //--- ReplicationOffset_P, //Coefficient of interval border displacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- //Display the last result on the screen show=true; ServiceFunction(); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," мс - time of execution"); //---------------------------------- return(0); } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Service function. Called up from UGA. | | //If there is no need for it, leave the function empty, like this: | // void ServiceFunction() | // { | // } | //-----------------------------------------------------------------------+ void ServiceFunction() { if(show==true) { //-----------------------Variables----------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //-------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Chromosome[u]; if(temp<=1.0) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } } ObjectsDeleteAll(0,-1,-1); for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; ObjectCreate (0,"BoxBackName"+(string)V,OBJ_TREND,0,Ti[V],Peaks[V],Ti[V+1],Peaks[V+1]); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_COLOR,Black); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_SELECTABLE,true); } ChartRedraw(0); Comment(PipsSum); } //---------------------------------------------------------------------- else return; } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Function of determining the fitness of the individual. Called up from UGA. | //-----------------------------------------------------------------------+ void FitnessFunction(int chromos) { //-----------------------Variables------------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //---------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Colony[u][chromos]; if(temp<=1.0) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); PeaksCount++; } } if(PeaksCount>1) { for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; Colony[0][chromos]=PipsSum; } else Colony[0][chromos]=-10000000.0; AmountStartsFF++; } //————————————————————————————————————————————————————————————————————————
このスクリプトを稼働させると、4939ポイントの利益を持つ頂点を得ることができます。さらに、直接計数に必要な3^100と比較し、17,929回のステップでポイントを得ることができましt。私のコンピューターでは、1.6e32年に対して21.7秒で完了しました。
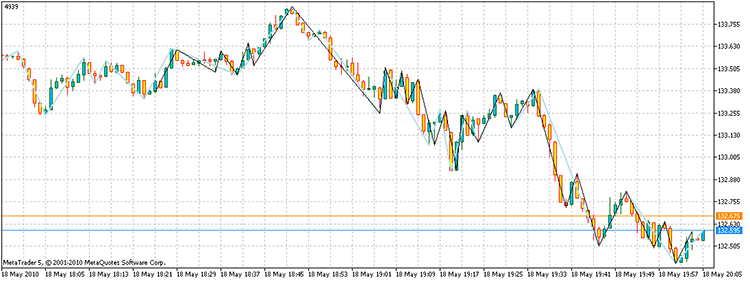
図14. 問題の解決の結果黒く塗られているセグメント - 代替ZZ、スカイブルー - ZZインジケーター
質問に対する答えは、次の「存在する」というものになるでしょう。
5. UGAを扱う際の推奨
- 適切な結果をアルゴリズムから得られるよう、FFに正しく予想される状況を設定しましょう。例2を振り返ってみてください。これはおそらくメインの推奨になると思います。
- Precisionパラメーターには小すぎる値を入れないでください。アルゴリズムは、ステップ0を扱うことができるが、合理的な精度の解法を要求してください。このパラメーターは、問題の次元を減らすことを意図しています。
- 種のサイズや、世代数の値を変化させてください。よい解法は、MaxOfCurrentEpochに表示される二倍大きいEpochパラメーターを挿入することになります。大きすぎる値を選択してはいけません。これにより、問題への解法の発見を加速できるというわけではありません。
- 遺伝子オペレーターのパラメーターを実験してみてください。普遍的なパラメーターはなく、タスクの状況に基づいて設定する必要があります。
発見
強力なスタッフィングターミナルストラテジーテスターと同様、MQL5言語は、複雑な問題を解決するトレーダーにとって強力なツールの作成を可能にします。とても柔軟で拡張性のある最適化アルゴリズムを獲得できます。そして、第一人者ではないにしても、この発見を恥じることなく誇りに思っています。
UGAは始め追加のオペレーターや計算ブロックと同調し、修正しやすくなるように設計されました。なので、読者の皆様は簡単に「人工的な」進化の開発へ貢献することができます。
読者の皆様が最適な解法を発見することを祈っています。この記事が手助けになることを願っています。がんばってください!
注意, ZigZagというインジケーターを記事にて使用しています。UGAのすべてのコードは添付されています。
ライセンス: 記事に添付されているソースコード(UGAコード)は、 BSD ライセンスの認可にて配布されています。
MetaQuotes Ltdによってロシア語から翻訳されました。
元の記事: https://www.mql5.com/ru/articles/55
 異なる国での異なるタイムゾーンに基づくトレーディング戦略例
異なる国での異なるタイムゾーンに基づくトレーディング戦略例
 「サル」でもわかるMQL:オブジェクトクラスの設計 構築方法
「サル」でもわかるMQL:オブジェクトクラスの設計 構築方法
 トレードレポートとSMS通知の作成と発行
トレードレポートとSMS通知の作成と発行
 初心者のための複数インディケータバッファの作成
初心者のための複数インディケータバッファの作成
- 無料取引アプリ
- 8千を超えるシグナルをコピー
- 金融ニュースで金融マーケットを探索