
Genetik Algoritmalar - Çok Kolay!
Giriş
Genetik algoritma ( GA ), pratik olarak önemli durumların çoğunda problem için kabul edilebilir bir çözüm sunan, ancak kararların doğruluğu matematiksel olarak kanıtlanmamış olan buluşsal algoritmayı ( EA ) ifade eder ve çoğunlukla analitik çözümü çok zor hatta imkansız olan problemler için kullanılır.
Bu sınıfın (NP sınıfı) bir probleminin klasik bir örneği, bir "gezgin satıcı sorunu"dur (en ünlü kombinatoryal optimizasyon problemlerinden biridir). Asıl zorluk, verilen şehirlerden en az bir kez geçen ve ardından ilk şehre dönen en avantajlı rotayı bulmaktır). Ancak hiçbir şey onları resmileştirmeye yol açan görevler için kullanmayı engellemez.
EA, önemli miktarda zaman alan tüm seçenekleri gözden geçirmek yerine, yüksek hesaplama karmaşıklığı içeren problemleri çözmek için yaygın olarak kullanılmaktadır. Desen tanıma gibi yapay zeka alanlarında, virüsten koruma yazılımlarında, mühendislikte, bilgisayar oyunlarında ve diğer alanlarda kullanılırlar.
MetaQuotes Software Corp.'un MetaTrader4 / 5 yazılım ürünlerinde GA kullandığı belirtilmelidir. Strateji test cihazını ve yerleşik bir strateji optimize ediciyi kullanarak ne kadar zaman ve emekten tasarruf edilebileceğini hepimiz biliyoruz; burada, tıpkı doğrudan numaralandırma ile olduğu gibi, GA kullanımıyla optimizasyon mümkündür. Ayrıca, MetaTrader 5 test cihazı, kullanıcı optimizasyon kriterlerini kullanmamıza olanak tanır. Belki okuyucu, doğrudan numaralandırma'nın aksine GA hakkındaki ve EA'nın sağladığı avantajlar hakkındaki makaleleri okumakla ilgilenecektir.
1. Biraz geçmişe gidelim
Bir yıldan biraz daha uzun bir süre önce, nöral ağlar konusunda eğitmek vermek için bir optimizasyon algoritmasına ihtiyacım oldu. Çeşitli algoritmaları hızla öğrendikten sonra GA'yı seçtim. Hazır uygulamalar için yaptığım araştırmalar sonucunda, genel erişime açık olanların ya optimize edilebilecek parametre sayısı gibi işlevsel sınırlamaları olduğunu ya da çok "ince ayarlı" olduğunu gördüm.
Yalnızca tüm nöral ağ türleri konusunda eğitim vermek için değil, aynı zamanda genel olarak herhangi bir optimizasyon problemini çözmek için de evrensel olarak esnek bir enstrümana ihtiyacım vardı. Yabancı "genetik oluşumlar" üzerine kapsamlı bir çalışmadan sonra, bunların nasıl çalıştığını hala anlayamamıştım. Bunun nedeni ya ayrıntılı bir kod stili ya da programlama konusundaki deneyim eksikliğim veya muhtemelen her ikisi birdendi.
Temel zorluklar, genleri ikili bir koda kodlamamdan ve daha sonra onlarla bu şekilde çalışmamdan kaynaklanıyordu. Her halükarda, gelecekte ölçeklenebilirlik ve kolay modifikasyona odaklanan sıfırdan bir genetik algoritma yazmaya karar verdim.
İkili dönüşümle uğraşmak istemedim ve genlerle doğrudan çalışmaya karar verdim; yani kromozomu gerçek sayılar şeklinde bir dizi genle temsil ettim. Kromozomların gerçek sayılarla temsil edildiği genetik algoritmamın kodu bu şekilde ortaya çıktı. Daha sonra, yeni bir şey keşfetmediğimi ve benzer genetik algoritmaların (bunlar gerçek kodlu GA olarak adlandırılır) 15 yıldan uzun bir süredir, onlar hakkında ilk yayınlar çıktığından beri var olduğunu öğrendim.
Pratik problemlerde kullanımıyla ilgili kişisel deneyime dayandığı için, GA işleyişinin uygulanmasına ve prensiplerine yaklaşım vizyonumun değerlendirmesini okuyucuya bırakıyorum.
2. GA Açıklaması
GA, doğanın kendisinden ödünç alınan prensipleri içerir. Bunlar, kalıtım ve değişkenlik prensipleridir. Kalıtım, organizmaların özelliklerini ve evrimsel özelliklerini yavrularına aktarma yeteneğidir. Bu yetenek sayesinde tüm canlılar yavrularına kendi türlerine ilişkin özellikler bırakırlar.
Canlı organizmalardaki genlerin değişkenliği, popülasyonun genetik çeşitliliğini garanti eder ve bu, rastlantısaldır; zira doğa gelecekte hangi özelliklerin en çok tercih edileceğini önceden bilmenin bir yoluna sahip değildir (iklim değişikliği, gıda azalması / artışı, rekabet eden türlerin ortaya çıkması vb.). Bu değişkenlik, habitatın yeni, değiştirilmiş koşullarında hayatta kalabilen ve geride yavru bırakabilen yeni özelliklere sahip yaratıkların ortaya çıkmasına izin verir.
Biyolojide, mutasyonların ortaya çıkmasından kaynaklanan değişkenlik mutasyonel, genlerin çiftleşme yoluyla daha fazla çapraz kombinasyonundan kaynaklanan değişkenlik kombinasyonel olarak adlandırılır. Bu tür varyasyonların her ikisi de GA'da uygulanmaktadır. Ayrıca, mutasyonların doğal mekanizmasını (DNA'nın nükleotid dizisindeki değişiklikler) taklit eden bir mutajenez uygulaması vardır - doğal (kendiliğinden) ve yapay (uyarılmış).
Algoritmanın ölçütüne göre en basit bilgi aktarımı birimi gendir ; bu, belirli bir özelliğin gelişimini kontrol eden kalıtımın yapısal ve işlevsel birimidir. İşlevin bir değişkenini gen olarak adlandıracağız. Gen, gerçek bir sayı ile temsil edilir. İncelenen işlevin gen değişkenleri kümesi, - kromozomun karakterize edici özelliğidir.
Kromozomu bir sütun şeklinde temsil ettiğimizi düşünelim. O zaman f (x) = x ^ 2 işlevinin kromozomu şu şekilde görünecektir:
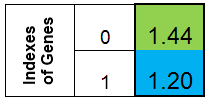
Şekil 1. F (x) = x ^ 2 işlevinin kromozomu
burada 0. indeks - bireylerin adaptasyonu olarak adlandırılan f (x) işlevinin değeri (bu işlevi uygunluk işlevi - FF ve işlevin değeri - VFF olarak adlandıracağız) ). Kromozomu tek boyutlu bir dizide saklamak uygundur. Bu, çift Kromozom [] dizisidir.
Aynı evrimsel dönemin tüm numuneleri bir popülasyonda birleştirilir. Ayrıca, popülasyon rastgele olarak iki eşit koloniye ayrılır; ana ve alt koloniler. Tüm popülasyondan seçilen ebeveyn türleri ve GA'nın diğer işleçlerini çaprazlamanın bir sonucu olarak, popülasyonun yarısına eşit olan ve popülasyondaki yavru kolonisinin yerini alan bir yeni yavru kolonisi vardır.
Minimum f (x) = x ^ 2 işlevinin aranması sırasında bireylerin toplam popülasyonu şu şekilde görünebilir:
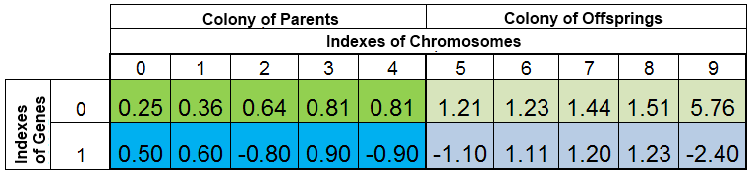
Şekil 2. Toplam birey popülasyonu
Popülasyon VFF'ye göre sıralanır. Burada, kromozomun 0. indeksi, en küçük VFF'ye sahip numune tarafından alınır. Yeni yavru, yalnızca soyundan gelen kolonideki bireylerin yerini tamamen alırken, ana koloni bozulmadan kalır. Ancak, çift numuneler yok edildiği, daha sonra yeni yavru ana kolonideki boşlukları doldurduğu ve geri kalanlar soyundan gelen koloniye yerleştirildiği için, ana koloni her zaman tam olmayabilir.
Diğer bir deyişle, popülasyon boyutu nadiren sabittir ve neredeyse doğada olduğu gibi çağdan çağa değişir. Örneğin, üreme öncesi ve üreme sonrası popülasyon şöyle görünebilir:
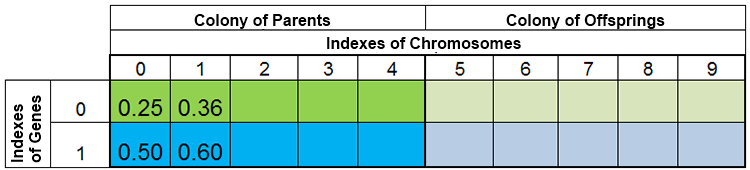
Şekil 3. Üreme öncesi popülasyon
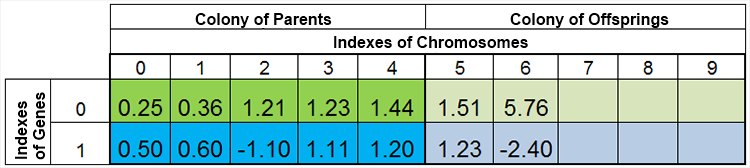
Şekil 4. Üreme sonrası popülasyon
Popülasyonun soylar tarafından "yarı" yerine getirilmesinin açıklanan mekanizmasının yanı sıra, çift olanların yok edilmesi ve bireylerin kendileriyle melezlenmesinin yasaklanmasının tek bir amacı vardır; "darboğaz etkisinden" kaçınmak (yani bir türün tamamen yok olmasına yol açabilecek bir dizi farklı nedenden dolayı kritik bir azalmanın sonucu olarak popülasyonun gen havuzunun azalması anlamına gelen bir biyolojik terim, GA, benzersiz kromozomların görünümünün sonuyla karşı karşıyadır ve yerel ekstremumların birinde "sıkışıp kalmak".)
3. UGAlib işlevinin açıklaması
GA Algoritması:- Bir proto-popülasyon oluşturmak. Genler, belirli bir aralıkta rastgele oluşturulur.
- Her bireyin uygunluğunun belirlenmesi veya diğer bir deyişle VFF'nin hesaplanması.
- Kromozom kopyalarını çıkardıktan sonra popülasyonun üreme için hazırlanması.
- Referans kromozomun izolasyonu ve korunması (en iyi VFF ile).
- UGA operatörleri (seçim, çiftleşme, mutasyon). Her çiftleşme ve mutasyon için her seferinde yeni ebeveynler seçilir. Popülasyonu bir sonraki döneme hazırlamak.
- En iyi yavruların genlerinin referans kromozomun genleriyle karşılaştırılması. En iyi yavrunun kromozomu referans kromozomdan daha iyiyse referans kromozomu değiştirin.
Belirli bir dönem sayısı için, referans olanlardan daha iyi olan kromozom kalmayana kadar paragraf 5'ten devam edin.
3.1. Genel değişkenler. Genel değişkenler
Genel düzeyde aşağıdaki değişkenler belirtildi:
//----------------------Global variables----------------------------- double Chromosome[]; //A set of optimized arguments of the function - genes //(for example: the weight of the neuron network, etc.)- of the chromosome int ChromosomeCount =0; //Maximum possible amount of chromosomes in a colony int TotalOfChromosomesInHistory=0;//Total number of chromosomes in the history int ChrCountInHistory =0; //Number of unique chromosomes in the base chromosome int GeneCount =0; //Number of genes in the chromosome double RangeMinimum =0.0;//Minimum search range double RangeMaximum =0.0;//Maximum search range double Precision =0.0;//Search step int OptimizeMethod =0; //1-minimum, any other - maximum int FFNormalizeDigits =0; //Number of symbols after the comma in the fitness value int GeneNormalizeDigits =0; //Number of symbols after the comma in the genes value double Population [][1000]; //Population double Colony [][500]; //Offspring colony int PopulChromosCount =0; //The current number of chromosomes in a population int Epoch =0; //Number of epochs without progress int AmountStartsFF=0; //Number of launches of the fitness function //————————————————————————————————————————————————————————————————————————
3.2. UGA. GA'nın ana işlevi
Aslında, programın gövdesinden çağrılan GA'nın ana işlevi yukarıda listelenen adımları gerçekleştirmektir; bu nedenle bununla ilgili olarak fazla ayrıntıya girmeyeceğiz.
Algoritmanın tamamlanmasının ardından, aşağıdaki bilgiler günlüğe kaydedildi:
- Burada toplamda kaç dönem olduğu (jenerasyonlar).
- Toplamda kaç hata olduğu.
- Benzersiz kromozom sayısı.
- FF'nin toplam başlatma sayısı.
- Geçmişteki toplam kromozom sayısı.
- Geçmişteki toplam kromozom sayısına kopyaların yüzde oranı.
- En iyi sonuç.
"Benzersiz kromozom sayısı" ve "FF'nin toplam başlatma sayısı" - aynı boyutlar, ancak farklı şekilde hesaplanır. Kontrol için kullanın.
//———————————————————————————————————————————————————————————————————————— //Basic function UGA void UGA ( double ReplicationPortion, // Proportion of replication. double NMutationPortion, // Proportion of natural mutations. double ArtificialMutation, // Proportion of artificial mutations. double GenoMergingPortion, // Proportion of borrowed genes. double CrossingOverPortion, // Proportion of cross -over. //--- double ReplicationOffset, // Coefficient of displacement of interval borders double NMutationProbability // Probability of mutation of each gene in% ) { //generator reset takes place only once MathSrand((int)TimeLocal()); //-----------------------Variables------------------------------------- int chromos=0, gene =0;//indexes of chromosomes and genes int resetCounterFF =0;//counter of resets of "Epochs without progress" int currentEpoch =1;//number of the current epoch int SumOfCurrentEpoch=0;//sum of "Epochs without progress" int MinOfCurrentEpoch=Epoch;//minimum of "epochs without progress" int MaxOfCurrentEpoch=0;//maximum of "Epochs without progress" int epochGlob =0;//total number of epochs // Colony [number of traits(genes)][number of individuals in a colony] ArrayResize(Population,GeneCount+1); ArrayInitialize(Population,0.0); // Colony of offspring [number of traits(genes)][number of individuals in a colony] ArrayResize(Colony,GeneCount+1); ArrayInitialize(Colony,0.0); // Chromosome bank // [number of traits (genes)][number of chromosomes in the bank] double historyHromosomes[][100000]; ArrayResize(historyHromosomes,GeneCount+1); ArrayInitialize(historyHromosomes,0.0); //---------------------------------------------------------------------- //--------------Verification of the correctness of incoming parameters---------------- //...the number of chromosomes mus be less than 2 if(ChromosomeCount<=1) ChromosomeCount=2; if(ChromosomeCount>500) ChromosomeCount=500; //---------------------------------------------------------------------- //====================================================================== // 1) Create a proto- population —————1) ProtopopulationBuilding(); //====================================================================== // 2) Determine the fitness of each individual —————2) //For the 1st colony for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=0;chromos<ChromosomeCount;chromos++) Population[0][chromos]=Colony[0][chromos]; //For the 2nd colony for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) for(gene=1;gene<=GeneCount;gene++) Colony[gene][chromos-ChromosomeCount]=Population[gene][chromos]; GetFitness(historyHromosomes); for(chromos=ChromosomeCount;chromos<ChromosomeCount*2;chromos++) Population[0][chromos]=Colony[0][chromos-ChromosomeCount]; //====================================================================== // 3) Prepare the population for reproduction ————3) RemovalDuplicates(); //====================================================================== // 4) Extract the reference chromosome —————4) for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; //====================================================================== ServiceFunction(); //The main cycle The main cycle of the genetic algorithm from 5 to 6 while(currentEpoch<=Epoch) { //==================================================================== // 5) Operators of UGA —————5) CycleOfOperators ( historyHromosomes, //--- ReplicationPortion, //Proportion of replication. NMutationPortion, //Proportion of natural mutation. ArtificialMutation, //Proportion of artificial mutation. GenoMergingPortion, //Proportion of borrowed genes. CrossingOverPortion,//Proportion of cross- over. //--- ReplicationOffset, //Coefficient of displacement of interval borders NMutationProbability//Probability of mutation of each gene in % ); //==================================================================== // 6) Compare the genes of the best offspring with the genes of the reference chromosome. // If the chromosome of the best offspring is better that the reference chromosome, // replace the reference. —————6) //If the optimization mode is - minimization if(OptimizeMethod==1) { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]<Chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Rest the counter of "epochs without progress" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //If the optimization mode is - minimization else { //If the best chromosome of the population is better than the reference chromosome if(Population[0][0]>Chromosome[0]) { //Replace the reference chromosome for(gene=0;gene<=GeneCount;gene++) Chromosome[gene]=Population[gene][0]; ServiceFunction(); //Reset the counter of "epochs without progress" if(currentEpoch<MinOfCurrentEpoch) MinOfCurrentEpoch=currentEpoch; if(currentEpoch>MaxOfCurrentEpoch) MaxOfCurrentEpoch=currentEpoch; SumOfCurrentEpoch+=currentEpoch; currentEpoch=1; resetCounterFF++; } else currentEpoch++; } //==================================================================== //Another epoch went by.... epochGlob++; } Print("Epochs went by=",epochGlob," Number of resets=",resetCounterFF); Print("MinOfCurrentEpoch",MinOfCurrentEpoch, " AverageOfCurrentEpoch",NormalizeDouble((double)SumOfCurrentEpoch/(double)resetCounterFF,2), " MaxOfCurrentEpoch",MaxOfCurrentEpoch); Print(ChrCountInHistory," - Unique chromosome"); Print(AmountStartsFF," - Total number of launches of FF"); Print(TotalOfChromosomesInHistory," - Total number of chromosomes in the history"); Print(NormalizeDouble(100.0-((double)ChrCountInHistory*100.0/(double)TotalOfChromosomesInHistory),2),"% of duplicates"); Print(Chromosome[0]," - best result"); } //————————————————————————————————————————————————————————————————————————
3.3. Bir Proto- popülasyon oluşturmak. Bir proto- popülasyon oluşturmak.
Optimizasyon problemlerinin çoğunda, işlev argümanlarının sayı doğrusunda nerede bulunduğunu önceden bilmenin bir yolu olmadığı için en iyi optimum seçenek, belirli bir aralıkta rastgele oluşturmadır.
//———————————————————————————————————————————————————————————————————————— //Creating a proto- population void ProtopopulationBuilding() { PopulChromosCount=ChromosomeCount*2; //Fill up the population with chromosomes with random //...genes in the range between RangeMinimum...RangeMaximum for(int chromos=0;chromos<PopulChromosCount;chromos++) { //beginning with the 1st indexes (the 0 index is reserved for VFF) for(int gene=1;gene<=GeneCount;gene++) Population[gene][chromos]= NormalizeDouble(SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); TotalOfChromosomesInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.4. GetFitness. Uygunluk sağlamak
Her kromozom için optimize edilmiş işlevi sırayla gerçekleştirir.
//------------------------------------------------ ------------------------ // Getting the fitness for each individual. void GetFitness ( double &historyHromosomes[][100000] ) { for(int chromos=0;chromos<ChromosomeCount;chromos++) CheckHistoryChromosomes(chromos,historyHromosomes); } //————————————————————————————————————————————————————————————————————————
3.5. CheckHistoryChromosomes. Kromozom tabanı aracılığıyla kromozomun doğrulanması
Her bireyin kromozomu taban üzerinden doğrulanır: Bunun için FF'nin hesaplanıp hesaplanmadığına bakılır ve varsa, hazır VFF tabandan alınır; yoksa, bunun için FF çağrılır. Böylece, FF'nin yinelenen kaynak açısından yoğun hesaplamalar hariç tutulur.
//———————————————————————————————————————————————————————————————————————— //Verification of chromosome through the chromosome base. void CheckHistoryChromosomes ( int chromos, double &historyHromosomes[][100000] ) { //-----------------------Variables------------------------------------- int Ch1=0; //Index of the chromosome from the base int Ge =0; //Index of the gene int cnt=0; //Counter of unique genes. If at least one gene is different //- the chromosome is acknowledged unique //---------------------------------------------------------------------- //If at least one chromosome is stored in the base if(ChrCountInHistory>0) { //Enumerate the chromosomes in the base to find an identical one for(Ch1=0;Ch1<ChrCountInHistory && cnt<GeneCount;Ch1++) { cnt=0; //Compare the genes, while the gene index is less than the number of genes and while there are identical genes for(Ge=1;Ge<=GeneCount;Ge++) { if(Colony[Ge][chromos]!=historyHromosomes[Ge][Ch1]) break; cnt++; } } //If there are enough identical genes then we can use a ready- made solution from the base if(cnt==GeneCount) Colony[0][chromos]=historyHromosomes[0][Ch1-1]; //If there is no such chromosome, then we calculate the FF for it... else { FitnessFunction(chromos); //.. and if there is space in the base, then save it if(ChrCountInHistory<100000) { for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } } //If the base is empty, calculate the FF for it and save it in the base else { FitnessFunction(chromos); for(Ge=0;Ge<=GeneCount;Ge++) historyHromosomes[Ge][ChrCountInHistory]=Colony[Ge][chromos]; ChrCountInHistory++; } } //————————————————————————————————————————————————————————————————————————
3.6. CycleOfOperators. UGA'da İşleç Döngüsü
Bu noktada, tüm bir yapay yaşam döneminin edebi kaderine karar veriliyor; yeni bir jenerasyon doğuyor ve ölüyor. Bu şu şekilde olur: Çiftleşmek için iki ebeveyn seçilir veya biri onun üzerinde bir mutasyon eylemi gerçekleştirir. GA'nın her işleci için uygun bir parametre belirlenir. Sonuç olarak, bir yavru elde ederiz. Bu, soyundan gelen koloni tamamen dolana kadar defalarca tekrarlanır. Ardından, soyundan gelenlerin kolonisi, her bireyin kendini olabildiğince iyi gösterebilmesi için habitata bırakılır ve VFF'yi hesaplarız.
"Ateş, su ve bakır borular" ile test edildikten sonra, soyundan gelen koloni popülasyona yerleşir. Yapay evrimde bir sonraki adım, gelecek jenerasyonlarda "kan"ın tükenmesini ve ardından yenilenen popülasyonun uygunluk derecesine göre sıralanmasını önlemek için klonlar kutsal öldürme işlemine tabi tutulacaktır.
//———————————————————————————————————————————————————————————————————————— //Cycle of operators of UGA void CycleOfOperators ( double &historyHromosomes[][100000], //--- double ReplicationPortion, //Proportion of replications. double NMutationPortion, //Proportion of natural mutations. double ArtificialMutation, //Proportion of artificial mutations. double GenoMergingPortion, //Portion of borrowed genes. double CrossingOverPortion,//Proportion of cross-over. //--- double ReplicationOffset, //Coefficient of displacement of interval borders double NMutationProbability//Probability of mutation of each gene in % ) { //-----------------------Variables------------------------------------- double child[]; ArrayResize (child,GeneCount+1); ArrayInitialize(child,0.0); int gene=0,chromos=0, border=0; int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,6); ArrayInitialize(fit,0.0); //Counter of planting spots in a new population. int T=0; //---------------------------------------------------------------------- //Set a portion of operators of UGA double portion[6]; portion[0]=ReplicationPortion; //Proportion of replications. portion[1]=NMutationPortion; //Proportion of natural mutations. portion[2]=ArtificialMutation; //Proportion of artificial mutations. portion[3]=GenoMergingPortion; //Proportion of borrowed genes. portion[4]=CrossingOverPortion;//Proportion of cross- overs. portion[5]=0.0; //------------------------Cycle of operators of UGA--------- //Fill up the new colony with offspring while(T<ChromosomeCount) { //============================ for(i=0;i<6;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(portion[i]-portion[5]); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[4][1]); for(u=0;u<5;u++) { if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; } //============================ switch(u) { //--------------------- case 0: //------------------------Replication-------------------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { Replication(child,ReplicationOffset); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 1: //---------------------Natural mutation------------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { NaturalMutation(child,NMutationProbability); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast- forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 2: //----------------------Artificial mutation----------------------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { ArtificialMutation(child,ReplicationOffset); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast-forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- case 3: //-------------The creation of an individual with borrowed genes----------- //If there is space in the new colony, create a new individual if(T<ChromosomeCount) { GenoMerging(child); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One space is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- default: //---------------------------Crossing-Over--------------------------- //If there is place in the new colony, create a new individual if(T<ChromosomeCount) { CrossingOver(child); //Settle the new individual into the new colony for(gene=1;gene<=GeneCount;gene++) Colony[gene][T]=child[gene]; //One place is occupied, fast forward the counter T++; TotalOfChromosomesInHistory++; } //--------------------------------------------------------------- break; //--------------------- } }//End of the cycle operators of UGA-- //Determine the fitness of each individual in the colony of offspring GetFitness(historyHromosomes); //Settle the offspring into the main population if(PopulChromosCount>=ChromosomeCount) { border=ChromosomeCount; PopulChromosCount=ChromosomeCount*2; } else { border=PopulChromosCount; PopulChromosCount+=ChromosomeCount; } for(chromos=0;chromos<ChromosomeCount;chromos++) for(gene=0;gene<=GeneCount;gene++) Population[gene][chromos+border]=Colony[gene][chromos]; //Prepare the population for the next reproduction RemovalDuplicates(); }//the end of the function //————————————————————————————————————————————————————————————————————————
3.7. Replikasyon. Replikasyon
Operatör, biyolojide DNA replikasyonu olarak adlandırılan doğal fenomene en yakın olandır, ancak bu, özünde aynı şey değildir. Ancak doğada buna daha yakın bir eşdeğer bulamadığım için bu adı kullanmaya devam etmeye karar verdim.
Replikasyon, ebeveyn kromozomlarının özelliklerini aktarırken yeni genler üreten en önemli genetik operatördür. Algoritmanın yakınsamasını sağlayan ana operatör.. GA, diğer operatörler kullanılmadan yalnızca onunla çalışabilir ama bu durumda FF başlatmalarının sayısı çok daha fazla olacaktır.
Replikasyon operatörünün prensibini göz önünde bulundurun. İki ebeveyn kromozomu kullanıyoruz. Yeni yavru gen
[C1-((C2-C1)*ReplicationOffset),C2+((C2-C1)*ReplicationOffset)] aralığında rastgele bir sayıdır.
Burada, C1 ve C2 ebeveyn genleri ReplicationOffset - Aralık sınırlarının yer değiştirme katsayısı [C1, C2] .
Örneğin, baba bireyden (mavi) ve anne bireyden (pembe) bir çocuk (yeşil) oluşturulabilir:
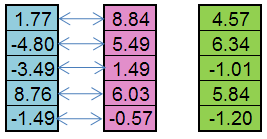
Şekil 5. Replikasyon operatörünün çalışma prensibi
Grafiksel olarak, yavru genin olasılığı şu şekilde özetlenebilir:
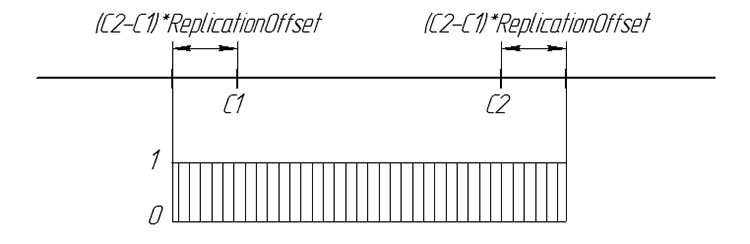
Şekil 6. Yavru genin bir sayı doğrusu üzerinde ortaya çıkma olasılığı
Diğer yavru genler de aynı şekilde üretilir.
//------------------------------------------------ ------------------------ // Replication void Replication ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- SelectTwoParents(address_mama,address_papa); //-------------------Cycle of gene enumeration-------------------------------- for(int i=1;i<=GeneCount;i++) { //----figure out where the father and mother came from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search had not gone over the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if we С1>C2, swi if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of the created gene Minimum = C1-((C2-C1)*ReplicationOffset); Maximum = C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search has not gone over the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- temp=RNDfromCI(Minimum,Maximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } //————————————————————————————————————————————————————————————————————————3.8. NaturalMutation. Doğal mutasyon
Mutasyonlar, süreçler boyunca canlı hücrelerde sürekli olarak meydana gelirler ve doğal seleksiyon için malzeme görevi görürler. Organizmanın tüm yaşamı boyunca normal habitat koşullarında, 10 ^ 10 hücre nesli başına bir sıklıkta kendiliğinden ortaya çıkarlar.
Biz meraklı araştırmacılar, doğal düzene uymak ve genin bir sonraki mutasyonunu bu kadar uzun süre beklemek zorunda değiliz. Yüzde olarak ifade edilen ve kromozomdaki her bir gen için mutasyon olasılığını belirleyen NMutationProbability parametresi bunu yapmamıza yardımcı olacaktır.
NaturalMutation işlecinde mutasyon, [RangeMinimum, RangeMaximum] aralığında rastgele bir genin üretilmesinden oluşur. NMutationProbability = %100, kromozomdaki tüm genlerin %100 mutasyonu ve NMutationProbability = %0 mutasyonların tamamen yokluğu anlamına gelir. En son seçeneğin pratik problemlerde kullanımı uygun değildir.
//------------------------------------------------ ------------------------ // The natural mutation. void NaturalMutation ( double &child[], double NMutationProbability ) { //-----------------------Variables------------------------------------- int address=0; double prob=0.0; //---------------------------------------------------------------------- if(NMutationProbability<0.0) prob=0.0; if(NMutationProbability>100.0) prob=100.0; //-----------------Parent selection------------------------ SelectOneParent(address); //--------------------------------------- for(int i=1;i<=GeneCount;i++) if(RNDfromCI(0.0,100.0)<prob) child[i]=NormalizeDouble( SelectInDiscreteSpace(RNDfromCI(RangeMinimum,RangeMaximum),RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits ); } //————————————————————————————————————————————————————————————————————————
3.9. ArtificialMutation. Yapay mutasyon
Operatörün ana görevi "taze" kanın üretilmesidir. İki ebeveyn kullanıyoruz ve yavrunun genleri, sayı doğrusundaki ana genler tarafından ayrılmamış boşluklardan seçiliyor. GA'yı yerel ekstremumlardan birine takılmaktan korur. Diğer operatörlere kıyasla daha büyük bir oranda, yakınsamayı hızlandırır; aksi taktirde yavaşlayarak, FF'nin başlatma sayısını artırır.
Replikasyonda olduğu gibi, iki ebeveyn kromozomu kullanıyoruz. Ancak ArtificialMutation operatörünün görevi, ebeveyn özelliklerini yavrulara aktarmak değil, çocuğu onlardan farklı kılmaktır. Bu nedenle, tam tersi olarak, aynı aralık sınırı yer değiştirme katsayısı kullanılarak, ancak genler, Replikasyon tarafından alınacak olan aralığın dışında üretilir. Yavrunun yeni geni, [RangeMinimum, C1-(C2-C1) * ReplicationOffset] ve [C2 + (C2-C1) * ReplicationOffset, RangeMaximum] aralıklarından rastgele bir sayıdır.
Grafiksel olarak, yavrudaki ReplicationOffset = 0,25 bir genin olasılığı şu şekilde temsil edilebilir:
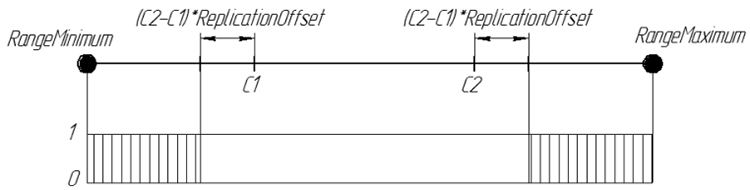
Şekil 7. Gerçek çizgi aralığında [RangeMinimum; RangeMaximum] bir genin soyundan gelen ReplicationOffset'teki olasılığı = 0,25
//———————————————————————————————————————————————————————————————————————— //Artificial mutation. void ArtificialMutation ( double &child[], double ReplicationOffset ) { //-----------------------Variables------------------------------------- double C1=0.0,C2=0.0,temp=0.0,Maximum=0.0,Minimum=0.0,p=0.0; int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //-------------------Cycle of genes enumeration------------------------------ for(int i=1;i<=GeneCount;i++) { //----determine where the mother and father are from -------- C1 = Population[i][address_mama]; C2 = Population[i][address_papa]; //------------------------------------------ //Mandatory verification to make sure that the search doesn't go beyond the specified range if(C1 < RangeMinimum) C1 = RangeMinimum; if(C1 > RangeMaximum) C1 = RangeMaximum; if(C2 < RangeMinimum) C2 = RangeMinimum; if(C2 > RangeMaximum) C2 = RangeMaximum; //------------------------------------------------------------------ //....determine the largest and smallest of them, //if С1>C2, we change their places if(C1>C2) { temp=C1; C1=C2; C2=temp; } //-------------------------------------------- //Specify the borders of creating the new gene Minimum=C1-((C2-C1)*ReplicationOffset); Maximum=C2+((C2-C1)*ReplicationOffset); //-------------------------------------------- //Mandatory verification to make sure that the search doesn't go beyond the specified range if(Minimum < RangeMinimum) Minimum = RangeMinimum; if(Maximum > RangeMaximum) Maximum = RangeMaximum; //--------------------------------------------------------------- p=MathRand(); if(p<16383.5) { temp=RNDfromCI(RangeMinimum,Minimum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } else { temp=RNDfromCI(Maximum,RangeMaximum); child[i]= NormalizeDouble(SelectInDiscreteSpace(temp,RangeMinimum,RangeMaximum,Precision,3),GeneNormalizeDigits); } } } //————————————————————————————————————————————————————————————————————————
3.10 GenoMerging. Ödünç alınan genler
Verilen GA operatörünün doğal bir eşdeğeri yoktur. Aslında bu harika mekanizmanın canlı organizmalarda nasıl işleyeceğini hayal etmesi zor. Ancak, bu, genleri çok sayıda ebeveynden (ebeveyn sayısı gen sayısına eşittir) yavrulara aktarma konusunda dikkate değer bir özelliğe sahiptir. Operatör yeni genler üretmez ve bir kombinatoryal arama mekanizmasıdır.
Bu, şu şekilde işler: İlk yavru gen için bir ebeveyn seçilir ve birinci gen ondan alınır, ardından ikinci gen için ikinci bir ebeveyn seçilir ve gen ondan alınır vb. Gen sayısı birden fazla ise uygulanması tavsiye edilir. Aksi takdirde, operatör kromozomların kopyalarını oluşturacağı için bu, devre dışı bırakılmalıdır.
//———————————————————————————————————————————————————————————————————————— //Borrowing genes. void GenoMerging ( double &child[] ) { //-----------------------Variables------------------------------------- int address=0; //---------------------------------------------------------------------- for(int i=1;i<=GeneCount;i++) { //-----------------Selecting parents------------------------ SelectOneParent(address); //-------------------------------------------------------- child[i]=Population[i][address]; } } //————————————————————————————————————————————————————————————————————————
3.11. CrossingOver. Çaprazlama
Çaprazlama (biyolojide melezleme olarak da bilinir) kromozom bölümlerinin değiş tokuş edilmesi olgusudur. GenoMerging'de olduğu gibi, bu bir kombinatoryal arama mekanizmasıdır.
İki ebeveyn kromozomu seçilir. Her ikisi de rastgele bir yerde "kesilir". Yavrunun kromozomu, ebeveyn kromozomlarının parçalarından oluşacaktır.
Bu mekanizmayı bir resimde göstermek en kolay yoldur:
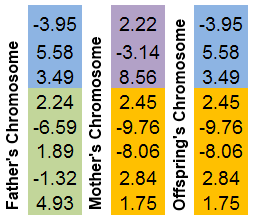
Şekil 8. Kromozom parçalarının değişim mekanizması
Gen sayısı birden fazla ise uygulanması tavsiye edilir. Aksi takdirde, operatör kromozomların kopyalarını oluşturacağı için bu, devre dışı bırakılmalıdır.
//———————————————————————————————————————————————————————————————————————— //Crossing-over. void CrossingOver ( double &child[] ) { //-----------------------Variables------------------------------------- int address_mama=0,address_papa=0; //---------------------------------------------------------------------- //-----------------Selecting parents------------------------ SelectTwoParents(address_mama,address_papa); //-------------------------------------------------------- //Determine the breakage point int address_of_gene=(int)MathFloor((GeneCount-1)*(MathRand()/32767.5)); for(int i=1;i<=GeneCount;i++) { //----copy the mother's genes-------- if(i<=address_of_gene+1) child[i]=Population[i][address_mama]; //----copy the father's genes-------- else child[i]=Population[i][address_papa]; } } //————————————————————————————————————————————————————————————————————————
3.12. SelectTwoParents. İki ebeveyn seçimi
Gen havuzunun tükenmesini önlemek için kendi kendisiyle melezleme yasağı vardır. Farklı ebeveynler bulmak için on girişimde bulunulur ve bir çift bulamazsak, kendi kendine melezlenmesine izin veririz. Temel olarak, aynı numunenin bir kopyasını elde ederiz.
Bir yandan, bireyleri klonlama olasılığı azalırken, diğer yandan makul sayıda adımda (farklı ebeveynler seçimi) bunu yapmanın neredeyse imkansız olacağı bir durum ortaya çıkabileceği için aramanın döngüselliği önlenir.
Replication, ArtificialMutation ve CrossingOver operatörlerinde kullanılır.
//———————————————————————————————————————————————————————————————————————— //Selection of two parents. void SelectTwoParents ( int &address_mama, int &address_papa ) { //-----------------------Variables------------------------------------- int cnt=1; address_mama=0;//address of the mother individual in a population address_papa=0;//address of the father individual in a population //---------------------------------------------------------------------- //----------------------------Selection of parents-------------------------- //Ten attempts to chose different parents. while(cnt<=10) { //For the mother individual address_mama=NaturalSelection(); //For the father individual address_papa=NaturalSelection(); if(address_mama!=address_papa) break; } //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.13. SelectOneParent. Bir ebeveyn seçimi
Burada her şey basittir; popülasyondan bir ebeveyn seçilir.
NaturalMutation ve GenoMerging operatörlerinde kullanılır.
//———————————————————————————————————————————————————————————————————————— //Selection of one parent. void SelectOneParent ( int &address//address of the parent individual in the population ) { //-----------------------Variables------------------------------------- address=0; //---------------------------------------------------------------------- //----------------------------Selecting a parent-------------------------- address=NaturalSelection(); //--------------------------------------------------------------------- } //————————————————————————————————————————————————————————————————————————
3.14. NaturalSelection. Doğal seleksiyon
Doğal seleksiyon - Bu, yararlı kalıtsal özelliklere sahip olan ve çevresel koşullara daha iyi uyum sağlayan bireylerin hayatta kalmasına ve tercihli üremesine yol açan süreçtir.
Operatör, geleneksel "Rulet" operatörüne benzer ( Rulet çarkı seçimi - Ruletin n "başlatması" olan bireylerin seçimi. Rulet çarkı, popülasyonun her bir üyesi için bir bölge içerir. i-th bölgesinin boyutu, karşılık gelen uygunluk değeriyle orantılıdır), ancak önemli farklılıkları vardır. Bireylerin konumunu en çok ve en az uygun olanlara göre dikkate alır. Ayrıca, en kötü genlere sahip bir birey dahi geride bir yavru bırakma şansına sahiptir. Bu adil, değil mi? Her ne kadar adaletle ilgili olmasa da, bu, doğada tüm bireylerin geride yavru bırakma fırsatına sahip olduğu gerçeğiyle ilgilidir.
Örneğin, maksimizasyon probleminde aşağıdaki VFF'ye sahip 10 kişi alın: 256, 128, 64, 32, 16, 8, 4, 2, 0, -1 - Burada daha büyük değerler daha iyi uygunluk anlamına gelir. Bu örnek, komşu bireyler arasındaki "mesafenin" önceki iki birey arasındakinden 2 kat daha büyük olduğunu görebilmemiz için verilmiştir. Ancak, pasta grafikte, her bireyin bir yavru bırakma olasılığı aşağıdaki gibidir:
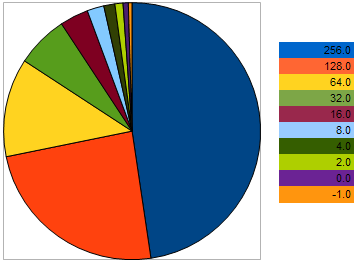
Şekil 9. Ebeveyn bireyleri seçme olasılığı grafiği
Bireylerin en kötüye yaklaşmasıyla şanslarının daha da kötüleştiğini gösterir. Buna karşılık birey daha iyi örneğe ne kadar yaklaşırsa üreme şansı o kadar iyi olur.

Şekil 10. Ebeveyn bireyleri seçme olasılığı grafiği
//———————————————————————————————————————————————————————————————————————— //Natural selection. int NaturalSelection() { //-----------------------Variables------------------------------------- int i=0,u=0; double p=0.0,start=0.0; double fit[][2]; ArrayResize (fit,PopulChromosCount); ArrayInitialize(fit,0.0); double delta=(Population[0][0]-Population[0][PopulChromosCount-1])*0.01-Population[0][PopulChromosCount-1]; //---------------------------------------------------------------------- for(i=0;i<PopulChromosCount;i++) { fit[i][0]=start; fit[i][1]=start+MathAbs(Population[0][i]+delta); start=fit[i][1]; } p=RNDfromCI(fit[0][0],fit[PopulChromosCount-1][1]); for(u=0;u<PopulChromosCount;u++) if((fit[u][0]<=p && p<fit[u][1]) || p==fit[u][1]) break; return(u); } //————————————————————————————————————————————————————————————————————————
3.15. RemovalDuplicates. Kopyaları çıkarma
İşlev, popülasyondaki çift kromozomları kaldırır ve kalan benzersiz kromozomlar (mevcut dönemin popülasyonuna özgü), optimizasyon türüne göre belirlenen VFF'ye göre sıralanır (yani azalan veya artan sırada).
//———————————————————————————————————————————————————————————————————————— //Removing duplicates sorted by VFF void RemovalDuplicates() { //-----------------------Variables------------------------------------- int chromosomeUnique[1000];//Array stores the unique trait //of each chromosome: 0-duplicate, 1-unique ArrayInitialize(chromosomeUnique,1); //Assume that there are no duplicates double PopulationTemp[][1000]; ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Ge =0; //Index of the gene int Ch =0; //Index of the chromosome int Ch2=0; //Index of the second chromosome int cnt=0; //Counter //---------------------------------------------------------------------- //----------------------Remove duplicates---------------------------1 //Chose the first from the pair for comparison... for(Ch=0;Ch<PopulChromosCount;Ch++) { //If it's not a duplicate... if(chromosomeUnique[Ch]!=0) { //Chose the second from the pair... for(Ch2=0;Ch2<PopulChromosCount;Ch2++) { if(Ch!=Ch2 && chromosomeUnique[Ch2]!=0) { //Zeroize the counter of identical genes cnt=0; //Compare the genes. while there are identical genes present for(Ge=1;Ge<=GeneCount;Ge++) { if(Population[Ge][Ch]!=Population[Ge][Ch2]) break; else cnt++; } //If there are the same amount of identical genes as total genes //..the chromosome is considered a duplicate if(cnt==GeneCount) chromosomeUnique[Ch2]=0; } } } } //The counter calculates the number of unique chromosomes cnt=0; //Copy the unique chromosomes into a temporary array for(Ch=0;Ch<PopulChromosCount;Ch++) { //If the chromosome is unique, copy it, if not, go to the next if(chromosomeUnique[Ch]==1) { for(Ge=0;Ge<=GeneCount;Ge++) PopulationTemp[Ge][cnt]=Population[Ge][Ch]; cnt++; } } //Assigning the variable "All chromosomes" the value of counter of unique chromosomes PopulChromosCount=cnt; //Return unique chromosomes back to the array for temporary storage //..of combined populations for(Ch=0;Ch<PopulChromosCount;Ch++) for(Ge=0;Ge<=GeneCount;Ge++) Population[Ge][Ch]=PopulationTemp[Ge][Ch]; //=================================================================1 //----------------Ranking the population---------------------------2 PopulationRanking(); //=================================================================2 } //————————————————————————————————————————————————————————————————————————
3.16. PopulationRanking. Popülasyonun sıralanması
Sıralama, VFF tarafından yapılır. Yöntem, ' kabarcıklı yönteme benzer (Algoritma, sıralanmış dizi boyunca tekrarlanan geçişlerden oluşur. Her geçiş için, öğeler sırayla ikili olarak karşılaştırılır ve bir çiftin sırası yanlışsa, bir öğe değişimi gerçekleşir. Diziden geçişler, geçişlerden biri artık değiş tokuşlara gerek olmadığını gösterene kadar tekrarlanır; yani dizi sıralanmıştır.
Algoritmadan geçerken, yerinde olmayan bir öğe, tıpkı sudaki bir kabarcık gibi istenen konuma "açılır"; bu nedenle algoritmanın adı, ancak bir fark vardır - dizinin içeriği değil yalnızca dizinin indeksleri sıralanır. Bu yöntem daha hızlıdır ve bir dizinin diğerine kopyalanmasından biraz farklıdır. Ve sıralanan dizinin boyutu ne kadar büyükse, fark o kadar az olur.
//———————————————————————————————————————————————————————————————————————— //Population ranking. void PopulationRanking() { //-----------------------Variables------------------------------------- int cnt=1, i = 0, u = 0; double PopulationTemp[][1000]; //Temporary population ArrayResize (PopulationTemp,GeneCount+1); ArrayInitialize(PopulationTemp,0.0); int Indexes[]; //Indexes of chromosomes ArrayResize (Indexes,PopulChromosCount); ArrayInitialize(Indexes,0); int t0=0; double ValueOnIndexes[]; //VFF of corresponding //..chromosome indexes ArrayResize (ValueOnIndexes,PopulChromosCount); ArrayInitialize(ValueOnIndexes,0.0); double t1=0.0; //---------------------------------------------------------------------- //Fill in the indexes in the temporary array temp2 and //...copy the first line from the sorted array for(i=0;i<PopulChromosCount;i++) { Indexes[i] = i; ValueOnIndexes[i] = Population[0][i]; } if(OptimizeMethod==1) { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]>ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } else { while(cnt>0) { cnt=0; for(i=0;i<PopulChromosCount-1;i++) { if(ValueOnIndexes[i]<ValueOnIndexes[i+1]) { //----------------------- t0 = Indexes[i+1]; t1 = ValueOnIndexes[i+1]; Indexes [i+1] = Indexes[i]; ValueOnIndexes [i+1] = ValueOnIndexes[i]; Indexes [i] = t0; ValueOnIndexes [i] = t1; //----------------------- cnt++; } } } } //Create a sorted-out array based on the obtained indexes for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) PopulationTemp[i][u]=Population[i][Indexes[u]]; //Copy the sorted-out array back for(i=0;i<GeneCount+1;i++) for(u=0;u<PopulChromosCount;u++) Population[i][u]=PopulationTemp[i][u]; } //————————————————————————————————————————————————————————————————————————
3.17. RNDfromCustomInterval. Belirli bir aralıktaki rasgele sayıların oluşturucusu
Kullanışlı bir özelliktir. UGA'da kullanışlıdır.
//———————————————————————————————————————————————————————————————————————— //Generator of random numbers from the selected interval. double RNDfromCI(double RangeMinimum,double RangeMaximum) { return(RangeMinimum+((RangeMaximum-RangeMinimum)*MathRand()/32767.5));} //————————————————————————————————————————————————————————————————————————
3.18. SelectInDiscreteSpace. Ayrık uzayda seçim
Arama alanını daraltmak için kullanılır. adım = 0,0 parametresi ile arama sürekli bir alanda gerçekleştirilir (dil sınırlamalarıyla sınırlıdır, MQL'de 15. önemli sembol dahil). GA algoritmasını daha doğru kullanmak için uzun sayılarla çalışmak üzere ek bir kitaplık yazmanız gerekir.
RoundMode = 1'deki işlevin çalışması aşağıdaki şekil ile gösterilebilir:
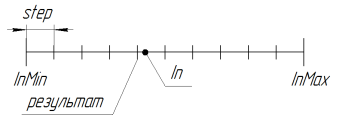
Şekil 11. SelectInDiscreteSpace işlevinin RoundMode = 1 pozisyonunda çalışması
//———————————————————————————————————————————————————————————————————————— //Selection in discrete space. //Modes: //1-closest below //2-closest above //any closest double SelectInDiscreteSpace ( double In, double InMin, double InMax, double step, int RoundMode ) { if(step==0.0) return(In); // secure the correctness of borders if( InMax < InMin ) { double temp = InMax; InMax = InMin; InMin = temp; } // during a breach - return the breached border if( In < InMin ) return( InMin ); if( In > InMax ) return( InMax ); if( InMax == InMin || step <= 0.0 ) return( InMin ); // bring to the specified scale step = (InMax - InMin) / MathCeil ( (InMax - InMin) / step ); switch( RoundMode ) { case 1: return( InMin + step * MathFloor ( ( In - InMin ) / step ) ); case 2: return( InMin + step * MathCeil ( ( In - InMin ) / step ) ); default: return( InMin + step * MathRound ( ( In - InMin ) / step ) ); } } //————————————————————————————————————————————————————————————————————————
3.19. FitnessFunction. Uygunluk işlevi
GA'nın bir parçası değildir. İşlev, VFF'nin hesaplanacağı popülasyondaki kromozomun indeksini alır. VFF, iletilen kromozomun sıfır indeksine yazılır. Bu işlevin kodu, her görev için benzersizdir.
3.20. ServiceFunction. Hizmet işlevi
GA'nın bir parçası değildir. Bu işlevin kodu, her belirli görev için benzersizdir. Dönemler üzerinde kontrol sağlamak için kullanılabilir. Örneğin, mevcut dönem için en iyi VFF'yi görüntülemek için.
4. UGA çalışmasına ilişkin örnekler
Optimizasyon sorunlarının tümü EA aracılığıyla çözülür ve iki türe ayrılır:
- Genotip, bir fenotiple tutarlıdır. Kromozom genlerinin değerleri, bir optimizasyon işlevinin argümanları tarafından doğrudan atanır. Örnek 1.
- Genotip fenotiple eşleşmiyor. Optimize edilmiş işlevi hesaplamak için kromozom genlerinin anlamının yorumlanması gerekir. Örnek 2.
4.1. Örnek 1
Algoritmanın çalıştığından emin olmak için sorunu bilinen bir yanıtla birlikte düşünün ve ardından çözümü birçok yatırımcının ilgisini çeken sorunu çözmeye devam edin.
Sorun: Minimum ve maksimum "Dış Görünüm" işlevini bulun:
segment [-5, 5].
Yanıt: fmin (3.07021,3.315935) = -4.3182, fmax (-3.315699; -3.072485) = 14.0606.
Şekil 12. [-5, 5] segmentindeki "Dış Görünüm" grafiği
Sorunu çözmek için aşağıdaki script dosyasını yazıyoruz:
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" #include "Skin.mqh"//testing function //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Gene pool parameter----"; input int ChromosomeCount_P = 50; //Number of chromosomes in a colony input int GeneCount_P = 2; //Number of genes input int FFNormalizeDigits_P = 4; //Number of fitness symbols input int GeneNormalizeDigits_P = 4; //Number of genes input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Operator parameters----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing-over. //--- input double ReplicationOffset_P = 0.5; //Coefficient of interval borders displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = -5.0; //Minimum range search input double RangeMaximum_P = 5.0; //Maximum range search input double Precision_P = 0.0001;//The required accuracy input int OptimizeMethod_P = 1; //Optim.:1-Min,other-Max //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double ERROR=0.0;//Average error in gen //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparing global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum range search RangeMaximum =RangeMaximum_P; //Maximum range search Precision =Precision_P; //Search step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of symbols in fitness GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//Proportion of crossing-over. //--- ReplicationOffset_P, //Coefficient of interval border replacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," mc - Time of implementation"); //---------------------------------- return(0); } //————————————————————————————————————————————————————————————————————————
Sorunu çözmek için script dosyasının tüm kodu buradadır. Çalıştırın, Comment () işlevi tarafından sağlanan bilgileri alın:
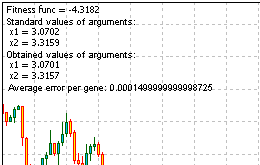
Şekil 13. Sorunun çözümünün sonucu
Sonuçlara baktığımızda algoritmanın çalıştığını görüyoruz.
4.2. Örnek 2
ZZ göstergesinin, devrilme alım satım sisteminin ideal girişlerini gösterdiğine yaygın olarak inanılmaktadır. Gösterge, "dalga teorisi" destekçileri ve onu "rakamların" büyüklüğünü belirlemek için kullananlar arasında çok popülerdir.
Sorun : Toplamda daha fazla teorik kazanç sağlayan, geçmiş veriler üzerinde ZZ tepe noktalarından farklı olan bir devrilme alım satım sistemi için başka giriş noktaları olup olmadığını belirleyin.
Denemeler için M1 100 çubuk için bir GBPJPY paritesi seçeceğiz. 80 noktada spread'i kabul edin (beş basamaklı fiyat teklifleri). Başlamak için en iyi ZZ parametrelerini belirlemeniz gerekir. Bunu yapmak için, basit bir script dosyası kullanarak ExtDepth parametresinin en iyi değerini bulmak için basit bir numaralandırma kullanırız:
#property script_show_inputs //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input int History=100; input double Spred =80.0; input int Depth =5; //For "one-time" use input bool loop =true;//Use enumeration or not //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //--------------------------The body of the program-------------------------------- void OnStart() { //-----------------------Variables------------------------------------- double ZigzagBuffer [];//For storing the buffer of the ZZ indicator double PeaksOfZigzag[];//for storing the values of the ZZ extremum int Zigzag_handle; //Indicator marker ArraySetAsSeries(ZigzagBuffer,true); ArrayResize(PeaksOfZigzag,History); int depth=3; double PipsSum=0.0; int PeaksCount=0; bool flag=true; //---------------------------------------------------------------------- if(loop==true) { while(depth<200 && flag==true) { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",depth); //--- reset the code error ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<100;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Could not copy the indicator buffer. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- PipsSum=0.0; PeaksCount=0; for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; //----------------------------------------------------------- if(PeaksCount<=2) flag=false; else { Print(depth," ",PeaksCount," ",PipsSum); depth+=1; } //----------------------------------------------------------- } } else { //----------------------------------------------------------- Zigzag_handle=iCustom(NULL,0,"ZigZag",Depth); //--- reser the error code ResetLastError(); //--- attempt to copy the indicator values for(int i=0;i<History;i++) { if(BarsCalculated(Zigzag_handle)>0) break; Sleep(1000); } int copied=CopyBuffer(Zigzag_handle,0,0,History,ZigzagBuffer); if(copied<=0) { Print("Was not able to copy the buffer indicator. Error =",GetLastError()," copied=",copied); return; } //----------------------------------------------------------- for(int u=0;u<History;u++) { if(NormalizeDouble(ZigzagBuffer[u],Digits())>0.0) { PeaksOfZigzag[PeaksCount]=NormalizeDouble(ZigzagBuffer[u],Digits()); PeaksCount++; } } //----------------------------------------------------------- for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(PeaksOfZigzag[V]-PeaksOfZigzag[V+1]))/Point(),Digits())-Spred; } Print(Depth," ",PeaksCount," ",PipsSum); //----------------------------------------------------------- } } //————————————————————————————————————————————————————————————————————————
Script dosyasını çalıştırarak ExtDepth = 3'te 4077 puan alıyoruz. On dokuz gösterge tepe noktası, 100 çubuğa "uygundur". ExtDepth'in artmasıyla ZZ tepe noktalarının sayısı azalır ve karlılık da düşer.
Şimdi UGA kullanarak alternatif ZZ'nin tepe noktalarını bulabiliriz. ZZ tepe noktaları, her çubuk için üç pozisyon içerebilir: 1) Yüksek, 2) Düşük, 3) Tepe noktasız. Tepe noktasının varlığı ve pozisyonu, her çubuk için her gen tarafından taşınacaktır. Böylece, kromozomun boyutu 100 gen olur.
Hesaplamalarıma göre (yanılıyorsam matematikçiler beni düzeltebilir), 100 çubukta 3 ^ 100 veya 5.15378e47 alternatif seçenek "zikzaklar" oluşturabilirsiniz. Bu, doğrudan numaralandırma kullanılarak dikkate alınması gereken seçeneklerin tam sayısıdır. Saniyede 100000000 seçenek hızıyla hesaplama sırasında, 1.6e32 yıla ihtiyacımız olacak! Bu, evrenin yaşından daha fazla. İşte o zaman, bu soruna bir çözüm bulabilme konusunda şüpheler duymaya başladım.
Ama başlayalım.
UGA, kromozomun gerçek sayılarla temsilini kullandığı için, tepe noktalarının konumunu bir şekilde kodlamamız gerekiyor. Bu, tam olarak kromozomun genotipinin fenotiple eşleşmediği durumdur. [0, 5] genleri için bir arama aralığı atayın. [0, 1] aralığının ZZ'nin Yüksek üzerindeki tepe noktasına, [4, 5] aralığının Düşük üzerindeki tepe noktasına ve (1, 4) aralığının tepe noktasının yokluğuna karşılık geldiğini kabul edelim.
Önemli bir noktayı dikkate almak gerekir. Proto- popülasyon, belirtilen aralıktaki genlerle rastgele oluşturulduğu için, ilk örnek, muhtemelen eksi işaretli birkaç yüz nokta ile dahi çok kötü sonuçlara sahip olacaktır. Birkaç jenerasyon sonra (birinci jenerasyonda olma ihtimali olsa da) genleri genel olarak tepe noktalarının yokluğu ile uyumlu olan örneğin görünümünü göreceğiz. Bu, alım satımın olmaması ve kaçınılmaz spread'in ödenmesi anlamına gelir.
Bazı eski yatırımcılara göre: "Alım satım için en iyi strateji, alım satım yapmamaktır". Bu birey yapay evrimin tepe noktasında olacaktır. Bu "yapay" evrimin alım satım yapan bireyleri ortaya çıkarmasını sağlamak (yani alternatif ZZ'nin tepe noktalarını düzenlemesini sağlamak için), tepe noktaları olmayan bireylerin uygunluğunu diğer bireylere kıyasla "-10000000.0" değerini evrimin en düşük basamağına bilerek yerleştirerek atarız.
Alternatif ZZ'nin tepe noktalarını bulmak için UGA'yı kullanan script dosyası kodu şu şekildedir:
#property script_show_inputs //+——————————————————————————————————————————————————————————————————————+ #include "UGAlib.mqh" //+——————————————————————————————————————————————————————————————————————+ //———————————————————————————————————————————————————————————————————————— //----------------------Incoming parameters-------------------------------- input string GenofundParam = "----Parameters of the gene pool----"; input int ChromosomeCount_P = 100; //Number of chromosomes in the colony input int GeneCount_P = 100; //Number of genes input int FFNormalizeDigits_P = 0; //Number of fitness symbols input int GeneNormalizeDigits_P= 0; //Number of gene symbols input int Epoch_P = 50; //Number of epochs without progress //--- input string GA_OperatorParam = "----Parameters of operators----"; input double ReplicationPortion_P = 100.0; //Proportion of replication. input double NMutationPortion_P = 10.0; //Proportion of natural mutations. input double ArtificialMutation_P = 10.0; //Proportion of artificial mutations. input double GenoMergingPortion_P = 20.0; //Proportion of borrowed genes. input double CrossingOverPortion_P = 20.0; //Proportion of crossing - over. input double ReplicationOffset_P = 0.5; //Coefficient of interval border displacement input double NMutationProbability_P= 5.0; //Probability of mutation of each gene in % //--- input string OptimisationParam = "----Optimization parameters----"; input double RangeMinimum_P = 0.0; //Minimum search range input double RangeMaximum_P = 5.0; //Maximum search range input double Precision_P = 1.0; //Required accuracy input int OptimizeMethod_P = 2; //Optim.:1-Min,other -Max input string Other = "----Other----"; input double Spred = 80.0; input bool Show = true; //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //----------------------Global variables----------------------------- double Hight []; double Low []; datetime Time []; datetime Ti []; double Peaks []; bool show; //———————————————————————————————————————————————————————————————————————— //--------------------------Body of the program-------------------------------- int OnStart() { //-----------------------Variables------------------------------------- //Preparation of global variables for UGA ChromosomeCount=ChromosomeCount_P; //Number of chromosomes in the colony GeneCount =GeneCount_P; //Number of genes RangeMinimum =RangeMinimum_P; //Minimum search range RangeMaximum =RangeMaximum_P; //Maximum search range Precision =Precision_P; //Searching step OptimizeMethod =OptimizeMethod_P; //1-minimum, any other - maximum FFNormalizeDigits = FFNormalizeDigits_P; //Number of fitness symbols GeneNormalizeDigits = GeneNormalizeDigits_P;//Number of gene symbols ArrayResize(Chromosome,GeneCount+1); ArrayInitialize(Chromosome,0); Epoch=Epoch_P; //Number of epochs without progress //---------------------------------------------------------------------- //Preparation of global variables ArraySetAsSeries(Hight,true); CopyHigh (NULL,0,0,GeneCount+1,Hight); ArraySetAsSeries(Low,true); CopyLow (NULL,0,0,GeneCount+1,Low); ArraySetAsSeries(Time,true); CopyTime (NULL,0,0,GeneCount+1,Time); ArrayResize (Ti,GeneCount+1);ArrayInitialize(Ti,0); ArrayResize(Peaks,GeneCount+1);ArrayInitialize(Peaks,0.0); show=Show; //---------------------------------------------------------------------- //local variables int time_start=GetTickCount(),time_end=0; //---------------------------------------------------------------------- //Очистим экран ObjectsDeleteAll(0,-1,-1); ChartRedraw(0); //launch of the main function UGA UGA ( ReplicationPortion_P, //Proportion of replication. NMutationPortion_P, //Proportion of replication of natural mutations. ArtificialMutation_P, //Proportion of artificial mutations. GenoMergingPortion_P, //Proportion of borrowed genes. CrossingOverPortion_P,//proportion of crossing- over. //--- ReplicationOffset_P, //Coefficient of interval border displacement NMutationProbability_P//Probability of mutation of each gene in % ); //---------------------------------- //Display the last result on the screen show=true; ServiceFunction(); //---------------------------------- time_end=GetTickCount(); //---------------------------------- Print(time_end-time_start," мс - time of execution"); //---------------------------------- return(0); } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Service function. Called up from UGA. | | //If there is no need for it, leave the function empty, like this: | // void ServiceFunction() | // { | // } | //-----------------------------------------------------------------------+ void ServiceFunction() { if(show==true) { //-----------------------Variables----------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //-------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Chromosome[u]; if(temp<=1.0 ) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); Ti [PeaksCount]=Time[u]; PeaksCount++; } } ObjectsDeleteAll(0,-1,-1); for(int V=0;V<PeaksCount-1;V++) { PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; ObjectCreate (0,"BoxBackName"+(string)V,OBJ_TREND,0,Ti[V],Peaks[V],Ti[V+1],Peaks[V+1]); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_COLOR,Black); ObjectSetInteger(0,"BoxBackName"+(string)V,OBJPROP_SELECTABLE,true); } ChartRedraw(0); Comment(PipsSum); } //---------------------------------------------------------------------- else return; } //———————————————————————————————————————————————————————————————————————— //———————————————————————————————————————————————————————————————————————— //-----------------------------------------------------------------------+ // Function of determining the fitness of the individual. Called up from UGA. | //-----------------------------------------------------------------------+ void FitnessFunction(int chromos) { //-----------------------Variables------------------------------------- double PipsSum=0.0; int PeaksCount=0; double temp=0.0; //---------------------------------------------------------------------- for(int u=1;u<=GeneCount;u++) { temp=Colony[u][chromos]; if(temp<=1.0) { Peaks[PeaksCount]=NormalizeDouble(Hight[u],Digits()); PeaksCount++; } if(temp>=4.0) { Peaks[PeaksCount]=NormalizeDouble(Low[u],Digits()); PeaksCount++; } } if(PeaksCount>1) { for(int V=0;V<PeaksCount-1;V++) PipsSum+=NormalizeDouble((MathAbs(Peaks[V]-Peaks[V+1]))/Point(),FFNormalizeDigits)-Spred; Colony[0][chromos]=PipsSum; } else Colony[0][chromos]=-10000000.0; AmountStartsFF++; } //————————————————————————————————————————————————————————————————————————
Script dosyasını çalıştırdığımızda, toplam karı 4939 puan olan tepe noktalarını alıyoruz. Ayrıca, doğrudan numaralandırma yoluyla ihtiyaç duyulan 3 ^ 100 ile karşılaştırıldığında, puanları saymak yalnızca 17.929 kez aldı. Bilgisayarımda bu, 1.6e32 yıla karşı 21,7 saniyedir!
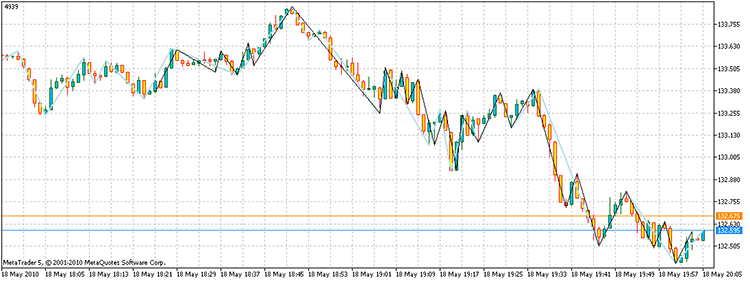
Şekil 14. Sorunun çözümünün sonucu. Siyah renkli bölümler - alternatif bir ZZ, gök mavisi - ZZ göstergesi
Yani sorunun yanıtı şu şekilde olacaktır: "Var."
5. UGA ile çalışmak için öneriler
- Algoritmadan yeterli bir sonuç bekleyebilmek için tahmin edilen koşulları FF'de doğru bir şekilde ayarlamaya çalışın. Örnek 2'yi tekrar düşünün. Bu, belki de benim temel tavsiyemdir.
- Kesinlik parametresi için çok küçük bir değer kullanmayın. Algoritma adım 0 ile çalışabilmesine rağmen, çözümün makul bir doğruluğunu talep etmelisiniz. Bu parametre, sorunun boyutunu indirgemek için tasarlanmıştır.
- Popülasyonun büyüklüğünü ve dönem sayısının eşik değerini değiştirin. MaxOfCurrentEpoch tarafından gösterilenden iki kat daha büyük bir Dönem parametresi atamak iyi bir çözüm olabilir. Çok büyük değerler seçmeyin; bu, soruna çözüm bulmayı hızlandırmayacaktır.
- Genetik operatörlerin parametreleriyle deneme yapın. Evrensel parametreler yoktur ve bunları önünüzdeki görevin koşullarına göre atamanız gerekir.
Bulgular
Çok güçlü bir personel terminali strateji test cihazı ile birlikte MQL5 dili, yatırımcı için daha az güçlü bir enstrüman oluşturmanıza izin vererek, gerçekten karmaşık sorunları çözmenize olanak tanır. Çok esnek ve ölçeklenebilir bir optimizasyon algoritması elde ediyoruz. Ve bunu ilk oluşturan ben olmamama rağmen, bu keşiften herhangi bir şekilde utanç hissetmeden gurur duyuyorum.
UGA başlangıçta ek operatörler ve hesaplama blokları ile kolayca değiştirilip genişletilebilecek şekilde tasarlandığı için okuyucu "yapay" evrimin gelişimine kolaylıkla katkıda bulunabilecektir.
Okuyuculara optimum çözümler bulmada başarılar dilerim. Umarım bu konuda onlara yardımcı olabilmişimdir. İyi şanslar!
Not. Makalede ZigZag göstergesi kullanılmıştır. UGA'nın tüm kaynak kodları ektedir.
Lisanslama: Makaleye eklenen kaynak kodları (UGA kodu), BSD lisans koşulları altında dağıtılmaktadır.
MetaQuotes Ltd tarafından Rusçadan çevrilmiştir.
Orijinal makale: https://www.mql5.com/ru/articles/55
Uyarı: Bu materyallerin tüm hakları MetaQuotes Ltd.'a aittir. Bu materyallerin tamamen veya kısmen kopyalanması veya yeniden yazdırılması yasaktır.
Bu makale sitenin bir kullanıcısı tarafından yazılmıştır ve kendi kişisel görüşlerini yansıtmaktadır. MetaQuotes Ltd, sunulan bilgilerin doğruluğundan veya açıklanan çözümlerin, stratejilerin veya tavsiyelerin kullanımından kaynaklanan herhangi bir sonuçtan sorumlu değildir.
 Farklı Kıtalardaki Saat Dilimi Farklılıklarına Dayalı Bir Alım Satım Stratejisi Örneği
Farklı Kıtalardaki Saat Dilimi Farklılıklarına Dayalı Bir Alım Satım Stratejisi Örneği
 "Yeni Başlayanlar" için MQL: Nesne Sınıfları Nasıl Tasarlanır ve Oluşturulur?
"Yeni Başlayanlar" için MQL: Nesne Sınıfları Nasıl Tasarlanır ve Oluşturulur?
 Alım Satım Raporları ve SMS Bildirimi Oluşturma ve Yayınlama
Alım Satım Raporları ve SMS Bildirimi Oluşturma ve Yayınlama
 Yeni Başlayanlar için Çoklu Gösterge Arabelleği ile Bir Gösterge Oluşturma
Yeni Başlayanlar için Çoklu Gösterge Arabelleği ile Bir Gösterge Oluşturma
- Ücretsiz alım-satım uygulamaları
- İşlem kopyalama için 8.000'den fazla sinyal
- Finansal piyasaları keşfetmek için ekonomik haberler
Gizlilik ve Veri Koruma Politikasını ve MQL5.com Kullanım Şartlarını kabul edersiniz
Lütfen bana hatalistesinigösterin.Kodçok eski, ama 14 yılsonra hatta kıracakbir şey yok.
sorun "ArtificialMutation" değişkeni ve fonksiyonu ile ilgili. Fonksiyonu ArtificialMutationP olarak değiştirdim ve çalışıyor.
ServiceFunction ve FitnessFunction hatası ile ilgili sorun yok, orada neler olduğunu anlıyorum. Ekran görüntülerini ekledim.
ZIP'ten çıkardıktan sonra doğrudan UGA_script veya UGA_the_alternative_ZigZag'ı derlemeye çalıştığımda UGAlib'den gelen hatalar alıyorum.
Fonksiyon adını (ve 2. durumdaki çağrısını) ArtificialMutationP olarak değiştirdiğimde derlendi ve çalıştı.
Hala tam olarak anlamak için güzel çalışmanız üzerinde çalışıyorum. Teşekkür ederim
Soru.
geçmiş veriler ( çubuk sayısı) gen sayısıdır, bu doğru mu?
sorun "ArtificialMutation" değişkeni ve fonksiyonu ile ilgili. Fonksiyonu ArtificialMutationP olarak değiştirdim ve çalışıyor.
ServiceFunction ve FitnessFunction hatası ile ilgili sorun yok, orada neler olduğunu anlıyorum. Ekran görüntülerini ekledim.
ZIP'ten çıkardıktan sonra doğrudan UGA_script veya UGA_the_alternative_ZigZag'ı derlemeye çalıştığımda UGAlib'den gelen hatalar alıyorum.
İşlev adını (ve 2. durumda çağrısını) ArtificialMutationP olarak değiştirdiğimde derlendi ve çalıştı.
Hala tam olarak anlamak için güzel çalışmanız üzerinde çalışıyorum. Teşekkür ederim.
bu dosyasınıdeneyin
Nazik sözleriniziçinteşekkür ederim.